Los últimos avances en las tecnologías de secuenciación han permitido mejorar el conocimiento en genómica. Ello se traduce a un mejor entendimiento de cómo es y cómo se estructura el genoma. Además, sirve para comprender con más detalle el rol que juega cada uno de los genes conocidos en las distintas funciones descritas. Sin embargo, las técnicas de Segunda Generación (NGS), todavía, son plataformas relativamente caras y que precisan de unas necesidades específicas. Los protocolos propios de dichos métodos, incorporan una etapa de amplificación del ADN a secuenciar, que puede llevar a la introducción de errores con facilidad. Además, las secuencias ricas en AT o AG dan lugar a sesgos que complican el posterior análisis bioinformático de ensamblaje, debido a una representación insuficiente en lecturas de estas regiones.

Por ello, la idea de buscar un alternativa más sencilla, rápida y eficaz, y al menor coste posible, es una cuestión que siempre ha estado y está en la mente de los científicos. Con este fin, nace lo que se conoce como SECUENCIACIÓN DE TERCERA GENERACIÓN. Aunque algunas de estas plataformas, o bien se encuentran todavía en fase de experimentación o requieren de la mejora en muchos de sus aspectos, se consideran como uno de los avances más esperados para esta segunda década del siglo XXI. Todas las estrategias se fundamentan en la idea de eliminar la etapa de PCR, y tratar de realizar la propia secuenciación a partir de una única molécula de ADN. Al mismo tiempo que se trata abaratar los costes, conseguir ofrecer lecturas más largas que faciliten el posterior ensamblado y ampliar el abanico de aplicaciones, como la detección de grandes variaciones en la estructura de los cromosomas o caracterización de haplotipos, entre otras.
Aunque a priori, parece algo prácticamente imposible, ya existe en el mercado la tecnología Single Molecule, Real-time (SMRT), desarrollada por la casa Pacific Biosciencies, que trata de ofrecer una secuenciación de estas características. Como su propio nombre nos lleva a intuir, es una plataforma que utiliza la fluorescencia para secuenciar una única molécula de ADN en tiempo real. El diseño se basa en nanopocillos, en cuya base existe un orificio, denominado zero-mode waveguide (ZMW), por donde es posible el paso de la luz. En cada ZMV, queda inmovilizada una única molécula junto con una sola enzima de ADN polimerasa. Tras la incorporación de los nucleótidos, cada uno marcado con un fluoróforo de un color distinto, por parte de la ADN polimerasa a la hebra en síntesis, se libera el fluoróforo que es excitado por el haz de luz, siendo detectable por el detector. De esta forma, es posible saber que nucleótido se ha incorporado, y así, elucidar la secuencia desconocida.
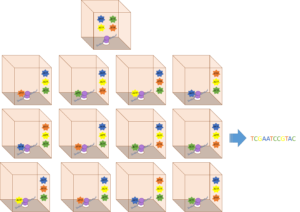
SMRT permite incorporar lecturas de unas 60.000 pares de bases (bp), por lo que supone un atractivo para aquellos estudios en los que se tratan de averiguar variaciones estructurales o que presentan dificultades en la amplificación de ciertas áreas del genoma. También tiene la ventaja de que es capaz de secuenciar bases modificadas, como la 5’-metil-citosina, gracias a que es posible la incorporación de este tipo de nucleótidos. Sin embargo, a día de hoy, se trata de una metodología que requiere de grandes cantidades de ADN para poder ofrecer un resultado sólido, ya que no amplifica, por lo que no es apto para un proyecto de secuenciación del genoma completo. Además, adquirir el equipamiento necesario supone una gran inversión inicial, y el posterior mantenimiento resulta muy caro. Todo limitaciones que están tratando de resolverse de cara a los próximos años.

Recientemente, en apenas dos años, ha surgido otra estrategia que ha entrado en el mercado como una auténtica revolución por su amplia versatilidad. Es el caso de la técnica de secuenciación mediante Nanoporos, un método de secuenciación que permite secuenciar secuencias de ADN individualizadas, de forma directa, barata y en el mismo momento. Aunque todavía se encuentra en vías de optimización, otra ventaja que supone frente al resto de tecnologías, es que será capaz de secuenciar toda la longitud de la secuencia a analizar, en lugar de generar lecturas de un tamaño específico. A esto es lo que se comienza a llamar como capacidad de ofrecer lecturas “ultra largas”
Todo esto se consigue, gracias a que esta tecnología está basada en el principio de leer directamente el fragmento de ADN a secuenciar mediante el paso de dicha molécula única por un nanoporo, a la vez que se va midiendo los efectos, a nivel iónico y eléctrico, que supone el avance del ADN.
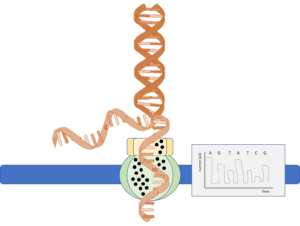
Al igual que en SMRT, la secuenciación mediante nanoporos también presenta la limitación de que se requiere de una gran cantidad de ADN, para poder secuenciar y obtener un resultado fiable. Secuenciar una única molécula, tiene el inconveniente, es que se necesita de la secuenciación de muchas moléculas únicas para poder obtener una secuencia consenso aceptable entre todas las idénticas y se eliminen los posibles errores introducidos.

Si se consiguiera superar esta barrera, y ofrecer una precisión de secuenciación lo suficientemente alta incrementando el rendimiento a unos niveles superiores a los ya existentes, este tipo de tecnologías podrían comenzar a implantarse en las disciplinas emergentes como la medicina personalizada o la farmacogenética, por citar solo un par de ejemplos.
Nos vemos pronto ?

