Muchas terapias están enfocadas a aliviar los síntomas de una determinada patología. Pero, ¿no sería genial desarrollar tratamientos que intenten solucionar directamente la causa? Actualmente hay muchas líneas de investigación trabajando en ello para muchas enfermedades. Un ejemplo es la regulación del splicing alternativo en la distrofia muscular de Duchenne.
A continuación vamos a ver cómo modificando el funcionamiento del splicing se puede tratar la distrofia muscular de Duchenne. Para ello, primero hablaremos sobre la distrofia muscular de Duchenne y sobre el mecanismo general del splicing.
Distrofia muscular de Duchenne
La distrofia muscular de Duchenne es una enfermedad genética de herencia recesiva. Está causada por mutaciones en el gen DMD, localizado en el cromosoma X, que codifica para la proteína distrofina. La función de dicha proteína es mantener la integridad de la membrana de las células musculares y, por lo tanto, fortalecer el músculo.
Los pacientes con esta patología presentan mutaciones en el gen DMD que hacen que la distrofina sintetizada no sea plenamente funcional o que no sea nada funcional, en los casos más graves. Los principales síntomas de la distrofia muscular de Duchenne son atrofia y debilidad muscular progresivas, lo que reduce considerablemente la esperanza de vida de los pacientes.
El DMD es el gen humano más largo en cuanto a secuencia que se conoce. Tiene un total de 79 exones (secuencias codificantes, que codifican para la síntesis de proteínas), pero el 99,5% de la secuencia del gen está compuesta por intrones, es decir, secuencias no codificantes (que no serán traducidas para sintetizar una proteína). En este gen se produce splicing alternativo, pues tiene una expresión específica para cada tejido. Es decir, la proteína distrofina no será igual en todos los tejidos, hay varias versiones de ella.
Además, en el gen DMD hay una región susceptible a mutaciones entre los exones 43 y 63, que pueden interrumpir el proceso de traducción de la distrofina antes de tiempo, de manera que se produce una proteína no funcional o parcialmente funcional.
Splicing constitutivo y splicing alternativo
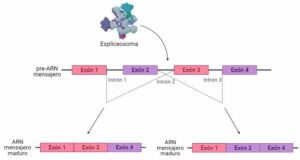
Cuando se produce la transcripción de un gen (el paso de la información genética del ADN al ARN) con más de un exón el primer producto que se obtiene es un pre-ARN mensajero, que todavía no está listo para ser traducido. Este pre-ARN mensajero contiene exones e intrones. Para obtener el ARN mensajero maduro, los intrones deben ser eliminados y los extremos de los exones deben unirse entre ellos. Este proceso de eliminación de intrones para formar el ARN mensajero listo para su traducción es el splicing o procesado del ARN.
El splicing tiene lugar de forma natural en las células para obtener mensajeros que se traduzcan y así sintetizar proteínas. En el caso del splicing constitutivo a partir de un ARN mensajero se obtiene siempre la misma proteína. Además de esto, existe también el splicing alternativo, que hace posible que a partir de un único gen se pueda obtener no solamente una, sino distintas proteínas. Durante el splicing alternativo se pueden quedar también algunos intrones, dando lugar a distintas combinaciones de secuencias, y por tanto, a distintos productos proteicos.
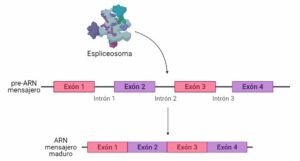
El encargado de llevar a cabo ambos tipos de splicing es el espliceosoma, un complejo formado por unas 150 proteínas y 5 riboproteínas pequeñas. El espliceosoma es el que reconoce determinadas secuencias a los extremos de los exones, que le indican que debe cortar la secuencia. Así escinde los intrones y luego une los extremos de los exones, dando lugar a un ARN mensajero cuya secuencia está preparada para ser traducida por los ribosomas y sintetizar proteínas.
Sin embargo, en el caso del splicing alternativo, lo que ocurre es que en determinados exones las secuencias que debe reconocer el espliceosoma son ligeramente diferentes a las que dicho complejo suele reconocer, por lo que en ocasiones hay determinados exones que no son reconocidos y se escinden junto a los intrones que los flanquean. Esto puede causar cambios en la pauta de lectura para los ribosomas, por lo que las proteínas que sinteticen serán diferentes. Como ocurre con todos los procesos celulares, es importante la correcta regulación del splicing alternativo y constitutivo, por eso las mutaciones que alteran su funcionamiento causan patologías.
Tratamiento para la distrofia muscular de Duchenne: exón skipping
No existe una cura para la distrofia muscular de Duchenne, pero se están desarrollando terapias que tratan de regular el splicing alternativo. Se buscar la forma de que se pueda producir, al menos, una proteína distrofina que no es completamente funcional, ya que eso es mejor que la ausencia de funcionalidad total de la proteína. Estas terapias inducen el “exón skipping”, es decir, que durante el splicing el espliceosoma se “salte” algunos exones. Concretamente se diseñan para que se salte aquellos exones donde hay una mutación de las que producen distrofina no funcional y los elimine junto a los intrones.
Actualmente hay varios fármacos aprobados por la FDA (Administración de Alimentos y Medicamentos de Estados Unidos) que funcionan, bien induciendo el exón skipping de un solo exón, o bien a través de otros tipos de mecanismos genéticos. Algunos ejemplos son Eteplirsen, Emflaza y Vyondys 53.
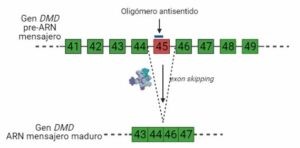
Una forma de inducir el exón skipping como tratamiento consiste en la administración de oligómeros u oligonucleótidos antisentido, que son secuencias de ARN complementarias a determinadas regiones del pre-ARN mensajero. Esos oligómeros se unen a dicho mensajero; entonces el espliceosoma no reconoce los exones a los que se han unido los oligómeros, se los “salta”.
Este mecanismo se puede utilizar por ejemplo para saltar el exón 45, ya que se ha visto que así se obtiene una distrofina funcional, aunque no llega a ser como la distrofina obtenida a partir del gen sin mutaciones. Esta forma de regulación del splicing alternativo supone una mejoría para los pacientes cuya distrofina no es nada funcional.
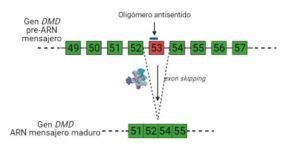
Otro ejemplo de terapias aprobadas por la FDA que utilizan el exón skipping es el fármaco Vitolarsen, que induce la omisión del exón 53. En los pacientes que han recibido este tratamiento se ha observado una mayor expresión de distrofina.
También se están desarrollando cócteles de oligómeros antisentido, que están dirigidos a más de un exón. Se ha probado, por ejemplo, con la secuencia entre los exones 45 al 55, de momento en células humanas y en ratón. Esos cócteles hacen que en el ARN mensajero del gen DMD se eliminen esos exones. No se consigue una distrofina completamente funcional, se obtiene una distrofina parcialmente funcional, por lo que su objetivo son los pacientes con fenotipo más severo (con una distrofina nada funcional). La ventaja de estos cócteles es que pueden abarcar un mayor rango de mutaciones. Es decir, un mismo cóctel se podría administrar a pacientes con distintas mutaciones en distintos exones, no es necesario diseñar un tratamiento específico para cada uno.
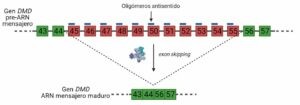
Los oligómeros antisentido para la distrofia muscular de Duchenne son solamente uno de los múltiples ejemplos de que regulando los procesos de splicing se pueden encontrar terapias prometedoras. Además, también se están investigando tratamientos similares en patologías como el cáncer.
Fuentes:
Black AJ, Gamarra JR, Giudice J. More than a messenger: Alternative splicing as a therapeutic target. Biochim Biophys Acta Gene Regul Mech. 2019 Nov-Dec;1862(11-12):194395. doi: 10.1016/j.bbagrm.2019.06.006. Epub 2019 Jul 2. PMID: 31271898; PMCID: PMC6875611.
Echigoya Y, Lim KRQ, Melo D, Bao B, Trieu N, Mizobe Y, Maruyama R, Mamchaoui K, Tanihata J, Aoki Y, Takeda S, Mouly V, Duddy W, Yokota T. Exons 45-55 Skipping Using Mutation-Tailored Cocktails of Antisense Morpholinos in the DMD Gene. Mol Ther. 2019 Nov 6;27(11):2005-2017. doi: 10.1016/j.ymthe.2019.07.012. Epub 2019 Jul 26. PMID: 31416775; PMCID: PMC6838919.
Gallego-Paez LM, Bordone MC, Leote AC, Saraiva-Agostinho N, Ascensão-Ferreira M, Barbosa-Morais NL. Alternative splicing: the pledge, the turn, and the prestige : The key role of alternative splicing in human biological systems. Hum Genet. 2017 Sep;136(9):1015-1042. doi: 10.1007/s00439-017-1790-y. Epub 2017 Apr 3. PMID: 28374191; PMCID: PMC5602094. https://pubmed.ncbi.nlm.nih.gov/28374191/
Tuffery-Giraud S, Miro J, Koenig M, Claustres M. Normal and altered pre-mRNA processing in the DMD gene. Hum Genet. 2017 Sep;136(9):1155-1172. doi: 10.1007/s00439-017-1820-9. Epub 2017 Jun 9. PMID: 28597072.
Si te ha gustado esta entrada y te interesa la genética, descubre nuestros nuevos cursos y formación universitaria.


