¡Hola, lectores de nuestro blog! Hoy os quiero hablar de una técnica indispensable en cualquier laboratorio que trabaje con genética. Me refiero a la mundialmente conocida “reacción en cadena de la polimerasa”, comúnmente denominada PCR. ¿Quieres saber más? ¡Sigue leyendo!
¿Qué es la PCR?
La PCR es una técnica utilizada en biología molecular que permite conseguir una gran cantidad de copias de un fragmento de ADN, partiendo de una cantidad ínfima de esta biomolécula. Esta técnica fue diseñada tal y como la conocemos por los bioquímicos Kary Banks Mullis y Michael Smith en la década de los 90, quienes consiguieron patentarla.
La PCR se basa en una actividad enzimática que sucede de forma normal en las células de nuestro organismo. En las células, las ADN polimerasas son capaces de replicar el ADN nuclear, para obtener dos copias idénticas, que después serán repartidas a las células hijas en la mitosis. Bien, pues, de igual modo, en la PCR las polimerasas serán capaces de replicar, cual fotocopiadora, un fragmento de ADN, en varios ciclos, para obtener una gran cantidad de copias igualitas a ella.
¿Para qué sirve una PCR?
Todo investigador que trabaje con moléculas de ADN conoce esta técnica. Y es que la PCR es una técnica imprescindible en múltiples ámbitos. Por ejemplo, en investigación bioquímica y médica, la PCR permite preparar fragmentos de ADN para su clonación en plásmidos bacterianos o virus para utilizarlos como vectores, paso imprescindible en el desarrollo de terapias génicas. Además, en Medicina, esta técnica es utilizada para diagnosticar enfermedades hereditarias. ¡Incluso se utiliza para identificar patógenos!
Por si fuera poco, la PCR es una técnica importantísima en las pruebas de paternidad y en los análisis forenses de la policía científica. Interesante, ¿verdad?

¿Qué se necesita para hacer una PCR?
Seguir el procedimiento de una técnica de laboratorio no es tan diferente de una receta de cocina. Antes de nada, necesitamos reunir los ingredientes. ¿Cuáles son? Los ingredientes esenciales para llevar a cabo una PCR son los siguientes:
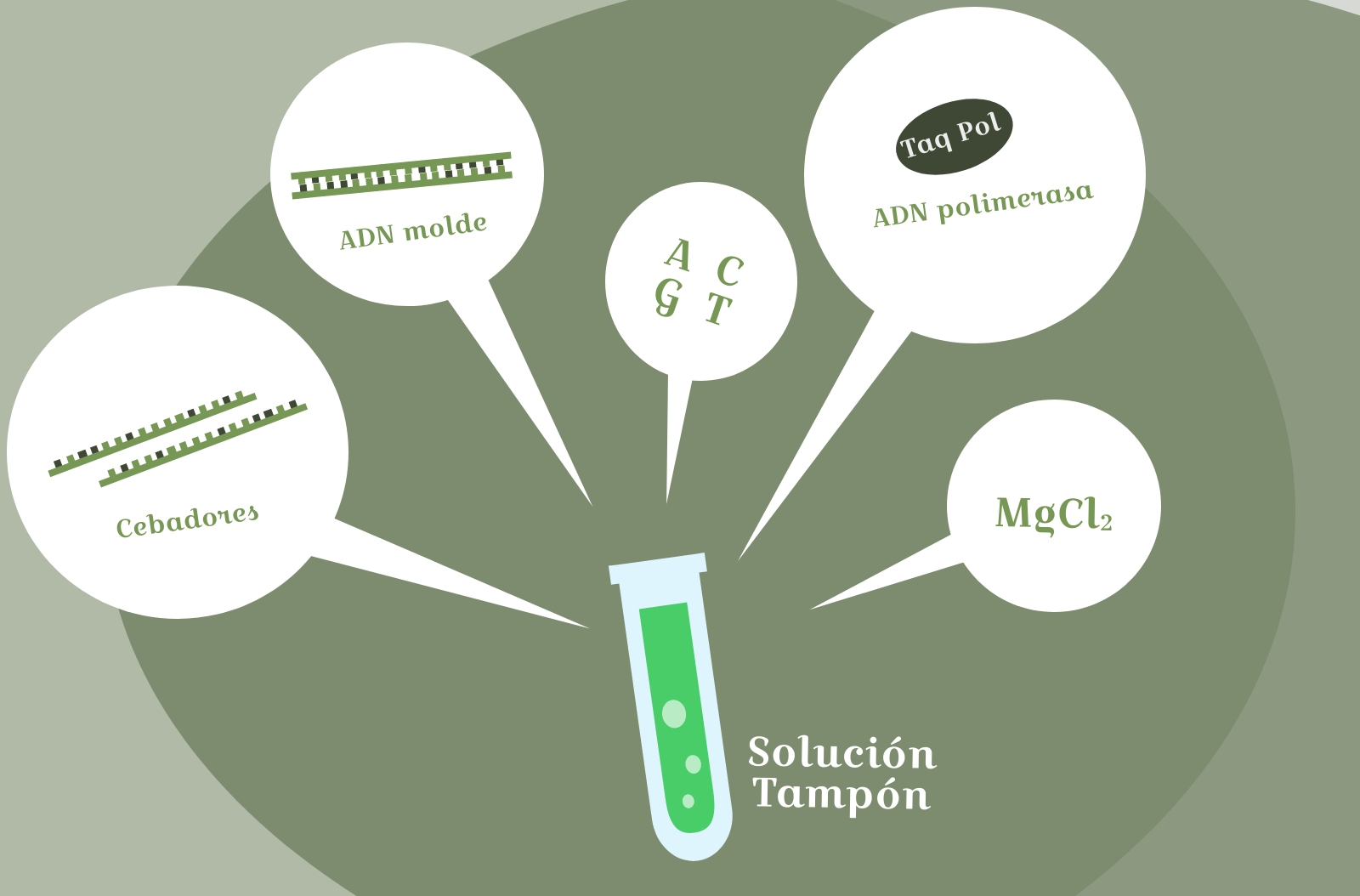
- ADN molde
Se trata del fragmento de ADN que queremos amplificar mediante la PCR.
- Desoxirribonucleótidos-trifosfato:
Como sabéis, el ADN está compuesto por 4 tipos de nucleótidos, formados por 4 bases nitrogenadas: adenina, guanina, citosina y timina. Es de esperar, entonces, que estos 4 desoxirribonucleótidos-trifosfato sean indispensables para poder obtener nuevas moléculas de ADN
- Cebadores (en inglés, primers)
Los cebadores son oligonucleótidos, secuencias cortas de ADN, que se unen a la molécula de ADN molde y sirven como punto de inicio para comenzar la síntesis de ADN. En la PCR necesitamos dos cebadores, cada uno complementario a una cadena del ADN que buscamos amplificar. Estos oligonucleótidos determinan la región del ADN a amplificar.
- ADN polimerasas
Las reinas de esta técnica bioquímica. En la PCR se pueden utilizar diferentes ADN polimerasas, obtenidas de varios organismos. Sin embargo, la más utilizada dada su efectividad, es la ADN polimerasa de la bacteria Thermus aquaticus, también llamada Polimerasa Taq. Esta polimerasa es idónea para la PCR por su resistencia a las altas temperaturas que se utilizan en el proceso.
- Iones divalentes de magnesio
En la PCR se utilizan iones de carga positiva como cofactores de la polimerasa. Estos cationes son esenciales para la función de la ADN polimerasa. Normalmente se añade cloruro de magnesio para que al disociarse se libere magnesio ( con carga +2).
- Solución tampón
Una solución tampón es una disolución que es capaz de regular el pH, es decir, las condiciones de acidez o basicidad, de nuestra PCR. Esto es muy importante, porque los cambios en el pH de la disolución pueden alterar los resultados de nuestra PCR o evitar que se produzca
Estos son los ingredientes básicos. Sin embargo, como en cualquier receta de cocina, necesitamos algo (un instrumento) para procesar los ingredientes y conseguir los resultados que buscamos. En el caso de la PCR es muy importante regular las condiciones de temperatura en las que se encuentran las reacciones. Para ello, se utiliza un “horno” especial, el termociclador.
- Termociclador
Un termociclador es un aparato que regula la temperatura en cada ciclo de la PCR. Gracias a él es posible realizar esta técnica en muchísimo menos tiempo, ya que para realizarla, se debe modificar la temperatura de la solución en varias ocasiones.
¡Bien! Ya sabemos qué elementos básicos necesitamos para realizar una PCR. Ahora, solo nos queda conocer el procedimiento a seguir en esta técnica:
¿Cómo funciona una PCR?
La PCR se compone de varios ciclos, que se repiten unas 30 veces, dependiendo de la cantidad de muestra que necesitamos. Cada ciclo comprende la desnaturalización de la doble hebra de ADN y la síntesis de la una nueva cadena de ADN por cada cadena haya presente. De este modo, si comenzamos con una única molécula de ADN en la muestra, obtendremos 2 en el primer ciclo, 4 en el segundo ciclo… ¡y 2 147 483 648 moléculas en el ciclo número 29!
En la primera parte del ciclo de una PCR, se desnaturalizan la moléculas de ADN de la muestra. Esto significa que las dos cadenas que forman cada molécula se separan, dando lugar a dos moléculas de ADN monocatenario. Normalmente, este primer paso se logra realizar mediante un aumento muy grande de la temperatura de la solución (aproximadamente a 95º).
El siguiente paso de un ciclo de la PCR implica unas moléculas muy importantes, de las que hablaba antes, los cebadores. En este paso, se disminuye la temperatura de la solución para favorecer la unión de los cebadores al ADN monocatenario que habíamos obtenido en el proceso anterior. Recordad que los cebadores se unen al ADN de forma específica, así que solo amplificaremos la región de las moléculas que nos interese.
Acto seguido, se ajusta la temperatura de la solución de nuevo, para que la ADN polimerasa pueda actuar a partir de los cebadores. La temperatura en este paso es dependiente de la ADN polimerasa que se esté utilizando. Por ejemplo, para la polimerasa Taq, la temperatura ideal está entre los 70 ºC y los 80 ºC. En este paso, la polimerasa utiliza como molde las cadenas de ADN monocatenario y va añadiendo al cebador los desoxirribonucleótidos-trifosfato complementarios a la cadena molde, formando una nueva cadena.
Aquí os muestro un esquema de la reacción en cada ciclo:
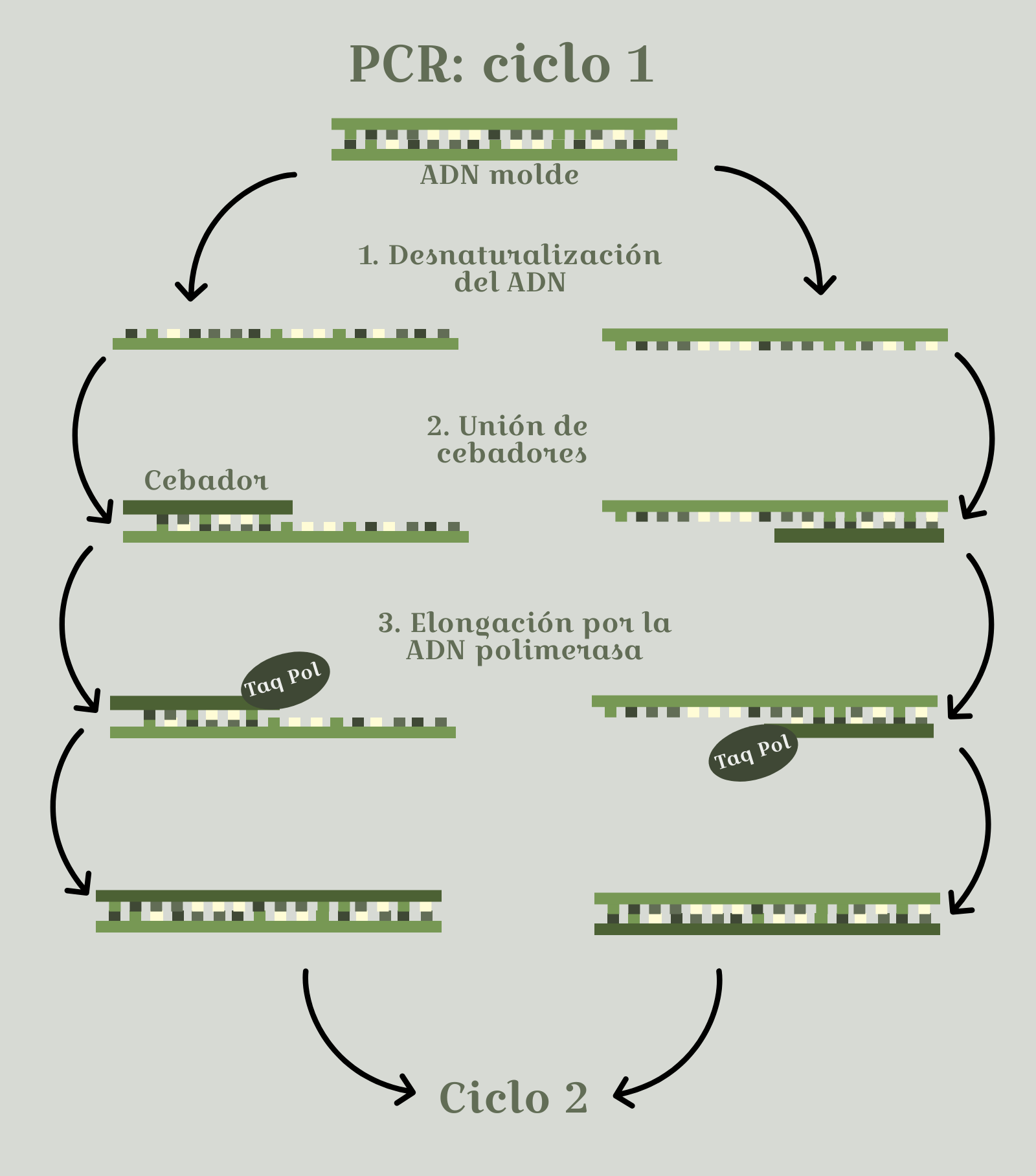
Una vez se ha realizado un ciclo, se repiten los pasos de nuevo. De este modo, las cadenas obtenidas en el primer ciclo se utilizan como molde en el segundo ciclo, las obtenidas en el segundo se usan como molde en el tercero y así consecutivamente.
Este es el tipo de PCR más sencillo. Sin embargo, existen muchas variaciones de esta técnica que nos permiten obtener diferentes resultados. Veamos algunas de ellas:
Otros tipos de PCR
PCR anidada: se trata de una variante de la PCR básica que utiliza dos pares de cebadores. En un primer paso, se realiza la amplificación de una región del genoma, para después concretar más la región mediante una segunda amplificación más específica. Esta PCR se utiliza para amplificar fragmentos muy específicos del genoma.
RT-PCR: Esta técnica convierte el ARN de una muestra en ADN. Para ello utiliza la transcriptasa inversa, una enzima utilizada por los retrovirus Se utiliza para múltiples objetivos. Por ejemplo se puede utilizar para saber si un gen se está expresando en una muestra biológica. También se utiliza para genotipar diferentes virus de ARN, como el SARS-Co-V o el VIH.
PCR cuantitativa: es una PCR que permite medir en tiempo real la cantidad de fragmentos que se van produciendo. Se utiliza a menudo para analizar la expresión de los genes.
PCR múltiple: en este tipo de PCR se realizan amplificaciones simultáneas de más de un fragmento de ADN. Para ello, se utilizan varios cebadores diferentes en una misma reacción.
PCR in situ: esta PCR se realiza en células o tejidos. Se utiliza para poder detectar secuencias de ADN en el interior de las células que no son detectables mediante otras técnicas.
PCR digital: es una de las últimas generaciones en técnicas de amplificación de ADN. Está basada en la separación de cada muestra en múltiples particiones (microgotas), de forma que la reacción de amplificación se produce de forma independiente cada una de ellas.
Estos solo son algunos de los tipos más conocidos de PCR, aunque también existen muchas otras variaciones de la PCR, como la PCR asimétrica, o la PCR específica de alelo, que consiguen diferentes resultados a partir de las muestras de ADN obtenidas.
Y hasta aquí el post de hoy. Espero que os haya resultado interesante y que os haya dejado con ganas de más, porque esta semana publicaremos un nuevo post sobre esta técnica. ¡No dudéis en escribir en los comentarios si tenéis cualquier duda!



