INTRODUCCIÓN
Los conjuntos de datos moleculares a gran escala, generados por las diferentes tecnologías experimentales, son la base para entender la vida como un sistema molecular y para desarrollar aplicaciones prácticas en la ciencia médica, la farmacéutica y la ambiental. En un contexto científico en el que cada vez se dispone de más información acerca de multitud de procesos moleculares y celulares, debemos aprovechar dicha información para conocer mejor determinadas enfermedades. Los avances realizados en el campo de las disciplinas ómicas son de gran utilidad para comprender de un modo más eficiente los mecanismos moleculares de algunas enfermedades, así como para dar explicación a la diferente respuesta a fármacos en pacientes (Meyer et al., 2013). Pueden ser empleadas varias aproximaciones desde diferentes perspectivas, no obstante, la integración de toda la información es un modo efectivo de realizar modelos predictivos y plantear hipótesis más dirigidas y concretas, que puedan ser más adelante, validadas experimentalmente.
La finalización del Proyecto del Genoma Humano (I.H.G.S. Consortium et al., 2004; Levy et al., 2007) ha traído consigo un gran avance en la medicina genómica. Este proyecto fue un importante motor para el uso del conocimiento sobre el genoma y para aplicaciones en el diagnóstico y tratamiento de enfermedades provocadas por la alteración de un único gen. Sin embargo, una mejor comprensión de las interacciones entre los diferentes genes y factores no genómicos que producen enfermedades está allanando el camino para una era de medicina genómica (Feero et al., 2010). Además, un aspecto a tener en consideración es la disminución progresiva de los costes de genotipado. Estas cifras se han reducido de los 10 $ (por nucleótido) que costaba en 1990 a 0.01 $ en 2005, lo que supone un aumento considerable de la información de la que disponemos hoy día con respecto a épocas anteriores.
La medicina genómica supone la aplicación de este conocimiento a la medicina, con el fin de alcanzar una medicina personalizada o de precisión (Conti et al., 2010). Por otro lado, la cantidad de información disponible nos permite realizar enfoques multidisciplinares en los que una enfermedad pueda ser estudiada como un sistema en el que intervienen multitud de componentes moleculares. Diversas disciplinas han surgido durante los últimos años debido a la necesidad de dar explicación a la diferente respuesta a fármacos observada en pacientes.
FARMACOGENÉTICA
En la actualidad, disciplinas como la farmacogenética han centrado su campo de estudio en los mecanismos moleculares de respuesta a fármacos para dar contestación a la pregunta de por qué existen diferentes respuestas celulares en función de la variabilidad genética. La farmacogenética se define como la disciplina que estudia el efecto de la variabilidad genética de un individuo en respuesta a determinados fármacos (Roses, 2000). Ya en el año 1959 se acuñó por primera vez el término de farmacogenética por Frederich Vogel para designar el estudio del papel que juega la variación genética individual en la respuesta a medicamentos.
Actualmente se desarrollan cada vez más estudios relacionados con la respuesta a fármacos desde una perspectiva basada en la farmacogenética, debido a la diferente respuesta que presentan algunos individuos frente a un determinado tratamiento.
Además, la interacción entre fármacos es un hecho a tener en consideración, ya que muchos de los tratamientos de algunas enfermedades consisten en la administración de varios fármacos. Es por ello que se hace necesario un enfoque multidisciplinar que considere una enfermedad como un sistema en el que intervienen muchos componentes y cuyas relaciones sean objeto de estudio, con el fin de comprender de un modo eficiente dicha enfermedad. Este tipo de enfoques son necesarios para la consecución de una medicina personalizada que nos permita tratar clínicamente a un paciente en función de su genotipo. En el campo de la farmacogenética es especialmente importante una familia de enzimas que metaboliza la mayor parte de fármacos, denominada citocromo P450. Cambios polimórficos en los genes que dan lugar a los transcritos de las proteínas de esta familia son suficientes para provocar en el individuo una respuesta variable a determinados fármacos (Ahmed et al., 2018). En función de los polimorfismos (SNPs, Single Nucleotide Polymorphisms) presentes en cada individuo y de sus diferentes combinaciones, es posible predecir en dicho individuo el fenotipo asociado al metabolismo de fármacos (Zanger et al., 2013):
- Metabolizador lento (Poor Metabolizer, PM): incapaz de procesar los fármacos por carecer de alelos funcionales. Estos individuos necesitan una menor dosis del fármaco para que este sea efectivo, debido a la dificultad de metabolizarlo, ya que éste queda retenido en el organismo, con sus consecuentes efectos nocivos.
- Metabolizador rápido o extensivo (Extensive metabolizer, EM): es considerado el fenotipo normal, ya que procesa los fármacos en tiempo y forma adecuada.
- Metabolizador intermedio (Intermediate Metabolizer, IM): procesa los fármacos un poco más lento que el fenotipo normal.
- Metabolizador ultrarrápido (Ultrafast Metabolizer, UM): estos individuos transforman los fármacos muy rápidamente. En estos casos la dosis recomendada debería ser más elevada de lo normal, debido a que, al ser eliminado el fármaco más rápidamente de lo normal, no hay tiempo suficiente para que éste dé lugar al efecto terapéutico deseado.
El estudio de estas variaciones polimórficas, así como el de qué grupo de fármacos es metabolizado por las diferentes enzimas, conllevará una mejora en los futuros tratamientos de la medicina personalizada. El genotipado de genes relacionados con el metabolismo de fármacos, como los de la familia de citocromos P450, se está convirtiendo en una estrategia útil para predecir los posibles fenotipos que caracterizan una diferente respuesta interindividual a fármacos concretos. No obstante, existen algunas limitaciones en este tipo de estudios. Debido a la alta complejidad de los locus relacionados con los citocromos P450 y la enorme variabilidad genética (incluidos SNPs, pequeñas inserciones o deleciones, variaciones en el número de copias o reordenamientos con genes no funcionales) presente en los diferentes individuos, se hace complicado interpretar e inferir el fenotipo resultante (Gaedigk, 2013). Actualmente, proyectos como HapMap (International HapMap Consortium, 2003) o el de 1.000 Genomas (1000 Genomes Projet Consortium, 2012), pretenden establecer un catálogo de variaciones genéticas comunes presentes en la especie humana, así como cómo éstas se distribuyen y se organizan a lo largo del genoma. De este modo, pueden establecerse haplotipos o regiones cromosómicas de polimorfismos asociados. Este tipo de proyectos será de gran utilidad en un futuro no muy lejano para extraer información valiosa relacionada con el campo de la farmacogenética, que han contribuido colectivamente a la creación de un catálogo de variaciones que ha mejorado nuestra comprensión acerca de la diversidad humana (Al-Ali, 2018). A medida, que la tecnología de genotipado ha progresado, los estudios de asociación de genoma completo (GWAS, Genome-Wide Association Study) han madurado y se postulan como uno de los posibles enfoques prometedores y efectivos para mapear genes subyacentes a fenotipos humanos (Motsinger-Reif, 2013). No obstante, existe alguna limitación para este tipo de estudios, como por ejemplo la necesidad de un tamaño muestral muy grande para extraer conclusiones relevantes. Este tipo de enfoques han puesto de manifiesto la existencia de variaciones genéticas entre etnias, lo que provoca una diferente respuesta a fármacos, no sólo entre individuos de la misma etnia, sino también en los que pertenecen a etnias diferentes (Qi y Zhang, 2017).
También es importante determinar si varios fármacos son sustrato, inductor o inhibidor de la misma enzima, ya que, en esos casos, existirá una interacción entre fármacos que supondría una efectividad variable de éstos en función de la dosis. Pueden darse diferentes combinaciones de fármacos y, fijándonos en el efecto (sustrato, inductor o inhibitorio) que ejercen sobre una enzima determinada, es posible predecir con mayor exactitud la dosis recomendada de cada uno de ellos. En estos casos es necesario un ajuste de la dosis de los fármacos empleados con el fin de optimizar el efecto terapéutico.
La interacción entre fármacos es un aspecto a tener en cuenta en politerapias (administración de más de un fármaco o tratamiento). En el caso de interacciones de dos o más fármacos con la misma enzima, existe la posibilidad de que su correcto funcionamiento se vea inhibido por la competición entre los fármacos (Ahmed, 2016), lo que significa que podría ser de utilidad reducir la dosis de los fármacos, debido a que permanecen más tiempo en el organismo que en monoterapias. Si se analiza la relación entre los fármacos con sus respectivas enzimas metabolizadoras es posible predecir la respuesta que tendrá un individuo tras la administración de éstos y, como consecuencia, ajustar la dosis del tratamiento, lo que traerá consigo una menor susceptibilidad de padecer efectos adversos por parte del paciente. Existen bases de datos como SuperCYP (Preissner et al., 2009) que aportan una amplia gama de información relacionada con enzimas metabolizadoras de la familia de citocromos P450. En ella, podemos hallar información, tanto acerca del tipo de relaciones (sustrato, inducción o inhibición) que se establecen entre los fármacos y las diferentes enzimas, como relativa a la existencia de posibles interacciones entre dos o más fármacos.
Algunos autores sugieren que variaciones polimórficas en algunos receptores pueden contribuir a una diferente respuesta a fármacos. Un ejemplo de ello es el estudio propuesto por Pál et al. (2017). En él se muestra el efecto de los SNPs en el receptor FCGR3A en la población húngara relacionados con artritis reumatoide y linfomas no Hodgkin. En tratamientos con el fármaco genérico rituximab fueron halladas diferencias significativas en la respuesta entre heterocigotos y homocigotos para el polimorfismo en cuestión. Además, se puso de manifiesto el hecho de que los individuos portadores de un alelo concreto presentaban una respuesta más eficaz al tratamiento. Este tipo de estudios sugiere la importancia de las variaciones genéticas en relación a la diferente respuesta a fármacos en individuos, tanto de la misma población como de poblaciones diferentes.
FARMACOGENÓMICA
Además, desde un enfoque farmacogenómico, también podemos estudiar los mecanismos que dan lugar a las diferentes enfermedades. La farmacogenómica es un campo de la genómica que lleva muchos años de progreso. Anteriormente se empleaban indistintamente los términos farmacogenética y farmacogenómica. No obstante existen ciertos matices que los diferencian. Por un lado, como se menciona en el apartado anterior, la farmacogenética se centra en el estudio de cómo las variaciones genéticas afectan a la respuesta a determinados fármacos. Por otro lado, la farmacogenómica estudia las bases moleculares y genéticas de enfermedades para desarrollar nuevas vías de tratamiento (Tello, 2006). El término farmacogenómica emergió por primera vez en el año 1998. No obstante, tras la secuenciación del genoma humano, esta disciplina ha dado un gran salto en el ámbito de la medicina personalizada.
Mediante el estudio de las relaciones que se establecen entre los genes que son diana (target) de determinados grupos de fármacos empleados en una enfermedad, podemos obtener una visión global de ella, con el fin de ser más precisos en la obtención de futuras dianas moleculares. Examinando las rutas moleculares (pathways) en las que participa un fármaco determinado es posible obtener una perspectiva global de cómo se regula un proceso celular determinado. Partiendo de la premisa de que existen multitud de relaciones entre diferentes genes, es posible determinar el tipo de relaciones existente entre ellos para poder desenmarañar la intrincada red molecular que regula un proceso celular determinado. Con este tipo de información podemos plantear un modelo top-down en el que se concrete cada vez más la importancia del tipo de relaciones que se dan entre los diferentes genes, para así poder, en un futuro, diseñar dianas moleculares más eficientes para una determinada enfermedad.
La extracción de información para el estudio de un proceso celular determinado no es trivial. Es necesario partir de una información lo más curada y fiable posible, con el fin de extraer conclusiones con la mayor relevancia clínica posible. Por ello, es crucial discriminar entre la información que proporciona una base de datos y la que proporciona otra.
Son muchas las bases de datos que existen en la actualidad. Sin embargo, no todas ellas ofrecen el mismo tipo de información. Una de las bases de datos ampliamente empleadas en investigación relacionadas con fármacos y rutas moleculares es KEGG (Kyoto Encyclopedia of Genes and Genomes) (Kanehisa, 1996). KEGG se divide a su vez en varias bases de datos en función de la información que ofrece. Entre ellas podemos encontrar KEGG DRUG, que muestra información acerca de dianas moleculares de diferentes fármacos, así como propiedades fisicoquímicas de éstos. En KEGG también podemos encontrar la base de datos de KEGG PATHWAY, la cual ofrece información acerca de los pathways moleculares que actúan en procesos celulares determinados. Mediante la integración de la información de ambas bases de datos es posible elaborar una red que muestre información acerca de las dianas moleculares de grupos de fármacos que actúan en una ruta determinado y que mostraría también el tipo de relaciones que se establecen entre ellas.
Tras la selección de la información más adecuada para el estudio, es necesaria la integración de ésta mediante herramientas informáticas. Para ello, existen softwares de programación informáticos como R (R development Core Team, 2008, disponible en https://cran.r-project.org) o Python (Python Software Foundation. Python Language Reference, disponible en http://www.python.org). Estos programas cuentan con un entorno y lenguaje de programación enfocado al análisis estadístico y ampliamente empleado en investigación. Tras integrar la información extraída de las bases de datos mediante esta herramienta, se construye la red (en la que se representan las relaciones entre las dianas moleculares de los fármacos escogidos) mediante otro tipo de herramientas informáticas. Para este cometido existen softwares como Cytoscape (Shannon et al., 2003). Esta red que muestra las relaciones entre dianas de fármacos puede ser estudiada en diferentes contextos, como el de expresión génica en tejidos relevantes para nuestro estudio. El estudio de las relaciones entre dianas y cómo éstas se expresan en un tejido determinado conllevará un mejor conocimiento acerca de las bases moleculares de enfermedades y una aplicación clínica para el desarrollo de nuevas dianas moleculares.
BIOINFORMÁTICA, BIOLOGÍA DE SISTEMAS Y REDES BIOLÓGICAS
Con la secuenciación del genoma humano tenemos la posibilidad de obtener un mejor conocimiento de las bases moleculares de diferentes enfermedades, así como de los mecanismos que subyacen a diferentes respuestas celulares. No obstante, pese a la cantidad de información de que disponemos, se hace complicado analizarla e integrarla para extraer una visión global de determinados procesos biológicos. Las herramientas informáticas son imprescindibles para tratar de dar solución a este problema. Asimismo, las bases de datos nos facilitan el almacenamiento de esta información, lo que permite a los investigadores acceder a ella de un modo ordenado y sencillo en la mayoría de los casos. Las bases de datos biológicas son librerías de información de ciencias de la vida provenientes de experimentos científicos, publicaciones científicas, experimentos con tecnología de alto rendimiento y análisis computacionales (Attwood et al., 2011). Para llevar a cabo estudios con la mayor fiabilidad posible, es necesario discernir ente los datos que son relevantes para nuestro estudio y los que no, así como entre información que ha sido curada de un modo más o menos minucioso. Por ello, la extracción de información de partida es de enorme relevancia para el futuro del proyecto.
Disciplinas como la biología de sistemas se nutren de todos estos datos generados a gran escala para obtener una visión en conjunto de un determinado proceso celular. La biología de sistemas trata de dar explicación a la organización biológica completa y a los procesos que constituyen las bases moleculares de dicha organización biológica. Una de las herramientas más empleadas en la biología de sistemas corresponde a la visualización, análisis e interpretación de redes biológicas, denominada teoría de grafos. Mediante esta teoría es posible representar esquemáticamente interacciones complejas que tienen lugar entre las diferentes entidades de un sistema. Matemáticamente, una red de relaciones (Figura 1) está representada por un grafo. Conceptualmente, un grafo está formado por los vértices o nodos, los cuales representan entidades biológicas (enfermedades, genes, proteínas o metabolitos podrían ser algunos de los casos) y aristas, que representan las relaciones que se establecen entre las distintas entidades biológicas y que conectan dichos nodos.

Las redes proporcionan vistas globales de las relaciones entre los nodos y, por tanto, permiten que las propiedades de la red se calculen en función de las conexiones de un nodo y su relación con el resto de la red (Berger e Iyengar, 2009). Por ejemplo, cada nodo tiene un grado, la cantidad de nodos con los que interactúa. Los estudios de muchas redes biológicas, a menudo interacción proteína-proteína o redes reguladoras de genes, se han centrado en la distribución de grados de la red global y encontraron que a menudo hay nodos que se conectan a, relativamente, muchos otros nodos (Barabasi y Albert 1999; Jeong et al., 2000). Éstos son a menudo importantes para el funcionamiento de procesos biológicos múltiples (Jeong et al., 2001). De manera similar, se han estudiado varias medidas diferentes de la centralidad del nodo para asignar valor a un nodo en función de su importancia relativa en una red (Jeong et al., 2001; Jovelin y Phillips, 2009; Vinogradov, 2009; Wang et al., 2008). Además de los estudios globales de redes celulares, el análisis de redes ha demostrado ser útil para estudios más enfocados que investigan una enfermedad o patofisiología particular. Se ha demostrado que las proteínas que interactúan entre sí están, frecuentemente, involucradas en un proceso biológico común (Luo et al., 2007). De manera similar, las proteínas interactuantes pueden estar involucradas en un proceso de enfermedad (Goh et al., 2007; Ozgur et al., 2008).
En la presente revisión se pretende proporcionar una visión holística o global de los mecanismos moleculares de respuesta a fármacos en diferentes enfermedades. Para ello, se emplean algunas herramientas bioinformáticas y, partiendo de una metodología basada en la biología de sistemas, podemos aspirar a obtener un mejor conocimiento de estos mecanismos moleculares, así como de las relaciones que se producen entre los componentes de un sistema determinado.
Anteriormente, la mayoría de los estudios se centraban en el estudio de la acción determinada de un gen, obviando la importancia que tienen las diferentes relaciones que se establece entre éste y otros componentes de un sistema. Por ello, se hace necesaria una mejor comprensión, no sólo del modo de acción de un gen concreto, sino también de los diferentes mecanismos que dan lugar a la acción de ese gen y de las diferentes relaciones con otros genes. Es posible obtener un mejor conocimiento de los mecanismos de respuesta a fármacos, desde una visión global, afrontándolo desde diferentes puntos de vista. Algunos de ellos, como se tratará en esta revisión, se basan en las relaciones que se establecen entre los genes diana de diferentes fármacos empleados en alguna enfermedad concreta o en las relaciones producidas entre grupos de fármacos y sus enzimas metabolizadoras.
En función de los diferentes puntos de vista desde los que se puede estudiar la respuesta a fármacos podemos obtener unos resultados u otros. El estudio de fármacos implicados en diferentes enfermedades y el tipo de relaciones que se establecen entre ellos, puede proporcionar información relevante, que diferirá dependiendo de la aproximación empleada.
El estudio de los efectos secundarios provocados por determinados grupos de fármacos es un posible enfoque para determinar los mecanismos moleculares de algunas enfermedades (Figura 2). Por ejemplo, muchos fármacos pueden causar arritmias cardíacas al bloquear los canales de potasio en el corazón, a pesar de que éste no era el objetivo principal o la indicación de estos medicamentos (Hoffmann y Warner, 2006). Los estudios de fármacos en la red han permitido la identificación de algunas de estas dianas secundarias de los fármacos. Campillos y colaboradores (2008) construyeron una red de fármacos conectados en función de si compartían, tanto un grado de similitud estructural, como perfiles de efectos secundarios similares.
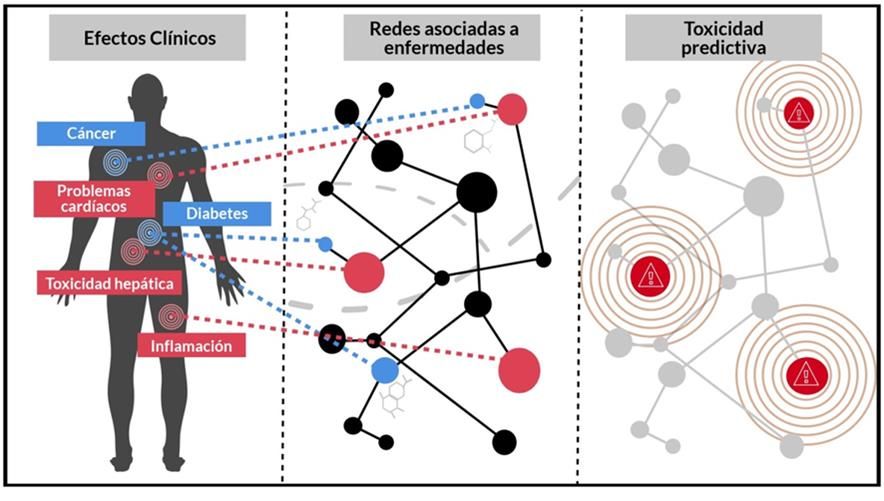
Otra forma en que los objetivos farmacológicos se pueden vincular en una red implicaría un enfoque quimioinformático (Figura 1). Keiser et al. (2007) han desarrollado un método de puntuación de la similitud entre los conjuntos de ligandos para diferentes receptores. Los autores utilizan esta puntuación para construir una red de receptores conectados entre sí si se unen ligandos estructuralmente similares. Este análisis mostró que muchas dianas farmacológicas relacionadas biológicamente se agruparon por similitud de ligandos, aunque los objetivos mismos tienen una similitud de secuencia mínima. Usando este enfoque (Figura 3), los autores hicieron predicciones específicas, como sugerir que la metadona interactúa con los receptores Muscarinic-3, y validaron experimentalmente las predicciones. Este tipo de análisis, basado en la capacidad de diferentes objetivos para unir el mismo ligando, permitió la identificación de objetivos no seleccionados para ciertos fármacos.
Existen hoy en día gran cantidad de bases de datos relacionadas con los efectos secundarios que producen diferentes fármacos. Una de ellas es Drugs.com (https://www.drugs.com/sfx/), que muestra información, tanto de efectos secundarios provocados por fármacos, como de las dosis recomendadas y contraindicaciones para embarazadas. Mediante la extracción y correcta clasificación de los datos pertenecientes a los efectos secundarios producidos por un fármaco determinado, podemos obtener un mejor conocimiento acerca del modo de acción del mismo. Para ello es necesario un proceso de estandarización y análisis de los datos, con el fin de obtener una información lo más fiable y relevante posible desde el punto de vista de la investigación.
La integración de todo este tipo de información mediante herramientas informáticas y el posterior análisis, desde un enfoque global, es clave para extraer una información lo más valiosa y relevante posible, enfocada a la búsqueda de una medicina personalizada y a una aplicación clínica factible.
CONTEXTO CIENTÍFICO ACTUAL
A pesar del desarrollo y puesta a punto de técnicas tradicionales, como el estudio de la función que posee un gen concreto sobre una enfermedad determinada, hoy día los esfuerzos se enfocan cada vez más en el uso de modelos matemáticos y herramientas informáticas para predecir diferentes respuestas celulares y realizar patrones de predicción. Debido al menor coste económico se hace más factible cada día este tipo de enfoques, en detrimento de otros más clásicos como los anteriormente mencionados. A su vez, el uso de este tipo de herramientas nos proporciona una visión global de un determinado tipo de enfermedad, por lo que, mediante otras técnicas más tradicionales, podríamos obviar información de gran relevancia.
Actualmente, son bastantes los proyectos relacionados con este tipo de técnicas, que intentan desenmarañar los procesos celulares que subyacen a alguna enfermedad concreta o a la respuesta de determinados fármacos empleados en ésta.
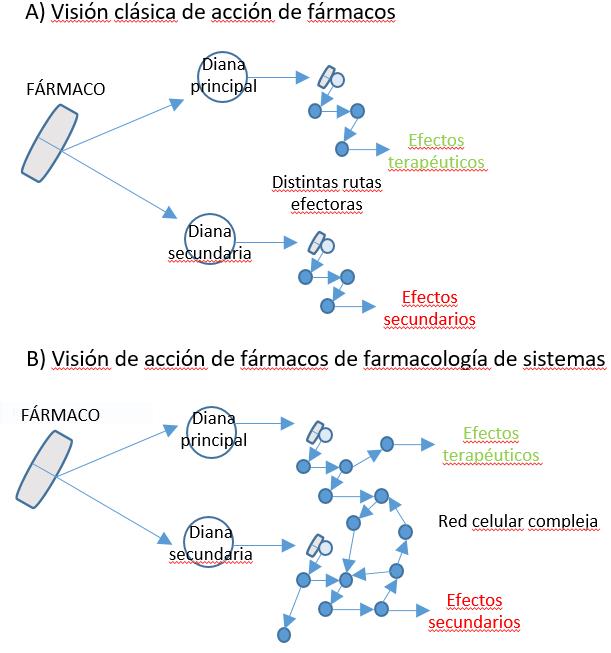
La farmacología de sistemas implica la aplicación de enfoques de biología de sistemas, que combinan estudios experimentales a gran escala con análisis computacionales, para el estudio de fármacos, dianas farmacológicas y efectos farmacológicos. El estudio de los fármacos en el contexto de las redes celulares, nos proporciona información sobre los eventos adversos causados por los objetivos no previstos de los mismos, así como las respuestas complejas de la red, mediada por eventos adversos. Esto permite la identificación rápida de biomarcadores para la susceptibilidad a los efectos secundarios. De esta forma, la farmacología de sistemas dará lugar, no sólo a terapias más efectivas, sino a medicamentos más seguros y con menos efectos secundarios (Berger e Iyengar, 2011).
El estudio de las proteínas de la familia de citocromos P450 también está en auge actualmente, ya que el conocimiento acerca del metabolismo de fármacos también es de especial interés para el desarrollo de nuevos tratamientos en medicina personalizada. Son diversos los proyectos relacionados con este tipo de enfoques. Existen estudios relacionados con la respuesta a fármacos enfocados en la funcionalidad de los citocromos P450. En ellos, se observa el efecto de un fármaco determinado prescrito para una enfermedad concreta. Como se muestra en la revisión de Wang y colaboradores (2011), los efectos secundarios provocados por un fármaco están, en gran medida, relacionados con el deficiente metabolismo de éstos. En la revisión se centran en explicar la relación entre el hecho de compartir enzimas metabolizadoras comunes y la existencia de efectos adversos. Los autores examinan el efecto adverso provocado por fármacos empleados en enfermedades cardiovasculares, agentes empleados para enfermedades infecciosas, fármacos antineoplásicos e inhibidores de la aromatasa (enzima fundamental en la biosíntesis de estrógenos). Para ello, realizan un estudio comparativo entre pacientes genotipados para enzimas implicadas en el metabolismo de los fármacos que le eran prescritos y pacientes control (sin genotipado)y se examina la diferencia entre un tipo de paciente y otro en función del riesgo de hospitalización, prestando especial atención a los síntomas provocados por los efectos secundarios del fármaco recetado. Los resultados del estudio muestran diferencias significativas en pacientes genotipados respecto a los pacientes control.
Por ello, los futuros estudios de farmacología deberían estar orientados al estudio del genotipo de genes relacionados con el metabolismo de fármacos, con el fin de ajustar la dosis necesaria de un fármaco en función del fenotipo del paciente.
Algunos proyectos de investigación están enfocados al estudio de la relación entre la posición de la diana molecular de un fármaco determinado en la red y los efectos secundarios provocados por el fármaco. En el estudio de Wang y colaboradores (2013) se examinan las relaciones de 4.199 efectos secundarios asociados a 996 fármacos y sus 647 dianas moleculares. En este caso, la información acerca de los fármacos y su efecto secundario asociado correspondiente fue extraída de la base de datos de PubChem (Wang et al., 2009) y SIDER2 (Kuhn et al., 2010), respectivamente. Los datos de las dianas moleculares de los fármacos escogidos fueron extraídos mediante el uso de la ID de UniProt disponible. Para encontrar factores clave que contribuyan a la incidencia de efectos secundarios se realizó un estudio estadístico consistente en regresión lineal generalizada. A continuación, se elaboró una red (Figura 4) en la que se muestran las proteínas diana de los fármacos y las interacciones compartidas, calculadas mediante el coeficiente de Jaccard. Wang y colaboradores (2013) examinan en este estudio la importancia de la centralidad y grado de proximidad en la red de una diana molecular y su relación con los efectos secundarios provocados por un fármaco determinado.
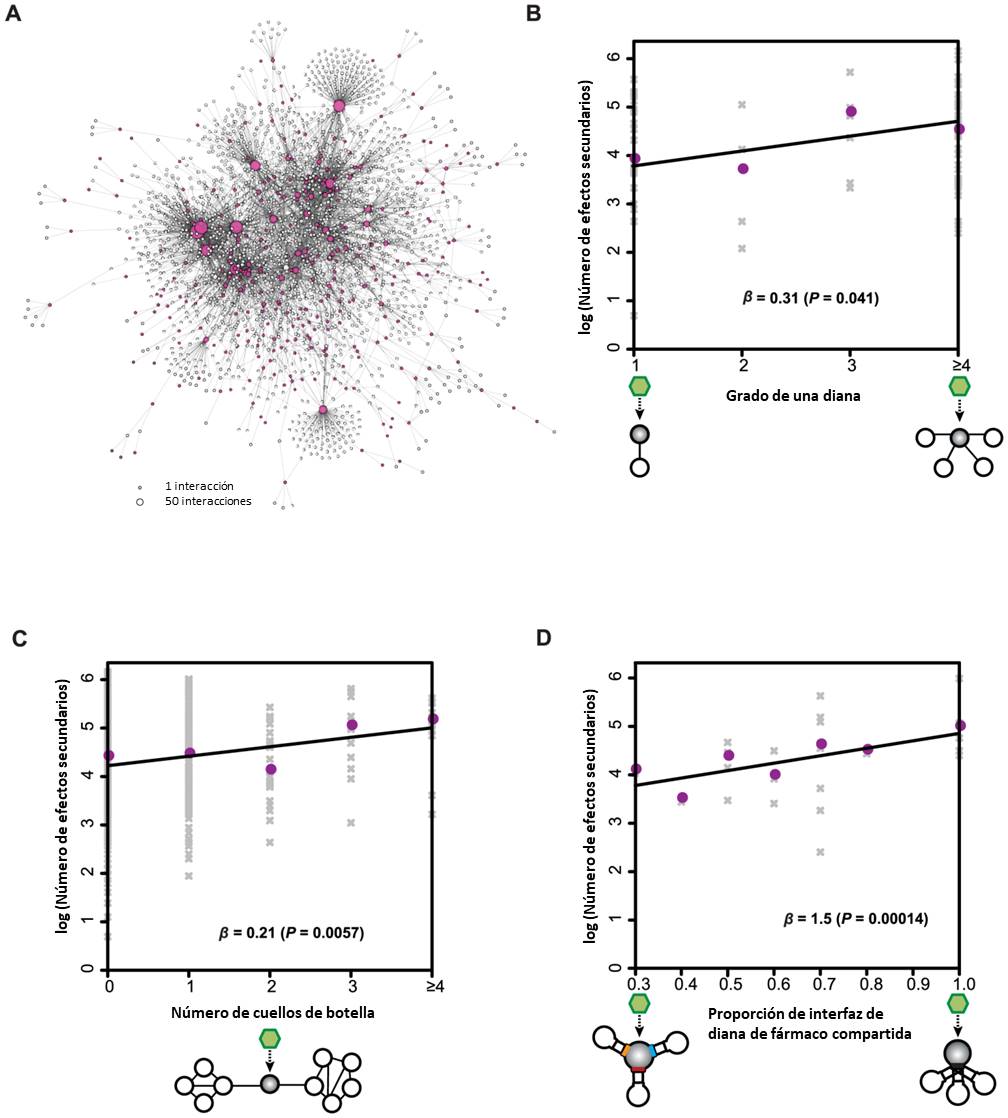
Existen otros estudios en los que se pretende conocer la asociación entre fármaco, proteína y efectos secundarios, como es el caso del estudio de Liu y Altman (2015) (Figura 5). En este proyecto se centran en predecir la unión de fármacos a dianas moleculares de baja afinidad. Es decir, dianas moleculares que, teóricamente, no serían de gran afinidad para el fármaco. Para evaluar estos posibles objetivos alternativos, emplean estructuras tridimensionales de 563 proteínas humanas esenciales para predecir su unión a 216 fármacos. Para ello, se combina la descomposición de valores singulares y el análisis de componentes canónicos (SVD-CCA, Singular Value Decomposition-Canonical Correlation Analysis), con el fin de predecir los efectos secundarios basados en estos nuevos perfiles de dianas moleculares.

La integración de todo este tipo de aproximaciones es un punto de partida para un mejor conocimiento de los mecanismos moleculares que actúan en la aparición de diferentes enfermedades. Este tipo de percepción, en la que se tiene en cuenta, no sólo la acción de un gen determinado, sino del conjunto de interacciones que se producen entre los diferentes componentes del sistema, traerá como consecuencia una mejora en la comprensión de las diferentes enfermedades, así como de la respuesta a determinados fármacos.
CONCLUSIONES Y APLICACIONES CLÍNICAS
La construcción de redes moleculares que muestren las conexiones entre diferentes componentes es de gran importancia para estudiar los mecanismos moleculares que subyacen a diferentes enfermedades. Examinar este tipo de relaciones, mediante la integración de diferentes perspectivas, nos llevará a una mejor comprensión de determinados procesos celulares y guiará las futuras investigaciones biomédicas hacía una búsqueda de hipótesis más factibles o viables para una posterior aplicación clínica, que más adelante pueden ser validadas mediante procedimientos experimentales.
La extracción de información de partida para el proyecto es de gran importancia, ya que todas nuestras conclusiones se basarán en todos estos datos. Es por ello, que es crucial partir de una información lo más curada y fiable posible. Este proceso puede ser llevado a cabo mediante una comparación sistemática entre las diferentes bases de datos a disposición de la investigación biomédica. La cantidad de información de la que disponemos crece a un ritmo vertiginoso, por lo que se hace necesario extraer y analizar ésta de un modo minucioso y con la máxima precaución posible. En función del enfoque del estudio, se valorarán las bases de datos relacionadas con éste.
La biología de sistemas y en este caso, la farmacología de sistemas, se nutre de toda la información extraída para analizarla desde una perspectiva global y poder dar explicación a determinados procesos celulares. Las redes moleculares nos permiten visualizar esta información de un modo relativamente sencillo, donde se muestran los tipos de relaciones que se establecen entre las diferentes moléculas objeto de estudio. La elaboración y estudio de estas redes puede ser llevada a cabo desde diferentes puntos de vista o enfoques. La posterior integración de todos estos enfoques supondrá la ventaja de poseer más información acerca de un proceso determinado que, más adelante será analizada y sintetizada mediante el uso de herramientas informáticas. Este tipo de estudios se basan en estrategias de procesamiento de información denominados top-down, es decir, de arriba abajo. Así se descartará la información sin relevancia o de poca relevancia para el proyecto en cuestión y podremos centrarnos en los detalles de importancia que posteriormente pueden ser analizados de un modo experimental en el laboratorio.
Dadas las múltiples formas en que un fármaco puede causar un efecto secundario, es poco probable que un único enfoque pueda predecir todos los tipos de efectos secundarios para todos los fármacos, así como el tipo de respuesta de un paciente a un tratamiento determinado. Sin embargo, el enfoque basado en la biología de sistemas nos permite identificar nuevas dianas terapéuticas mediante la exploración de las dianas moleculares conocidas en el contexto de sus interacciones. Actualmente, con la cantidad de información de que disponemos, se hace necesario un enfoque multidisciplinar que conecte todas las piezas del rompecabezas de un proceso biológico concreto.
Gracias a enfoques basados en farmacogenómica y farmacogenética, organismos como la FDA han realizado modificaciones en las etiquetas de muchos fármacos conocidos. Parece que estas disciplinas se postulan, junto con otras, como adecuadas para el desarrollo de una medicina personalizada. El estudio de la diferente respuesta a fármacos y los efectos nocivos provocados por éstos, es uno de los primeros pasos para el desarrollo de nuevas dianas moleculares y terapias individualizadas.
AGRADECIMIENTOS
A PGA le gustaría agradecer a la línea de investigación de Medicina Genómica Reproductiva y de Sistemas, dirigida por la Dra. Patricia Díaz Gimeno, la posibilidad de participar en sus proyectos de investigación, así como el trato recibido, en especial, por parte de Patricia Sebastián León y por Fundación IVI.
