INTRODUCCIÓN
Los hongos filamentosos son organismos eucariotas cosmopolitas, ya que poseen una amplia distribución en el medio ambiente. Desde tiempos ancestrales, en la cultura oriental, los hongos filamentosos se han empleado como materia prima para la elaboración de productos alimenticios fermentados de sabor y de valor nutritivo agregados (Van der Strat et al., 2014; Michelson et al., 2010; Schuster et al., 2002; Nødvig et al., 2015; Egbuta et al., 2016). Actualmente, los hongos filamentosos se utilizan ampliamente como fuentes de compuestos bioactivos, pigmentos, proteínas y enzimas en diversos sectores industriales alimentarios y no-alimentarios, para la obtención de nuevas fuentes de bioenergía (bioetanol), desarrollo de nuevos biomateriales a partir de su micelio (Mycelium Technology), como medios de control biológico de plagas y enfermedades en la agricultura sostenible, así como también en procesos de micorremediación de suelos contaminados con hidrocarburos recalcitrantes, entre otros compuestos orgánicos tóxicos y contaminantes (Naranjo-Briceño et al., 2019). Aspergillus niger, Aspergillus oryzae, Trichoderma reesei y Penicillium spp., son los géneros de hongos filamentosos que más se emplean en la industria (Tabla 1). Además de poseer una poderosa maquinaria enzimática altamente activa y de fácil transporte al medio extracelular, estos microorganismos son considerados como GRAS (por sus siglas en inglés, Generally Recognized as Safe) por la FDA (por sus siglas en inglés, Food and Drug Administration, USA), por lo que su uso es seguro para la producción de metabolitos y enzimas destinados para el uso alimenticio humano. Por los motivos antes mencionados, ha existido la necesidad de contar con las herramientas técnicas apropiadas para estudiar y aprovechar las capacidades metabólicas de los hongos filamentosos y descubrir nuevas moléculas con interesantes aplicaciones biotecnológicas (Deng et al., 2017a).
Se han descrito diversas herramientas moleculares para manipular el genoma de los hongos filamentosos, entre las que se incluyen la recombinación homóloga, etiquetado de transposones y la integración mediada por agrobacterias. Sin embargo, los métodos mencionados presentan ciertas limitaciones, asociadas con la recombinación homóloga ineficiente y la reducida cantidad de marcadores de selección que requieren pasos adicionales para el reciclaje de los mismos. Con respecto al marcaje de transposones, no se puede controlar la manipulación dirigida y se obtiene una gran cantidad de transformantes, mientras que en la integración mediada por agrobacterias influyen varios factores en la eficiencia de este método, principalmente, el tipo de material fúngico inicial (protoplastos, esporas o hifas) (Wang et al., 2017; Schuster et al., 2019; Li et al., 2017). Además, se pueden generar complicaciones para determinar la función de genes en hongos filamentosos diploides y multinucleados porque se requiere la mutación de todos los alelos para determinar cómo afecta el gen de interés en el desarrollo de un determinado fenotipo (Wang y Coleman et al., 2019).
Entre las tecnologías de edición de genomas también se incluyen las proteínas diseñadas para modificar loci específicos, como las nucleasas dedos de zinc (ZFNs) (por sus siglas en inglés, zinc-finger nucleases) y las nucleasas efectoras de transcripción similares a activadores (TALENs) (por sus siglas en inglés, transcription activator-like effector nucleases), siendo éstas últimas implementadas en la edición genómica de hongos filamentosos (Liu et al., 2017; Wang et al., 2017). No obstante, estas tecnologías presentan dificultades en los procesos de diseño, síntesis y validación de las proteínas que reconocen los loci específicos de interés (Doudna y Charpentier, 2014), y requieren una cantidad considerable de tiempo para su implementación (Song et al., 2019; Deng et al., 2017a), por lo que éstas tecnologías no han sido ampliamente aplicadas en la edición de genomas de hongos. Además, ZFN y TALEN no tienen la capacidad para editar múltiples genes a la vez (Song et al., 2019).
Todas estas dificultades para la edición de genomas han sido superadas con el surgimiento y reciente aplicación del sistema de edición de genomas CRISPR/Cas9. En comparación con ZFN y TALEN, CRISPR/Cas9 posee la capacidad de editar múltiples genes a la vez, mejorando la eficiencia en la edición de genomas, porque a partir de una sola transformación se pueden generar mutantes que poseen múltiples loci editados. Además, CRISPR/Cas9 es un sistema que está conformado por componentes simples, permitiendo que su aplicación sea rápida y versátil (Doudna y Charpentier, 2014; Deng et al., 2017a). Por el desarrollo de la técnica CRISPR/Cas9 como un método para la edición del genoma, las investigadoras Emmanuelle Charpentier y Jennifer A. Doudna, han recibido el Premio Nobel de Química de 2020.
Desde que la técnica CRISPR/Cas9 llegó a la comunidad científica, se han expuesto diferentes estrategias para la edición de genes en hongos filamentosos (Nødvig et al., 2015). Las estrategias difieren en el uso de promotores tanto para la expresión heteróloga del gen CAS9 como del sgRNA, así como también difieren en el uso de marcadores de selección de transformantes positivos. El objetivo del presente artículo de revisión es precisamente describir las últimas tendencias hasta la fecha en el uso de promotores (constitutivos e inducibles) para la expresión heteróloga de la proteína Cas9 y del sgRNA, y en los marcadores de selección de transformantes o cotransformantes.
Tabla 1. Principales hongos filamentosos empleados como biofactorías en la industria (continúa en siguiente página).
| Hongo filamentoso | Tipo de Biomolécula | Aplicación | Referencias | ||
| Salud | Agricultura / Ambiente | Industria | |||
| Aspergillus flavus | Uricasa, lipasa, proteasa | Gota | Biorremediación, biocontrol | Detergentes, elaboración de cueros | Singh et al., 2016; Kurniati et al., 2014; Abbas et al., 2011 |
| A. niger | Glucoamilasa, ácido cítrico, ácido glucónico, pythase | NA | Biorremediación | Cosméticos, adhesivos | Shah et al., 2017; Tang et al., 2015; Majumder et al., 2010; Schuster et al., 2002 |
| A. oryzae | Ácido kpojico, α-amilasa, proteasa, lipasa, | Desórdenes digestivos, antioxidantes | NA | Cosméticos, alimentos, detergentes | Singh et al., 2016; Ogawa et al., 1995; Francis et al., 2003 |
| A. terreus | Ácido itacónico, lovostatina, | Antibióticos | NA | Polímeros sintéticos, biocombustible | Van der Strat et al., 2014; Jahromi et al., 2012; Dwiarti et al., 2007; Pushalkar y Rao, 1998 |
| Blakeslea trispora | Caroteno, licopeno | NA | Alimentación, Textil | NA | Mantzouridou y Naziri, 2017; Nanou y Roukas, 2016 |
| Botrytis cinerea | Ácido abcísico | NA | NA | NA | Marumo et al., 1982 |
| Cunninghamella echinulata | γ-Ácido linoleico | NA | NA | NA | Fakas et al., 2008 |
| Fusarium fujikuroi | Giberelina | NA | NA | NA | Shi et al., 2017 |
| F. oxysporum | Bioetanol, clicosporina | Antibióticos | Biocontrol | Biocombustible | Sharmila et al.,
2012; Kaur et al., 2010; Panagiotou et al., 2004 |
| F. verticillioides | Bioetanol | NA | NA | Biocombustible | de Almeida et al., 2013 |
| Myceliophthora thermophila | Celulasa | NA | NA | Biocombustible | Liu et al., 2017 |
| Monascus spp. | Pigmentos naturales: monascina, ankaflavina, rubropunctatina, monascorubrina | Anticancerígeno | Alimentación | Textil | Xiong et al., 2014; Chen et al., 2017a |
| Mortierella alpina | Ácido araquidónico | Ji et al., 2014 | |||
| M. isabellina | γ-Ácido linoleico | Mo et al., 2002 | |||
| M. ramanniana | γ-Ácido linoleico | Dyal et al., 2005 | |||
| Mucor circinelloides | γ-Ácido linoleico | Zhang et al., 2017 | |||
| Neurospora crassa | Bioetanol | Biocombustible | Davis y Perkins, 2002; Colvin et al., 1973 |
PRINCIPIOS Y COMPONENTES DEL SISTEMA CRISPR/Cas9
El sistema CRISPR/Cas tiene su origen en los loci CRISPR-Cas de bacterias y arqueas, en las que funciona como un sistema de inmunidad adaptativa mediante el cual estos microorganismos se defienden natural y espontáneamente de los virus. Cada locus está estructurado principalmente por secuencias cortas repetitivas de ADN, intercaladas por espaciadores, y por un operon de genes cas codificantes de componentes proteicos Cas (Doudna y Charpentier, 2014). Las secuencias espaciadoras incorporadas en los loci provienen de material genético de virus que han causado infecciones anteriores. El ARN precursor asociado a CRISPR (pre-crRNA) es formado a partir de la transcripción de las secuencias repetitivas en conjunto con los espaciadores, que posteriormente al proceso de maduración, forman ARNs asociados a CRISPR (crRNAs) listos para generar una escisión específica al ADN invasor gracias a la actividad catalítica de proteínas Cas. Esto ocurre en sitios específicos de complementariedad del crRNA con el ADN invasor.
Estudios recientes sugieren dos clases y seis tipos de sistema CRISPR/Cas. El sistema de clase 1 incluye los tipos I, III y IV, mientras que el sistema de clase 2 incluye los tipos II, V y VI. De estos sistemas, los de clase 2 tipo II tienen un uso más extendido en la comunidad científica, debido a la simplicidad de su mecanismo (Makarova et al., 2015; Shmakov et al., 2017).
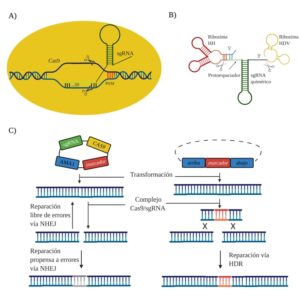
El sistema de CRISPR/Cas de tipo II está compuesto por la proteína Cas9 (nucleasa), el crRNA maduro, el trans-activador de crRNA (tracrRNA) y la RNasa III. Este sistema fue optimizado para constituir un sistema de dos componentes principales (Jinek et al., 2012), tal como muestra la Figura 1A: una endonucleasa Cas9 y un RNA guía (sgRNA, por sus siglas en inglés, single guide RNA) formado por la fusión del crRNA maduro con el tracrRNA. La proteína Cas9, aislada originalmente de la bacteria Streptococcus pyogenes, presenta actividad endonucleasa debido a sus dos distintos dominios catalíticos, un dominio de nucleasa HNH, que permite escindir la cadena de ADN complementaria a la secuencia del ARN guía, y un dominio de nucleasa tipo RuvC, responsable de escindir el ADN de cadena opuesta a la complementaria (Chen et al., 2014). El sgRNA está conformado por una secuencia de andamiaje (scaffold) en el extremo 3’ y un espaciador de alrededor de 20 nucleótidos en el extremo 5’ que define el objetivo en la secuencia de ADN (Jinek et al., 2012).
El complejo de ribonucleoproteína (RNP) CRISPR/Cas9 se forma cuando ambos componentes se unen a través de las interacciones de la secuencia de andamiaje del sgRNA con la superficie cargada positivamente de la proteína Cas9. Esta unión provoca un cambio conformacional en Cas9 que la activa y permite la unión del sgRNA a la secuencia objetivo de ADN complementario (Jinek et al., 2014). Corriente arriba de la secuencia objetivo, se ubica el motivo adyacente al protoespaciador PAM (por sus siglas en inglés, Protospacer Adjacent Motif). La unión de la proteína Cas9 en la secuencia objetivo ocurre a través de una señal de unión que es mediada por PAM, la cual es una secuencia altamente conservada que presenta una longitud de alrededor de 3 a 5 nucleótidos. Esta secuencia es NGG para la Cas9 de S. pyogenes (Gasiunas et al., 2012). Una vez reconocido este motivo adyacente, la proteína Cas9 sufre un segundo cambio conformacional permitiendo unir los dominios de la endonucleasa al ADN objetivo y escindirlo. El producto de la escisión del complejo CRISPR/Cas9 es una ruptura de la doble cadena del ADN objetivo (DSB, por sus siglas en inglés Double-Strand Break) (Nishimatsu et al., 2014) que activa dos mecanismos de reparación de ADN de doble cadena (dsDNA).
La Figura 1B muestra los mecanismos de reparación del dsDNA, los cuales se realizan mediante la unión de extremos no homólogos (NHEJ, por sus siglas en inglés, Non-homologous End Joining) o mediante la vía de reparación dirigida por homología de alta fidelidad (HDR, por sus siglas en inglés, Homology-Directed Repair) (Sander y Joung 2014). El mecanismo de reparación NHEJ inserta y deleciona nucleótidos (INDELs) aleatoriamente hasta conseguir rellenar y aparear nuevamente los dos extremos de las cadenas y, finalmente, restaurar/ligar el dsDNA escindido por Cas9. Al haber insertado/delecionado nucleótidos de manera aleatoria, la secuencia del gen se habrá alterado, provocando la inactivación del gen (vía mutación NHEJ). En contraste, el sistema de reparación HDR es más eficiente, pudiendo editar el DSB empleando secuencias molde de dsDNA externas que presentan en sus extremos 5’ y 3’ fragmentos homólogos que flanquean a la secuencia de ADN de interés u objeto de estudio. Es decir, este sistema facilita la introducción de secuencias de dsDNA previamente inexistentes en el genoma, lo cual permite tanto incorporar mutaciones específicas como corregirlas (vía edición-substitución), así como introducir fragmentos de ADN homólogos o heterólogos de gran tamaño que portan información de interés. El mecanismo de reparación NHEJ puede ocurrir en células que se encuentran o no en división celular, mientras que el mecanismo HDR sólo ocurre en células en división celular, lo cual constituye una limitación en este mecanismo de reparación de ADN (Wang y Coleman, 2019).
La edición de genomas en hongos filamentosos mediado por endonucleasas Cas9 tipo II es de uso extendido. Sin embargo, estudios recientes han incorporado el uso de endonucleasas tipo V Cpf1 en hongos filamentosos, las cuales han sido acopladas en la edición de genomas de manera exitosa (Jiménez et al., 2020; Vanegas et al., 2019). A diferencia de las endonucleasas Cas9, las Cpf1 reconocen regiones PAMs ricas en timina (Zetsche et al., 2015; Kim et al., 2017) y poseen arreglos de crRNAs que no dependen de tracrRNAs (Zetsche et al., 2015) para la rotura de ADN de las secuencias objetivo. Estas características permiten simplificar el diseño de los sistemas CRISPR y ampliar, en teoría, los sitios de modificación no disponibles para Cas9. Las ventajas de estas endonucleasas tipo V Cpf1 aún deben seguir siendo estudiadas en la edición de genomas en hongos filamentosos (Fu et al., 2019).
Los sistemas CRISPR/Cas9, a pesar de su alta eficiencia, pueden presentar dificultades por la inespecificidad del sistema para sitios que comparten secuencias homólogas con la secuencia objetivo, produciendo modificaciones en genes diferentes al de interés, denominadas off-targets (Zhang et al., 2015). Para reducir la posibilidad de generar off-targets, han surgido modificaciones en el sistema CRISPR en las cuales se generan mutantes de Cas9 denominadas nickasas Cas9 (Cas9n), que tienen uno de los dos dominios catalíticos inactivos. Cas9n genera una rotura simple de ADN con el objetivo de inducir principalmente el mecanismo de reparación HDR, reduciendo de esta manera mutaciones off-targets (Cong et al., 2013). El sistema editor de base dependiente de CRISPR/Cas9 fue aplicado exitosamente en el hongo A. Niger, fusionando la función citidina desaminasa con una Cas9 nickasa para inactivar el gen auxótrofo de uridina pyrG y el gen de pigmento fwnA (Huang et al., 2019). Por otro lado, versiones modificadas de Cas9 han sido creadas para mejorar la especificidad en la edición genómica, tales como eSpCas9 (Slaymaker et al., 2016) diseñada para tener interacciones débiles entre Cas9 y sitios off-targets, y Cas9-HF1 (Kleinstiver et al., 2016), que evita el contacto no específico con regiones off-targets. La versión eSpCas9 ha sido utilizada de manera exitosa en el hongo Sporisorium scitamineum para el silenciamiento del gen Mfa2 (Lu et al., 2017) mientras que Cas9-HF1 aún no ha sido implementada en hongos filamentosos.
En relación al sistema CRISPR/Cas9, el diseño in silico del sgRNA es un paso determinante para disminuir el riesgo por off-targets. Actualmente, varias plataformas bioinformáticas cuentan con diferentes algoritmos destinados a diseñar sgRNA con el menor número de off-targets posible, sin embargo, no todos incluyen genomas de hongos filamentosos en sus bases de datos. La comprensión del mecanismo de acción del sistema CRISPR/Cas9, y el creciente aumento de genomas secuenciados gracias al desarrollo de nuevas tecnologías de secuenciación como la secuenciación masiva (NGS por sus siglas en inglés, Next Generation Sequencing), han hecho posible mejorar el diseño de los sgRNA. La Tabla 2 resume los principales programas bioinformáticos para el diseño de sgRNA que cuentan con genomas de hongos filamentosos en sus bases de datos.
Tabla 2. Principales programas bioinformáticos empleados en el diseño de sgRNAs para la edición de genomas de hongos filamentosos.
| Nombre | Sitio Web | Genomas disponibles | Lista de genomas (enlace) | Referencia | |
| N° de especies | Hongos filamentosos | ||||
| CRISPR direct | http://crispr.dbcls.jp/ | >200 | Si | https://gist.github.com/meso-cacase/88157c741a17c0920c7c57dcd2b39a3d
https://gist.github.com/meso-cacase/ef314e1e2ac4399fe20cd50fec77ce24 |
Naito et al., 2015 |
| EuPaGDT | http://grna.ctegd.uga.edu/ | >250 | Si | En el mismo sitio web | Peng y Tarleton, 2015 |
| Benchling | https://www.benchling.com/crispr/ | >1000 | Si | http://fungi.ensembl.org/index.html | Benchling, Inc. |
| Breakig-Cas | http://bioinfogp.cnb.csic.es/tools/breakingcas | >1000 | Si | En el mismo sitio Web | Oliveros et al., 2016 |
| Cas-Designer | http://www.rgenome.net/cas-designer/ | >200 | Si | En el mismo sitio Web | Park et al., 2015 |
| E-CRISPR | http://www.e-crisp.org/E-CRISP/ | 55 | Si | En el mismo sitio Web | Heigwer et al., 2014 |
| CCtop | https://cctop.cos.uni-heidelberg.de | 50 | Si | En el mismo sitio Web | Stemmer et al., 2015 |
| CRISPOR | http://crispor.tefor.net | 430 | Si | En el mismo sitio Web | Haeussler et al., 2016 |
| CHOPCHOP | http://chopchop.cbu.uib.no | 91 | Si | En el mismo sitio Web | Montague et al., 2014 |
CRISPR/Cas9 EN LA EDICIÓN DE GENOMAS DE HONGOS FILAMENTOSOS: ÚLTIMAS TENDENCIAS
La herramienta CRISPR/Cas para la edición de genomas ha revolucionado los laboratorios de todo el mundo, siendo el sistema tipo II de CRISPR/spCas9 de S. pyogenes el que ha demostrado el mayor rendimiento en genómica funcional en hongos filamentosos (Jinek et al., 2012; Komor et al., 2017), mostrando una alta precisión y simplicidad. Para el diseño y desarrollo de una nueva estrategia de edición de genomas de hongos filamentosos basados en el sistema CRISPR/Cas9, es necesario considerar cuatro aspectos medulares: a) estrategias para establecer el sistema CRISPR/Cas9 en células fúngicas, b) marcadores de selección positiva de transformantes (casetes de selección), c) mecanismos de expresión del gen CAS9 y, d) mecanismos de expresión de sgRNA.
Estrategias para implementar el sistema CRISPR/Cas9 en células fúngicas
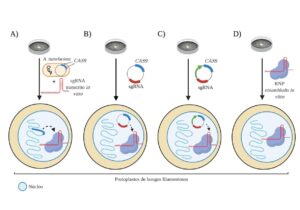
Para poder realizar la edición del genoma mediada por el sistema CRISPR/Cas9, tanto la endonucleasa como el sgRNA, deben estar presentes en el núcleo del organismo objetivo. En el caso de los hongos filamentosos, los métodos más comunes de transformación génica incluyen la transformación mediada por polietilenglicol (PEG), la transformación mediada por agrobacterias (AMT, por sus siglas en inglés Agrobacterium-mediated transformation) (Song et al., 2019) y la transformación mediada por electroporación (Schuster et al., 2019; Song et al., 2019). Los componentes del sistema CRISPR/Cas9 ingresan a las células fúngicas mediante vectores que portan casetes de expresión de CAS9 y sgRNA. Por lo general, estos casetes pueden ser transportados por un solo vector que transporte ambos casetes o por dos vectores que transportan un casete cada uno. Sin embargo, Zhang y colaboradores (2016) mostraron en Aspergillus fumigatus que la precisión y eficiencia del uso de un sistema de expresión de vector único es significativamente mayor que el sistema de expresión de vector doble. (Liu et al., 2015; Chen et al., 2017; Kuivanen et al., 2016; Kuivanen et al., 2017). Hasta la fecha, se han descrito principalmente cuatro estrategias para establecer el sistema CRISPR/Cas9 en hongos filamentosos (Figura 2): la integración de CAS9 en el genoma fúngico, la expresión transitoria (TE, por sus siglas en inglés, Transient Expression) de CAS9, la utilización de plásmidos de replicación autónoma (no integrativos) y el uso de ribonucleoproteínas (RNPs).
– Integración de CAS9 y sgRNA en sistemas de expresión transitoria (TE)
T. reesei fue el hongo filamentoso en el que se implementó el sistema CRISPR/Cas9 por primera vez, mediante AMT y utilizando un gen CAS9 con codones optimizado. La integración exitosa del gen CAS9 generó una cepa que requería únicamente el sgRNA para la edición del genoma, proporcionando un método dirigible rápidamente a múltiples genes simultáneamente (Liu et al., 2015), como se detalla en la Figura 2A. Sin embargo, la integración aleatoria del gen CAS9 en el genoma del huésped puede inducir mutaciones no deseadas.
Obtener una cepa fúngica que exprese Cas9 puede requerir, además de la detección de varios transformantes, la optimización del uso de codones del gen CAS9, el cual será diferente para cada especie hospedadora estudiada. Para poder superar estas limitaciones, se han utilizado sistemas TE de CAS9 y sgRNA en hongos filamentosos (Figura 2B). Matsu-ura y colaboradores (2015) desarrollaron un sistema CRISPR/Cas9 de expresión transitoria en el hongo filamentoso modelo Neurospora crassa, en el cual no existió ningún problema en la expresión de la endonucleasa y del sgRNA, lo que sugiere que la estrategia es eficiente y podría aplicarse a otros hongos filamentosos. Posteriormente, se realizaron trabajos con hongos que tenían múltiples núcleos, como el hongo filamentoso fitopatógeno Sclerotinia sclerotiorum. CAS9 y sgRNA pudieron expresarse de forma transitoria en los protoplastos y funcionar correctamente. Sin embargo, fragmentos de plásmidos de varios tamaños se integraron en el sitio de escisión de Cas9/sgRNA por la vía de reparación NHEJ generando mutantes con inserciones de plásmidos (Li et al., 2018).
– Plásmidos de replicación autónoma
Debido a que los hongos filamentosos no pueden mantener estables la mayoría de los plásmidos, varios replicadores cromosómicos como AMA1 y MATE han proporcionado un posible mecanismo para el mantenimiento extracromosómico de plásmidos en hongos filamentosos, como se muestra en la Figura 2C. Los plásmidos portadores del fragmento AMA1 poseen algunas ventajas, que incluyen el mejoramiento significativo en la transformación y la baja probabilidad de ser insertados en el genoma fúngico durante la transformación. Uno de los primeros estudios en utilizar este método fue descrito por Nodvig y colaboradores (2015), quienes silenciaron el gen yA en A. nidulans, provocando un cambio de color en los conidios de amarillo a verde.
Estrategias de selección de transformantes positivos: marcadores más empleados
Cuando se desea modificar el genoma de un organismo, en general, es determinante diseñar una estrategia de detección que permita confirmar dicha transformación. Es decir, desarrollar el mecanismo adecuado para la selección eficiente y eficaz de los transformantes positivos. Como indica su nombre, los marcadores de selección permiten distinguir aquellos organismos sobre los cuales la transformación de su genoma fue ejecutada correctamente. En hongos filamentosos, existe una extensa variedad de marcadores funcionales de selección, los cuales se pueden clasificar en dos grandes grupos: marcadores de selección dominante y marcadores de auxotrofía (i.e. la auxotrofía de algún aminoácido, restauración de pigmentación, habilidad de emplear alguna fuente de nitrógeno, etc.). Por ejemplo, entre los marcadores de selección más comunes que confieren resistencia a antibióticos destacan: hph, bar, nptII, ip, nat y ble, que confieren resistencia a la higromicina B, bialaphos, G418 (geneticina), carboxina, nourseothricina y bleomicina, respectivamente. Entre los marcadores de selección más empleados que restauran auxotrofías predominan: pyrG/pyr4, argB, y amdS, los cuales restauran la auxotrofía a uracilo, arginina o confieren la capacidad de utilizar acetamida como única fuente de nitrógeno, respectivamente.
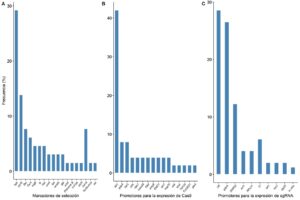
Los resultados obtenidos en la presente revisión, muestran que el marcador de selección más empleado en los sistemas CRISPR/Cas9 en hongos filamentosos es el hph con un 29.7% (Figura 3A), el cual confiere resistencia a la higromicina (Nødvig et al., 2015; Li et al., 2018; Darma et al., 2019). Sin embargo, los genes de resistencia a antibióticos no son recomendados en construcciones de cepas industriales, debido a que, en la mayoría de los países, las autoridades reguladoras se oponen al uso de los mismos por el riesgo potencial que estos representan a la salud humana. Además, sostienen que la propagación de cepas fúngicas que portan casetes de resistencia a antibióticos podría llegar a generar resistencia a gran escala en la sociedad humana. Es por ello que, convenientemente, se emplean marcadores de selección que restauren algún fenotipo determinado mediante la inserción estratégica en la cepa parental por procesos de mutación al azar o de ingeniería genética.
El segundo marcador de selección más empleado que se ha identificado es el gen pyrG con un 14.1% (Figura 3A). Dicho gen codifica la orotidina-5′-monofosfato decarboxilasa, una enzima implicada en la biosíntesis del aminoácido uracilo. Cuando se aplica el sistema CRISPR/Cas9 para la edición del genoma en una cepa de hongo filamentoso cuyo gen pyrG está inactivo (que confiere auxotrofía de uracilo), el gen pyrG es usado como marcador homólogo de selección. Cuando se introduce el DSB por la ribonucleasa Cas9 y se suministra una copia del gen pyrG (flanqueado con los brazos homólogos a la región donde ocurrió la incisión), se activa el mecanismo de reparación HDR. Así, el hongo que haya incorporado la copia funcional del gen pyrG será capaz de crecer en un medio de cultivo mínimo que no haya sido suplementado con uridina, pues ya habrá complementado la auxotrofía y volverá a ser protótrofo (Matsuda et al., 2018; Katayama et al., 2019; Kuivanen et al., 2019). Este es el principio fundamental en el que se basan todos los marcadores de selección destinados a restaurar una auxotrofía determinada.
Estrategias para la expresión del gen CAS9 en hongos filamentosos: promotores más empleados
La aplicación eficiente del sistema CRISPR/Cas9 requiere de la expresión heteróloga del gen CAS9 fusionado a una secuencia NLS (por sus siglas del inglés, Nuclear Localization Signal), así como también de la expresión simultánea del sgRNA. Tomando en cuenta estas dos consideraciones, se han establecido dos estrategias principales para la expresión del gen CAS9: in vitro e in vivo (Shi et al., 2017). En el primer caso, CAS9 y sgRNA son transcritos simultáneamente in vitro y luego forman el complejo de RNP Cas9/sgRNA para editar los genes diana. Dicha estrategia fue inicialmente evaluada y reportada por Pohl y colaboradores (2016) en Penicillium chrysogenum. En cambio, cuando se desea expresar el gen CAS9 in vivo, con frecuencia se emplea el gen CAS9 de S. pyogenes con el uso de codones optimizados para humanos (hSpCas9) fusionado a una NLS del virus 40 vacuolado del simio (SV40 NLS: 5’… CCCAAGAAGAAGAGGAAGGTG…3’) (Shi et al., 2017). Sin embargo, al ser una construcción general y relativamente simple, esta estrategia no es adecuada para todos los organismos de estudio. Es por ello que, con el fin de mejorar y asegurar la precisión y la eficiencia de edición de genomas, algunos investigadores han preferido optimizar las secuencias de CAS9 y NLS en función del uso de codones específico de los hongos filamentosos sobre los cuales se desea realizar la edición genómica (Tabla 3). En otros casos, los investigadores han escogido seleccionar directamente un NLS más fuerte para dirigir de manera eficiente la proteína de fusión al núcleo de la célula (Matsu-Ura et al., 2015; Fuller et al., 2015; Shi et al., 2017).
Además, la exitosa expresión heteróloga del gen CAS9 depende del tipo de promotor utilizado. La activación con la que los promotores dirigen la transcripción es un factor importante que afecta a la eficiencia de expresión de los genes exógenos, como es, en este caso, la expresión del gen CAS9. Por esta razón, la selección de un promotor adecuado es de gran importancia para un funcionamiento eficiente del sistema CRISPR/Cas9 (Song et al., 2019). Una amplia gama de promotores ha sido evaluada para la expresión de CAS9 en hongos filamentosos. De estos, los promotores constitutivos son usados con mayor frecuencia.
En este trabajo, los resultados muestran (Figura 3B) que entre los promotores más empleados en los sistemas de expresión heteróloga destacan: el promotor del gen tef1 que codifica el factor 1α de elongación de la traducción de A. nidulans (39.1%), el promotor del gen gpdA que codifica la gliceraldehído-3-fosfato deshidrogenasa de A. nidulans (8.3%) y el promotor del gen trpC de la biosíntesis de triptófano de A. nidulans (8.3%) (Arazoe et al., 2015; Matsu-Ura, 2015 et al.; Zhang et al., 2016). No obstante, esta última estrategia no permite controlar la estabilidad del sistema CRISPR/Cas9 en hongos filamentosos y, por tanto, existe la posibilidad de obtener off-targets en loci distintos del genoma al que se desea editar (Shi et al., 2017).
Para tener un mayor control sobre el sistema CRISPR/Cas9, se han empleado algunos promotores inducibles en hongos filamentosos, tales como: el promotor del gen amyB que codifica la TAKA-amilasa A de A. oryzae, el cual es inducido por almidón o maltosa y reprimido por glucosa; el promotor del gen cbh1 que codifica una celobiohidrolasa I de T. reesei, el cual es inducido por una serie de oligosacáridos o celulosa y reprimido por glucosa; el promotor del gen glaA, que codifica una glucoamilasa de A. niger que es inducido por glucosa y reprimido por xilosa; el promotor del gen niiA que codifica la nitrito reductasa de A. nidulans, el cual es inducido por nitrato y reprimido por amonio y el promotor del gen xlnA, que codifica una endoxilanasa de A. nidulans, el cual es inducido por xilosa o xilano y reprimido por glucosa (Liu et al., 2015a; Zhang et al., 2016; Katayama et al., 2016; Pohl et al., 2016; Zheng et al., 2018), entre otros. Así mismo, L. Naranjo (comunicación personal), propone el empleo del promotor inducible del gen oat1 que codifica una ornitina w-aminotransferasa en P. chrysogenum Wisconsin 54-1255, el cual se induce fuertemente por L-lisina, L-ornitina y L-arginina y se reprime por amonio (Naranjo et al., 2004). Las ventajas comparativas de este tipo de promotores inducibles es que permiten controlar la expresión del gen CAS9 con el fin de minimizar el número de off-targets y el consecuente mosaicismo en las cepas transformadas.
Estrategias para la expresión del sgRNA en hongos filamentosos: promotores más empleados
La expresión del sgRNA se puede realizar por dos vías: in vivo o in vitro. Para la expresión in vivo, se han desarrollado dos estrategias principales que se basan en el empleo de los promotores de la RNA polimerasa II o la RNA polimerasa III. Por ejemplo, U6 es un tipo de promotor endógeno para la RNA polimerasa III (Pol III) y genes de snRNA (por sus siglas del inglés small nuclear RNA) (Ryan y Cate, 2014). Para poder identificarlos y luego emplearlos en hongos filamentosos, es necesario comparar las secuencias snRNA entre distintas especies, cuyos promotores U6 ya han sido identificados previamente, con el fin de encontrar aquellas con al menos un 65% de identidad y, posteriormente, evaluar si efectivamente son capaces de transcribir de manera eficiente el sgRNA (Katayama et al., 2016; Schuster et al., 2016; Zhang et al., 2016). Adicionalmente, también se ha empleado el promotor del gen SNR52 que, al igual que U6, también es un promotor endógeno de la Pol III (Fuller et al., 2015; Matsu-Ura et al., 2015; Min et al., 2016; Shapiro et al., 2018; Vyas et al., 2018).
Sin embargo, esta estrategia de expresión de sgRNA puede resultar compleja porque algunos promotores endógenos de RNA polimerasa II (Pol II) de hongos filamentosos son actualmente difíciles de identificar en el genoma o no son adecuados para la transcripción de sgRNAs. En otros casos, los promotores U6 no son conservados en el genoma del organismo de interés, como es el caso de Ustilaginoidea virens. En dicho hongo, como sustituto del promotor U6 (ausente en el genoma), se utilizó la secuencia promotora del gen Gln-tRNA como promotor de la Pol III (Liang et al., 2018). Es por ello, que se desarrolló otra estrategia basada en promotores de la Pol II. En este sentido, Nødvig y colaboradores (2015) emplearon dos secuencias de ribozimas: 5′-end hammerhead (HH) y 3′-end virus hepatitis delta (HDV), con la finalidad de flanquear el sgRNA y permitir su transcripción in vivo. Finalmente, para asegurar una robusta transcripción de la secuencia que contiene el sgRNA, este grupo de investigación utilizó el promotor fuerte del gen constitutivo gpdA en A. nidulans (PgdpA). Esta importante investigación obtuvo como producto final la construcción de cuatro plásmidos CRISPR/Cas9 funcionales para la edición de genomas de hongos filamentosos: pFC330, pFC331, pFC332, pFC333 (disponibles en el repositorio de plásmidos de Addgene), los cuales poseen distintos marcadores de selección: pyrG, argB, hbh y ble, respectivamente. Estos plásmidos, que usan PgdpA como promotor de la Pol II, han sido usados ampliamente en numerosas investigaciones. Cabe destacar que, adicionalmente al PgdpA, también se ha empleado el promotor del gen utp25, el cual codifica una proteína U3 asociada a snRNA y es reconocido como un promotor de la Pol II (Van den Berg et al., 2008; Pohl et al., 2016). Así, los tres promotores para la expresión in vivo del sgRNA más empleados que hemos identificado en la presente investigación son U6 (34.1%), PgdpA (31.7%) y SNR52 (14.6%). Estos resultados sobre los promotores más empleados para la expresión de sgRNAs en hongos filamentosos se muestran en la Figura 3C. Además de las estrategias de expresión del sgRNA in vivo mencionadas anteriormente, también se han empleado estrategias de transcripción in vitro en las que se emplean específicamente los promotores U6 o T7 (Tabla 3).
Tabla 3. Estrategias para la expresión del gen CAS9, sgRNA y marcadores de selección para la edición de genomas de hongos filamentosos.
| Especie | Expresión de sgRNA | Expresión de Cas9 | Marcador de selección | Tipo de replicación | Método de transformación | Método de edición | Eficiencia | Referencia |
| Trichoderma reesei | Transcrito in vitro | pdc, cbh1 (codón optimizado) | ura5 | TEg | PMTf | NHEJe/HDRd | 93%/4.2-45% | Liu et al., 2015 |
| Pyricularia oryzae | U6, trpC | tef1 (codón optimizado) | bar | TE | PMT | NHEJ/HDR | 36.1-80.5 | Arazoe et al., 2015 |
| Aspergillus spp. | gdpA | tef1 (pFC330, pFC331, pFC332, pFC333) | pyrG, argB, hph, ble | AMA1a | PMT | NHEJ | – | Nødvig et al., 2015 |
| Neurospora crassa | SNR52 | trpC (codón optimizado) | bar | TE | PMT | NHEJ/HDR | – | Matsu-Ura et al., 2015 |
| Aspergillus fumigatus | SNR52 | tef1 (hSpCas9) | hph, ble | TE | PMT | NHEJ | 25%-53% | Fuller et al., 2015 |
| Phytophthora sojae | RPL41 | Ham34 (hSpCas9) | nptII | TE | PMT | NHEJ/HDR | – | Fang y Tyler 2016 |
| Ustilago maydis | U6 | Otef (tef1 modificado), hsp70 (codón optimizado) | ip | ARSc | PMT | NHEJ | 50%-90% | Schuster et al., 2016 |
| Aspergillus fumigatus | U6 | niiA, gpdA (hSpCas9) | pyr4, hph | AMA1 | PMT | HDR | 95%-100% | Zhang et al., 2016 |
| Aspergillus oryzae | U6 | amyB (codón optimizado) | pyrG | TE | PMT | NHEJ | 10%-20% | Katayama et al., 2016 |
| Penicillium chrysogenum | U6, utp25 | xlnA (hSpCas9) | amdS | AMA1/TE | PMT | NHEJ/HDR | 100% | Pohl et al., 2016 |
| Candida albicans | SNR52 | ENO1 (codón optimizado) | Nat | TE | Lithium acetate | NHEJ/HDR | – | Min et al., 2016 |
| Aspergillus niger | gdpA | tef1 (pFC332) | pyrG, hph | AMA1 | PMT | NHEJ/HDR | 37-5%-100% | Kuivanen et al., 2016 |
| Aspergillus fumigatus | gdpA | TetON (pFC332) | pyrG | AMA1 | PMT | NHEJ/HDR | – | Weber et al., 2016 |
| Leptosphaeria maculans | act1 | act1 (codón optimizado) | ip, nptII, hph | – | AMTb | NHEJ | – | Idnurm et al., 2017 |
| Coprinopsis cinerea | U6 | CcDED1 (codón optimizado para basidiomycete) | gfp | – | Electroporación | NHEJ | 10.5 | Sugano et al., 2017 |
| Myceliophthora thermophila | U6 | tef1 (codón optimizado) | bar | TE | PMT/AMT | NHEJ/HDR | 15%-95% | Liu et al., 2017 |
| Myceliophthora heterothallica | ||||||||
| Talaromyces atroroseus | gdpA | tef1 (pFC330) | hph | AMA1 | PMT | NHEI | – | Nielsen et al., 2017 |
| Aspergillus carbonarius | gdpA | tef1 (pFC332) | hph | AMA1 | AMT | NHEJ/HDR | 27% | Weyda et al., 2017 |
| Aspergillus niger | gdpA | tef1 (pFC332)
|
hph | AMA1 | PMT | NHEJ | – | Kuivanen et al., 2017 |
| Beauveria bassiana | Transcrito in vitro | gdpA (codón optimizado) | gfp, ura5, bar | – | PMT | NHEJ/HDR | 5%-50% | Chen et al., 2017b |
| Alternaria alternata | gdpA | gdpA (pFC332) | pyr4, hph | AMA1 | PMT | NHEJ | – | Wenderoth et al., 2017 |
| Shiraia bambusicola | U6 | trpC (codón optimizado) | hph | – | PMT | NHEJ | – | Deng et al., 2017b |
| Shiraia bambusicola | U6 | trpC (codón optimizado) | hph | – | PMT | NHEJ/HDR | 32% | Deng et al., 2017c |
| Nodulisporium sp. | U6, T7 | trpC (codón optimizado) | bar | TE | PMT | NHEJ/HDR | – | Zheng et al., 2017 |
| Mucor circinelloides | Transcrito in vitro | SpCas9 | pyr4 | TE | PMT | NHEJ/HDR | 100% | Nagy et al., 2017 |
| Ustilago maydis | U6 | Otef (tef1 modificado), hsp70 (codón optimizado) | ip | ARS | PMT | NHEJ | 50%-100% | Schuster et al., 2018 |
| Fusarium graminearum | tef1 | gdpA (codón optimizado) | fludioxonil | – | PMT | NHEJ/HDR | 1%-10% | Gardiner y Kazan, 2018 |
| Aspergillus niger | gdpA | tef1 (pFC332) | hph | AMA1 | PMT | NHEJ | – | Kuivanen y Richard, 2018 |
| Candida albicans | SNR52 | SpCas9 | Nat, Phloxine B | – | Electroporación | NHEJ | 53%-98% | Shapiro et al., 2018 |
| Aspergilli | gdpA | tef1 (pFC332)
|
arg, pyrG | AMA1 | AMT | HDR | 15%-90% | Nødvig et al., 2018 |
| B. dermatitidis | gdpA | tef1 (pFC332) | hph | AMA1 | AMT | NHEJ | 22%-73% | Kujoth et al., 2018 |
| Aspergillus niger | U6 | glaA (pFC332, codón optimizado) | hph, amdS | AMA1 | PMT | NHEL/HDR | 33.3%-100% | Zheng et al., 2018 |
| Candida albicans | SNR52 | ENO1 (codón optimizado) | Nat | ARS | Electroporación | HDR | 25%-100% | Vyas et al., 2018 |
| Phytophthora sojae | RPL41 | Ham34 (hSpCas9) | nptII | TE | PMT | NHEJ | Miao et al., 2018 | |
| Fusarium oxysporum | Transcrito in vitro | pFC332 | hph | AMA1 | PMT | NHEJ/HDR | 20%-53.8% | Wang et al., 2018a |
| Ustilaginoidea virens | Gln-tRNA | pdc, cbh1 (codón optimizado) | nptii | – | AMT PMT | NHEJ | – | Liang et al., 2018 |
| Cordyceps militaris | T7 | tef1 (codón optimizado) | 5-FOA, blcR | – | AMT/PMT | NHEJ/HDR | – | Chen et al., 2018 |
| Cryptococcus neoformans | U6 | tef1 (codón optimizado) | Ntc | – | Electroporación | HDR | 96.5%-100% | Wang et al., 2018b |
| Sclerotinia sclerotiorum | T7 | tef1 (hSpCas9) | hph | – | PMT | NHEJ/HDR | 38%-100% | Li et al., 2018 |
| Aspergillus novofumigatus | gdpA | tef1 (pFC332) | pyrG | AMA1 | PMT | NHEJ | – | Matsuda et al., 2018 |
| Aspergillus niger | gdpA | tef1 (pFC332) | argB, pyrG | AMA1 | PMT | HDR | 100% | Leynaud-Kieffer et al., 2019 |
| Aspergillus oryzae | U6 | amyB (codón optimizado) | pyrG | – | PMT | HDR | 50%-100% | Katayama et al., 2019 |
| Alternaria alternata | gdpA | tef1 (pFC332) | hph | AMA1 | PMT | NHEJ | Igbalajobi et al., 2019 | |
| Aspergillus niger | Transcrito in vitro | codón optimizado | pyrG | – | PMT | NHEJ/HDR | 100% | Kuivanen et al., 2019 |
| Mucor circinelloides | Transcrito in vitro | SpCas9 | pyr4 | – | PMT | HDR | 100% | Nagy et al., 2019 |
| Leptosphaeria maculans | act1 | act1 (hSpCas9) | hph | – | AMT | NHEJ | – | Darma et al., 2019 |
| Ashbya gossypii | SNR52 | tef1 (hSpCas9) | nptII | – | Electroporación | NHEJ | 44%-85% | Jiménez et al., 2019 |
| Duddingtonia flagrans | Transcrito in vitro | tef1 (pFC332) | hph | AMA1 | PMT | HDR | – | Youssar et al., 2019 |
a: Apical Membrane Antigen 1; b: Agrobacterium-mediated transformation; c: Autonomously Replicating Sequence; d: Homology-Directed Repair; e: Non-homologous end joining; f: Protoplast-mediated transformation; g: Transient Expression.
Los autores Liu y colaboradores (2015a) realizaron el primer sistema CRISPR/Cas9 para edición del genoma de T. Reesei. En este sistema, se empleó el promotor T7 para la expresión in vitro del sgRNA y los promotores cbh1 o pdc, Pcgh1 y Ppdc. Además, para la expresión heteróloga de CAS9, los autores optimizaron el uso de codones para T. reesei y, como marcador de selección, emplearon el gen hph que confiere resistencia a la higromicina. La transformación se realizó en un plásmido TE alcanzando una eficiencia de edición entre 93 y 100%.
En otro ejemplo, Pohl y colaboradores (2016) desarrollaron un repertorio de herramientas moleculares para la edición del genoma en P. chrysogenum tanto in vivo como in vitro. Mientras que el gen hcas9 funcional en humanos fue expresado in vivo bajo el control del promotor inducible del gen xlnA, el sgRNA fue expresado in vivo e in vitro bajo el control del promotor T7. Como marcador positivo de selección de transformantes, los autores emplearon el gen amdS que confiere la capacidad de utilizar acetamida como única fuente de carbono y energía (Kelly y Hynes, 1985). Los plásmidos de transformación evaluados por los autores fueron de replicación autónoma (AMA1) y de expresión transitoria. Los resultados obtenidos con el sistema propuesto mostraron una eficiencia de modificación del genoma entre 33-100%.
Alternativamente, Zhang y colaboradores (2016) propusieron el uso de un sistema CRISPR/Cas9 que expresaba sgRNA in vitro y bajo el control del promotor U6 en la especie A. fumigatus. En este caso, se empleó un gen CAS9 con uso de codones optimizado para humanos expresada bajo el control del promotor Ptef1. Como marcadores de selección, se utilizaron el gen pyr4 (restauración de la auxotrofía de uracilo) y el gen hph que confiere resistencia a la higromicina en plásmidos de replicación autónoma, alcanzando un porcentaje de modificación de entre 95 y 100%. En otro caso, con A. fumigatus, Weber y colaboradores (2016) expresaron el gen CAS9 bajo el promotor inducible TetON y se empleó la auxotrofía de uridina/uracilo (pyrG–) como marcador de selección. Los plásmidos de transformación contenían la región AMA1 para una replicación autónoma, logrando eficiencias de edición de hasta un 80%.
La Figura 3 muestra los marcadores de selección positiva de transformantes y los promotores más empleados en los sistemas CRISPR/Cas9 en hongos filamentosos que se han descrito en la literatura científica indexada entre los años 2015-2020.
Complejos de ribonucleoproteínas (RNPs)
Para la optimización de todos los sistemas CRISPR/Cas9 en hongos filamentosos se requiere una cantidad considerable de tiempo y esfuerzo. En consecuencia, los intentos más recientes han utilizado un método de entrega basado en un complejo de ribonucleoproteína (RNP) Cas9:sgRNAs para la edición del genoma en diferentes especies, tales como Arabidopsis, la mosca de la fruta, el pez cebra y en líneas celulares humanas. Las RNPs consisten en una proteína Cas9 purificada a homogeneidad y un sgRNA transcrito sintetizado in vitro, formando un complejo que posteriormente se transfecta directamente en las células huésped (Figura 2D).
En comparación con el uso de plásmidos, los complejos RNPs Cas9:sgRNAs presentan varias ventajas. La expresión del gen CAS9 y la transcripción del sgRNA no dependen de la maquinaria del huésped, los RNPs ensamblados tienen actividad de escisión inmediata y la eficiencia de escisión de los diferentes sgRNAs puede probarse in vitro permitiendo elegir los sgRNAs más adecuados. Las RNPs se agotan en un periodo corto de tiempo de las células huésped mediante la vía de degradación de proteínas, reduciendo de esta manera la posibilidad de actividad fuera del objetivo de Cas9 (Wang et al., 2019).
El método CRISPR/Cas9 basado en RNPs Cas9:sgRNAs se ha utilizado en varias especies de hongos filamentosos. Un estudio reciente utilizó dos cepas de A. fumigatus (WT y ΔakuB) para emplear el mecanismo de reparación HDR mediado por el sistema CRISPR/Cas9 con el objetivo de alterar el gen pksP, responsable del color verde de los conidios. Como resultado, ambas cepas mostraron el fenotipo deseado, obteniendo un 97% de eficiencia en la cepa ΔakuB, mientras que en la cepa WT se estuvo una eficiencia entre 40 y 74% (Al Abdallah et al., 2017). En otro estudio, se desarrolló un método de deleción genética en el hongo fitopatógeno Fusarium proliferatum (Ferrara et al., 2019). En este sistema se realizó una ruptura de doble cadena de ADN mediada por dos RNPs con recombinación de microhomología. Este sistema redujo el riesgo de mutaciones fuera del objetivo y minimizó el riesgo de alterar cualquier gen adyacente al gen objetivo. En este estudio, se eliminó el gen FUM1 requerido para la biosíntesis de la micotoxina fumonisina. Tras la cuantificación de fumonisina en los transformantes positivos, se determinó que ninguna de las cepas mutantes presentó cantidades detectables de esta micotoxina (Ferrara et al., 2019).
Se han descrito varios métodos basados en complejos de RNPs Cas9:sgRNA en otras especies de hongos filamentosos como P. chrysogenum, Mucor circinelloides, Magnaporthe oryzae y Fusarium oxysporum (Pohl et al., 2018; Nagy et al., 2017; Foster et al., 2018; Wang et al., 2018). En conjunto, estos estudios indican que el establecimiento del sistema CRISPR/Cas9 mediado por RNPs tiene cada vez más aplicaciones en la edición de genomas de hongos filamentosos por su alta eficiencia y versatilidad.
CRISPRi y CRISPRa EN HONGOS FILAMENTOSOS
La flexibilidad del sistema CRISPR/Cas9 para la manipulación del genoma ha permitido el desarrollo de nuevas alternativas para la interferencia (CRISPRi) y activación (CRISPRa) de genes. Estos nuevos sistemas pueden regular la expresión transcripcional y están basados en una endonucleasa Cas9 mutada (dCas9, por sus siglas en inglés nuclease-dead Cas9) que ha perdido su actividad mediante la inactivación de sus dominios RuvC y HNH. La dCas9 está dirigida a regiones promotoras específicas a través del sgRNA, para lograr el impedimento estérico de la ARN polimerasa bloqueando de esta manera el inicio o alargamiento de la transcripción (Wang et al., 2019). Además, dCas9 puede fusionarse con represores o activadores transcripcionales para disminuir o aumentar la expresión génica, respectivamente.
En el caso de CRISPRa, Roux y colaboradores (2020) utilizaron un sistema CRISPR/dLbCas12a-VPR para la activación de un grupo de genes biosintéticos fúngicos (BGC) en la especie Aspergillus nidulans, tales como el gen nativo micA que codifica una enzima tipo péptido no ribosómico, responsable de la biosíntesis de la microperfuranona. Los autores utilizaron sistemas CRISPRs direccionados a dos loci. Mediante análisis cuantitativos, los autores determinaron que la implementación de estos sistemas condujo a un incremento significativo en la biosíntesis de microperfuranona en A. nidulans, respecto a los controles. En el hongo levaduriforme Candida albicans se han aplicado sistemas represores y activadores transcripcionales basados en CRISPR, fusionando la enzima dCas9 a dominios de activación transcripcionales Gal4 y/o VP64, y con el represor Nrg1, demostrando la regulación específica del gen codificante de catalasa citosólica CAT1 (Román et al., 2019).
En C. albicans también se ha empleado CRISPRi a través de un sistema optimizado CRISPRi dCas9-Mxi1, con la finalidad de reprimir la expresión del gen HSP90 que codifica una chaperona molecular esencial en esta levadura. En la fase experimental del estudio, se emplearon dos sgRNA distintos para cada cepa, teniendo como resultado que ambas cepas positivas dCas9 mostraron fenotipos que corresponden a la disminución de HSP90 (Wensing et al., 2019). Estos estudios evidencian que los sistemas CRISPRi y CRISPRa presentan un alto potencial y una valiosa expansión en el creciente repertorio de herramientas moleculares CRISPR para hongos filamentosos.
CONCLUSIONES Y PERSPECTIVAS
A medida que transcurre el tiempo se consolida cada vez más la importancia industrial que tienen los hongos filamentosos como factorías celulares en la producción de una ilimitada variedad de productos de alto valor biotecnológico. Además de contribuir a la descarbonización neta de la economía, contribuir a la mitigación del cambio global en su bioproceso y a la economía circular, estos productos han mostrado ser económicamente rentables, socialmente justos y ecológicamente viables. Estos microorganismos cosmopolitas, que nos han acompañado desde tiempos ancestrales, poseen un alto valor estratégico en diversas áreas, tales como la salud humana y animal, la alimentación, el desarrollo de nuevos biomateriales y biomoléculas, la implementación de una agricultura bajo un enfoque agroecológico, la generación de nuevas fuentes de bioenergía y la biocatálisis ambiental, entre otros sectores. Todas estas áreas son ecológicamente trascendentales para preservar la vida en sobre el planeta Tierra.
El estudio y la aplicación del sistema CRISPR/Cas9 como herramienta de edición genómica de hongos filamentosos ha abierto una brecha sin retorno que brinda un sinfín de posibilidades, donde el límite está solamente en la imaginación del investigador. Debido a la flexibilidad del sistema CRISPR/Cas9, se han desarrollado diferentes alternativas, como los sistemas CRISPRi y CRISPRa, que presentan a futuro una prometedora estrategia de regulación transcripcional en hongos filamentosos. Además de la gran variedad de sistemas, la tecnología CRISPR/Cas puede ampliar sus fronteras moleculares implementando nucleasas diferentes a Cas9, como es el caso de las endonucleasas Cpf1 y nickasas.
En relación a las estrategias tecnológicas que han predominado para obtener una mayor frecuencia de modificación genética y como consecuencia una mayor probabilidad de modificación genómica, se han desarrollado las siguientes tendencias. Respecto al uso de marcadores positivos de selección de transformantes, se ha evidenciado la utilización con mayor frecuencia del gen hph (29.7%), el cual confiere resistencia a la higromicina, seguido por el grupo de genes pyrG/pyr4/ura5/ura3 que restaura la auxotrofía de uracilo (24.4%). En los mecanismos de expresión heteróloga del gen CAS9, existe una clara tendencia en la utilización del promotor Ptef1 (39.6%), aunque provoca una fuerte expresión aumentando de esta manera las posibilidades de producir off-targets. Es por ello que existe un gran interés en estudiar y aplicar promotores inducibles, tales como amyB, cbh1, niiA, glaA y xlnA, con el fin de controlar/modular la expresión del gen CAS9 y reducir la generación de off-targets y concomitante mosaicismo. Referente a los mecanismos de expresión del sgRNA, se observa una clara inclinación por la expresión in vivo con el uso del promotor U6 (34.1%), un tipo de promotor endógeno para la RNA polimerasa III. Para la expresión in vitro del sgRNA en hongos filamentosos, se ha optado por emplear vectores de expresión bajo el control de los promotores U6 o T7, a pesar de que esta estrategia ha sido muy poco utilizada hasta la fecha (2.4%).
La edición de genomas de hongos filamentosos empleando CRISPR/Cas9 ha tenido un crecimiento sostenido desde su origen, dícese en los últimos 5 años, pues cada vez se han obtenido mejores eficiencias de transformación empleando una variedad importante de promotores constitutivos e inducibles. Sin embargo, aún existen numerosos promotores inducibles que no han sido evaluados hasta la fecha y que podrían solucionar inconsistencias que han surgido en la edición de genomas de ciertos hongos filamentosos. Además, la lista de genomas secuenciados de estos microorganismos ha aumentado vertiginosamente, lo que abre oportunidades para nuevos desafíos orientados al diseño de nuevas herramientas moleculares basados en CRISPR/Cas9. Esto, a su vez, exige seguir perfeccionando las herramientas bioinformáticas y algoritmos disponibles para el diseño de los sgRNAs con el fin de disminuir los off-targets y las probabilidades de error en la edición de genomas de hongos filamentosos.
CONFLICTO DE INTERESES
Los autores del trabajo manifiestan ningún conflicto de interés relacionado.