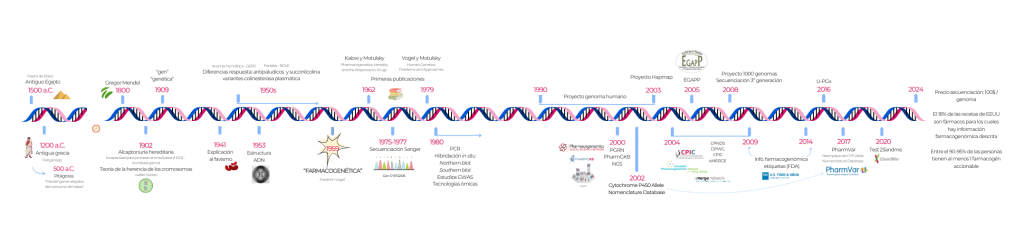
Desde que en los años 70 se presentó el primer método de secuenciación desarrollado por Sanger hemos asistido a una revolución de las tecnologías de secuenciación genética, incentivado por el proyecto genoma humano en el año 2000 que llevó a la secuenciación por vez primera del genoma completo (Roberts, 2001).
La farmacogenética y la farmacogenómica son áreas de conocimiento cuyo objetivo es la identificación de variantes genéticas o genómicas que proporcionarán la información necesaria para la definición de un tratamiento farmacológico individualizado basado en el perfil genético de cada paciente, contribuyendo así a la Medicina Personalizada de Precisión (Informes Anticipando, 2021).
Hoy en día sabemos que las diferentes respuestas que los pacientes experimentan ante los tratamientos se deben a polimorfismos genéticos heredados: pequeñas modificaciones en la secuencia genética de los individuos que pueden modificar la expresión y la función de las proteínas que codifican (Informes Anticipando, 2021). Sin embargo, la historia de la farmacogenética comienza mucho antes de que existieran términos como «genoma» o «ADN». Para comprender su desarrollo como área, debemos retroceder en el tiempo y explorar los acontecimientos que asentaron sus bases.
ANTES DE LA FARMACOGENÉTICA
El Papiro de Ebers, uno de los textos médicos más antiguos y extensos que se conocen, recoge, mezclando magia y sustancias curativas, la comprensión de la Medicina que tenían los antiguos egipcios hasta el año 1500 a.C. Poco después los antiguos griegos desarrollaron la teoría de la «pangénesis», que nace “de la observación de que tanto los rasgos normales como los patológicos se transmiten a la descendencia y deben hacerlo a través de un “portador de información”, definiendo la herencia” (Müller y Rizhanovsky, 2020). Así, se puede considerar que ya en esas civilizaciones se empezaban a delinear y definir los conceptos científicos de la herencia y el tratamiento médico que posteriormente la farmacogenética fusionaría.
Primera observación farmacogenética
Aunque el concepto de genética estaba aún lejos de nacer, la primera observación farmacogenética se le atribuye al científico griego Pitágoras (570-490 a.C.), quien aconsejó evitar el consumo de habas, usadas comúnmente para tratar el estreñimiento, al notar que causaban una reacción adversa en ciertas personas, pero no en otras. Este fenómeno se bautizó como favismo. Pitágoras advirtió también que esta enfermedad era más común en la región del Mediterráneo (Müller y Rizhanovsky, 2020; Bousman et al., 2023).
La genética mendeliana y los conceptos de “gen” y “genética”
No es hasta el desarrollo de la genética mendeliana clásica, a mediados del siglo XIX, cuando vuelve a haber hitos relevantes en el campo de la farmacogenómica, que había quedado un poco estancada.
Mendel publicó sus estudios en guisantes «Experiments in Plant Hybridization» en la revista Proceedings of the Brunn Natural History Society en 1866 ( Mendel, 1866). Sin embargo, no fue hasta principios del siglo siguiente (1900), momento en el que su trabajo se redescubierto y confirmado por tres científicos, Hugo DeVries, Carl Correns y Erich von Tschermak, de manera independiente, cuando su trabajo genera verdaderamente un impacto (Roberts, 1929). Poco después, en 1902, otro científico, William Bateson, uno de los primeros genetistas y defensores de las ideas de Mendel, publica Mendel’s Principles of Heredity: A Defence, with a Translation of Mendel’s Original Papers on Hybridisation, un libro en el que expone sus descubrimientos de que la primera ley de Mendel no es aplicable sólo a las plantas, sino también a los animales, además de introducir los conceptos de alelo, homocigoto y heterocigoto (Bateson y Mendel, 2009). Ese mismo año (1902) Walter Sutton enuncia la Teoría Cromosómica de la Herencia de los cromosomas, en la que se propone que los cromosomas son los portadores de los factores mendelianos (Sutton, 1903).
Esta comprensión más profunda de la herencia y la variación de los alelos hizo posible una predicción fiable de enfermedades hereditarias y trajo avances como como el descubrimiento de la alcaptonuria hereditaria en 1902 por Archibald Garrod. Esta condición surge debido a una mutación autosómica recesiva en la enzima homogentisato 1,2-dioxigenasa (HGD), que provoca la incapacidad de metabolizar dos aminoácidos (Garrod, 1908).
Siete años más tarde, en 1909, el botánico danés Wilhelm Johannsen acuñó la palabra gen para describir las unidades mendelianas de la herencia. La palabra deriva del griego genos, que significa «nacimiento». Cuatro años antes, el ya mencionado William Bateson, había ya utilizado la palabra genética en una carta, al sentir la necesidad de un término nuevo para describir el estudio de la herencia y las variaciones heredadas (National Human Genome Research Institute).
La explicación al favismo y otras observaciones farmacogenéticas
La explicación al favismo llegó en el siglo XX, tras conocerse la teoría “un gen, una enzima” (Beadle y Tatum, 1941). El consumo de habas provocaba en algunas personas una anemia hemolítica aguda, debido a la existencia de variantes genéticas en el gen que codifica la glucosa-6-fosfato deshidrogenasa (G6PD) (Müller y Rizhanovsky, 2020). Una deficiencia en esta misma enzima fue la responsable de que, durante la Segunda Guerra Mundial, los soldados de ascendencia africana, al tomar antipalúdicos como la primaquina, desarrollaran una serie de efectos secundarios adversos, incluyendo anemia hemolítica. Esta deficiencia era frecuente en los africanos porque, en heterocigosis, era una ventaja que protegía al portador de la malaria, pero era rara en países sin malaria (Kalow, 2005; Ampong, 2019).
Arno Motulsky describió esta observación en el artículo “Reacciones a Medicamentos, Enzimas y Genética Bioquímica”, publicado en JAMA en 1957 (Motulsky, 1957). Motulsky se ganó el título de padre de la farmacogenómica al impulsar a otros científicos a destacar el papel de la genética en el estudio de la variabilidad de respuesta a los medicamentos (Müller and Rizhanovsky, 2020).
En 1955 Ernst Beutler demostró lo que Motulsky ya sospechaba: que la anemia hemolítica era causada por una deficiencia en la enzima G6PD, misma enzima responsable de las diferencias de respuesta a algunos medicamentos como el antimalárico primaquina (Beutler, 1959). Por otro lado, Lehmann y Ryan observaron que el fármaco anestésico succinilcolina, un relajante muscular, prolongaba la parálisis e inducía reacciones fatales en algunos individuos con una variante genética mutada vinculada a la ausencia de la enzima butirilcolinesterasa (Lehmann y Ryan, 1956). Así, en la década de 1950, ya se habían observado y demostrado múltiples respuestas atípicas a medicamentos (Motulsky, 1957; Ampong, 2019).
FARMACOGENÉTICA: EL AÑO 0
En el año 1953, James Watson y Francis Crick, apoyándose en las contribuciones de Rosalind Franklin, publicaron la estructura del ADN (Watson y Crick, 1953), justo un año después de conocerse que los genes estaban formados de esta molécula (Hershey y Chase, 1952). Este importante hito, seguido rápidamente por el descubrimiento de la secuenciación de ADN por Berg, Gilbert y Sanger a principios de la década de 1970 (Gayon, 2016), tras el descubrimiento del código genético en 1966, impulsó mucho los estudios de genética en ese momento.
1959 – El año 0
El término ‘farmacogenética’ fue acuñado finalmente en 1959 por el científico Friedrich Vogel (Vogel, 1959). El estrecho contacto entre la genética y la farmacología comenzaba a hacerse cada vez más evidente. Los estudios que lo inspiraron a acuñar este término fueron los realizados por Harris y colaboradores sobre la variabilidad de la percepción del gusto de la feniltiourea y los trastornos tiroideos. También influyó que, durante su carrera, Vogel mostró especial interés por la porfiria hepática, una enfermedad autosómica dominante, relativamente rara en humanos, causada por una deficiencia en la enzima ALA sintasa, cuyos síntomas parecían verse empeorados por algunos medicamentos como los barbitúricos, sulfonal y glutetimida en algunos individuos, pero no causaba efectos tóxicos negativos en otros (Müller and Rizhanovsky, 2020).
Aportaciones a la consolidación del nuevo campo
Durante la postguerra, Werner Kalow tuvo un importante papel en la consolidación de la farmacogenética como un campo. Su entrada en este campo se da en 1947, cuando dos pacientes mueren en Berlín tras recibir dosis rutinarias de procaína (Novocaía), un anestésico tópico. Ante la sospecha de que la desnutrición endémica había dejado a los pacientes con una deficiencia de una enzima metabólica crucial, Kalow comenzó a investigar cómo las diferencias individuales en el metabolismo podían influir en las reacciones a los medicamentos. Así determinó que la afinidad de una enzima por un fármaco dependía de su estructura y que la estructura de la enzima, a su vez, estaba determinada por la secuencia de aminoácidos. Por lo tanto, las diferencias individuales en las reacciones a los medicamentos debían tener una base genética.
Kalow también publicó la primera obra dedicada en su totalidad al campo de la farmacogenética, un libro de 222 páginas que consiguió elevar la farmacogenética desde tan sólo una subespecialidad a un campo completo: la monografía Pharmacogenetics: Heredity and the Response to Drugs (1962), donde los lectores podían encontrar desde una introducción sobre la genética de bacterias, insectos y mamíferos, hasta discusiones sobre reacciones anormales a medicamentos causadas por variaciones metabólicas. Además, incluyó su propio trabajo sobre la colinesterasa sérica y exploró cuestiones curiosas como por qué los espárragos alteran el aroma de la orina en algunas personas, pero no en otras. (Kalow, 1962; Jones, 2013).
Kalow también planteó que la variación observada entre especies podría encontrarse también entre individuos de una misma especie y demostró por primera vez diferencias entre poblaciones (Jones, 2013; Müller and Rizhanovsky, 2020).
Descubrimiento del gen CYP2D6
El descubrimiento del gen CYP2D6 del citocromo P450 en 1977 a través del estudio del metabolismo de la debrisoquina, ayudó a explicar las diferencias entre individuos en la capacidad de hidroxilación y la respuesta al fármaco (Mahgoub et al., 1977). Esto se consiguió gracias a poder evaluar fenotípicamente la capacidad de las enzimas metabolizadoras de fármacos, mediante la administración de un fármaco de prueba y la medición de la proporción entre el fármaco y su metabolito (Gonzalez et al., 1988; Pirmohamed, 2011).
La enzima que codifica este gen es responsable del metabolismo primario de aproximadamente el 25% de los medicamentos de uso clínico, entre los que se encuentran analgésicos, antidepresivos, antihipertensivos y el agente anticancerígeno tamoxifeno (Taylor et al., 2020). Las diferencias en la función vienen respaldadas por las diferencias en la arquitectura genética, pues se han descrito más de 100 variantes alélicas para este gen. La identificación de esta familia de genes hizo posible llevar a cabo estudios poblacionales a gran escala y esclareció algunas de las variaciones interétnicas en las reacciones a los medicamentos (Müller and Rizhanovsky, 2020).
LA FARMACOGENÉTICA CONSTITUÍDA
Gracias al desarrollo de la técnica de reacción en cadena de la polimerasa (PCR) en 1985, la hibridación fluorescente in situ en 1980, y las técnicas de northern y southern blotting en la década de 1990, las nuevas técnicas de genética molecular estuvieron fácilmente disponibles para estudiar deleciones, duplicaciones y translocaciones en el ADN. Desde entonces el interés en el campo ha experimentado un aumento exponencial. A medida que avanzaron las tecnologías genómicas, como los estudios de asociación del genoma completo (GWAS), que permitían estudiar cómo combinaciones complejas de variantes genéticas en múltiples genes pueden influir en la respuesta a los tratamientos farmacológicos, comienza a usarse el término farmacogenómica de manera generalizada y sin atribuirse a ningún individuo en específico (Motulsky, 2002; Müller y Rizhanovsky, 2020).
Acciones tomadas para la instauración del campo
En octubre de 1990 se lanzó de manera oficial el Proyecto Genoma Humano, que terminó siendo completado en abril de 2003. Ese mismo año se lanzó el proyecto HapMap, diseñado para identificar y catalogar las variantes genéticas comunes en diferentes poblaciones humanas. Este proyecto facilitó la investigación sobre cómo estas variaciones afectan la salud, las enfermedades y las respuestas a los tratamientos farmacológicos (Ampong, 2019).
En el año 2000 se habían fundado ya la Pharmacogenomics Research Network (PGRN) y la base de datos PharmGKB, con el objetivo de facilitar el acceso y la organización de la información genética relevante en el campo de la farmacología (Pharmacogenomics Global Research Network). En 2002 se creó una base de datos centrada en las variantes alélicas del citocromo P450 (CYP), la Cytochrome P450 (CYP) Allele Nomenclature Database (Human Cytochrome P450 (CYP) Allele Nomenclature Committee). En Estados Unidos, las subvenciones de la PGRN financiadas por los Institutos Nacionales de Salud (NIH) a través del Instituto Nacional de Ciencias Médicas Generales (NIGMS), y las subvenciones eMERGE del Instituto Nacional de Investigación del Genoma Humano (NHGRI) impulsaron la investigación al proporcionar acceso a técnicas nuevas y muy costosas, como los GWAS y la secuenciación de nueva generación (NGS), que nace en 2005 (Margulies et al., 2005; Weinshilboum y Wang, 2017; ‘welcome to eMerge’, no date). Esto generó un rápido crecimiento del conocimiento farmacogenómico clínicamente relevante, teniendo información de medicamentos utilizados en prácticamente todas las especialidades médicas. Además, generó un fuerte estímulo para la implementación clínica de la farmacogenómica, que coincidió con la implementación de los registros médicos electrónicos, esenciales tanto para almacenar la creciente cantidad de datos genómicos como para proporcionar herramientas que permitan entregar esa información de manera inmediata a los médicos en el momento de la prescripción (Weinshilboum y Wang, 2017; eMerge).
Entre 2004 y 2009, surgieron consorcios importantes como la Canadian Pharmacogenomics Network for Drug Safety (CPNDS), el Dutch Pharmacogenetics Working Group (DPWG) y el Clinical Pharmacogenetics Implementation Consortium (CPIC), cuyo objetivo fue traducir los descubrimientos genéticos en guías clínicas prácticas para el uso de medicamentos.
En 2005 nació la Iniciativa para la Evaluation of Genomic Applications in Practice and Prevention (EGAPP), establecida por la Oficina Nacional de Genómica en la Salud Pública de los Centros para el Control y la Prevención de Enfermedades (CDC), con la función de respaldar el desarrollo e implementación de un proceso riguroso y basado en evidencia para evaluar pruebas genéticas y otras aplicaciones genómicas destinadas a la práctica clínica y de salud pública en los Estados Unidos (Teutsch et al., 2009).
Hacia 2008 comenzó el ambicioso Proyecto 1000 Genomas, cuyo objetivo era secuenciar los genomas de individuos de diferentes orígenes étnicos para ofrecer una visión más detallada de la diversidad genética y sus implicaciones clínicas (Ampong, 2019). En esta fecha comienza también a desarrollarse la secuenciación de tercera generación: secuenciación de lecturas largas con la promesa de superar las limitaciones de la NGS en lo que a la corta longitud de las lecturas respecta (Scarano et al., 2024).
En 2009, la FDA dio un gran paso al comenzar a incluir información farmacogenómica en las etiquetas de los medicamentos, promoviendo así una medicina personalizada basada en el perfil genético de los pacientes. Este avance marcó el inicio de la integración formal de la farmacogenómica en la práctica clínica, permitiendo a los médicos tomar decisiones de tratamiento basadas en el perfil genético del paciente. La FDA ha seguido actualizando estas etiquetas, lanzando en 2014 una segunda publicación de estas (Ampong, 2019; FDA). Posteriormente, el CPIC ha publicado guías específicas para el uso de medicamentos, ayudando a los médicos a tomar decisiones más informadas en función del genotipo de sus pacientes. A partir de ahí y hasta la actualidad se han ido publicando más guías y recomendaciones (Bousman et al., 2023; CPIC).
En 2016 nace el Consorcio de Farmacogenómica Europeo (U-PGx – Ubiquitous Pharmacogenomics Consortium) con el objetivo de que la optimización efectiva del tratamiento sea accesible para todo ciudadano europeo (Weinshilboum y Wang, 2017; Ubiquitous Pharmacogenomics (U-PGx).
En 2017, se estableció PharmVar, un repositorio central de variaciones farmacogenética que se centra en la estructura de haplotipos y la variación alélica, que nace como sucesor del Human Cytochrome P450 (CYP) Allele Nomenclature Database, incluyendo otros genes farmacogenéticos importantes, convirtiéndose en una plataforma centralizada para la estandarización de la nomenclatura de las variantes (Bousman et al., 2023; PharmVar).
Además, en 2020 la FDA aprobó el test de 23andMe, basado en variantes del CYP2C19, permitiendo que las pruebas genéticas personalizadas fueran más accesibles al público general (Bousman et al., 2023).
La farmacogenómica en la actualidad
En la actualidad, la farmacogenómica ha alcanzado un punto de inflexión gracias a los avances tecnológicos en secuenciación y almacenamiento de datos. El costo de la secuenciación ha disminuido drásticamente, desde los 300 millones de dólares necesarios para secuenciar el primer genoma humano en 2003, hasta aproximadamente 100 dólares por genoma en la actualidad. Esto, sumado a la capacidad de completar la secuenciación de un genoma en tan solo 8 horas, ha permitido integrar esta herramienta en los flujos clínicos, haciéndola más viable (Relling y Evans, 2015; Cecchin y Stocco, 2020; Sadee et al., 2023).
A pesar de estos avances, la implementación clínica de la farmacogenómica sigue siendo limitada. Solo un pequeño porcentaje de los medicamentos aprobados contiene información farmacogenómica en sus etiquetas, y menos del 7% son accionables en términos de recomendaciones basadas en genotipos (Ehmann et al., 2015). Sin embargo, estos medicamentos representan aproximadamente el 18% de las recetas en los Estados Unidos, lo que destaca su relevancia en la práctica médica (Dunnenberger et al., 2015). Asimismo, aunque genotipos clínicamente accionables para al menos un «farmacogén» están presentes entre el 90% y el 95% de las personas, persisten desafíos logísticos, como garantizar el reembolso de las pruebas, gestionar la portabilidad de datos, y mantener la privacidad y seguridad de la información genética (Relling y Evans, 2015). La lucha contra estos desafíos la lideran las grandes organizaciones de farmacogenómica ya mencionadas, gracias a los esfuerzos realizados para el reconocimiento e implantación clínica de esta disciplina a través de manuales, guías clínicas, plataformas y bases de datos actualizadas y accesibles, así como evaluaciones de la utilidad clínica y de la validez de aplicaciones genómicas emergentes y la búsqueda de financiación.
Declaración de conflicto de intereses
Paula Muñoz Yélamo forma parte de la empresa Duponte Investigación y Desarrollo S.L.

