INTRODUCCIÓN
El análisis del genoma humano ha avanzado significativamente en las últimas décadas, gracias a tecnologías como la secuenciación de última generación (NGS, del inglés, next generation sequencing). Sin embargo, la detección de variantes estructurales complejas (como inversiones, translocaciones o grandes deleciones) sigue siendo un desafío, requiriendo en muchos casos la aplicación de varias técnicas como son el cariotipo, el FISH y los microarrays genómicos. En este contexto, el Mapeo Óptico del Genoma (MOG) ha surgido como una herramienta prometedora ya que permite estudiar, en un único experimento, alteraciones numéricas y estructurales (balanceadas y no balanceadas) con mayor resolución y sensibilidad que el cariotipo.
Este artículo explora en qué consiste esta técnica, sus aplicaciones, limitaciones y los retos actuales para su implementación generalizada.
¿EN QUÉ CONSISTE EL MAPEO ÓPTICO DEL GENOMA?
El mapeo óptico del genoma es una tecnología basada en la obtención de imágenes de moléculas de ADN que presentan un patrón de marcaje único en las diferentes regiones del genoma. De esta forma, mediante la comparación de cada una de las moléculas obtenidas con la del genoma de referencia permite identificar translocaciones balanceadas y no balanceadas, inversiones, inserciones, ganancias y/o pérdidas (Why Structural Variation Matters, 2025). En la Tabla 1 se muestra la sensibilidad máxima y resolución para cada una de las alteraciones (Bionano System Application Specifications, 2025).
| Tipo de alteración | Sensibilidad máxima | Resolución | |
| Análisis constitucional | Análisis muestras tumorales | ||
| Inserciones | 1% | ≥100 Kb | ≥3 Kb |
| Deleciones | 1% | ≥100 Kb | ≥3 Kb |
| Duplicaciones | 3% | ≥50 Kb | ≥50 Kb |
| Inversiones | 1% | ≥50 Kb | ≥50 Kb |
| Translocaciones | 5% | ≥70 Kb | ≥70 Kb |
| Ganancias, pérdidas y aneuploidías (>500 Kb) | 10-20% | ≥500 Kb | ≥500 Kb |
La técnica de mapeo óptico del genoma se puede llevar a cabo en muestras de sangre periférica, médula ósea, tejido fresco congelado o amniocitos. En todos los casos, se puede extraer de la muestra fresca o congelada, con un mínimo de 1.5 millones de células de material de partida. Además, en el caso de la médula ósea, es importante congelar o procesar la muestra dentro de las primeras 24 horas.
La técnica consta de 3 pasos (Saphyr system, 2025) (Figura 1):
· Extracción de ADN de alto peso molecular (longitud mínima de 150Kb) mediante un protocolo específico.
No sirve ADN almacenado extraído por métodos convencionales. Además, al ser de textura muy viscosa, en ocasiones, es difícil la total homogenización del ADN por lo que se recomienda dejar a temperatura ambiente durante 3 días tras su extracción.
Actualmente, la extracción de ADN se lleva a cabo de forma manual pero Bionano Genomics ya tiene contemplado un robot para la extracción automática del ADN (Ionic®)(Ionic® Purification System, 2025). También es importante tener en cuenta que dada la alta fragilidad del ADN se recomienda evitar su transporte una vez extraído. Tras la extracción de ADN hay un control de calidad cuantitativo (cuantificación de ADN de doble cadena), se requiere una cantidad mínima de 500ng para proceder al paso 2(DNA isolation protocols, 2025).
· Marcaje enzimático (DLE-1) en secuencia específica (CTTAAG) que se repite de media cada 6Kb aproximadamente, seguido de un paso de tinción del resto de la molécula de ADN.
El marcaje con fluorescencia genera un patrón de etiquetas para la identificación de cada una de las regiones genómicas. En este paso también se lleva a cabo una tinción de todo el ADN para permitir la detección de las moléculas individuales de ADN. Tras este paso hay un segundo control de calidad cuantitativo (cuantificación del ADN de doble cadena), de forma que el ADN marcado debe tener una concentración mínima de 4 y máxima de 16ng/uL (Bionano Prep® DLS-G2 Protocol, 2025).
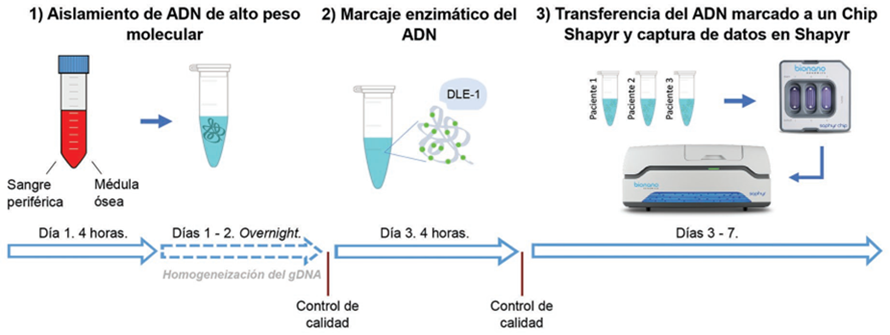
· Carga de la muestra en el chip para su posterior escaneado.
Actualmente hay dos máquinas que permiten el escaneado: el Saphyr y el Stratys. El Saphyr funciona con chips con 3 celdas de flujo (cada celda de flujo corresponde a una muestra) y permite cargar hasta 6 muestras diferentes.
El escaneado se produce en paralelo de cada una de las celdas de flujo. En el caso del Stratys, cada chip es para una única muestra y se pueden cargar hasta 12 chips en la máquina (lo que corresponde a 12 muestras) (Saphyr system, 2025; StratysTM System, 2025).
En cada carrera se programa la cantidad de ADN a capturar (en Gb del ADN total). En general, para muestras de cáncer se recomienda capturar 1500Gb para obtener una cobertura final de 300-400x, lo que permitirá identificar alteraciones con una sensibilidad del 5%. En el caso de estudios constitucionales, se recomienda capturar 800Gb para obtener una cobertura del 80x y así detectar alteraciones presentes en más del 40% de la muestra. Una vez el chip está en la máquina, a través de micro y nanocanales, el ADN se va linearizando y un sistema óptico captura imágenes de alta resolución de las moléculas de ADN.
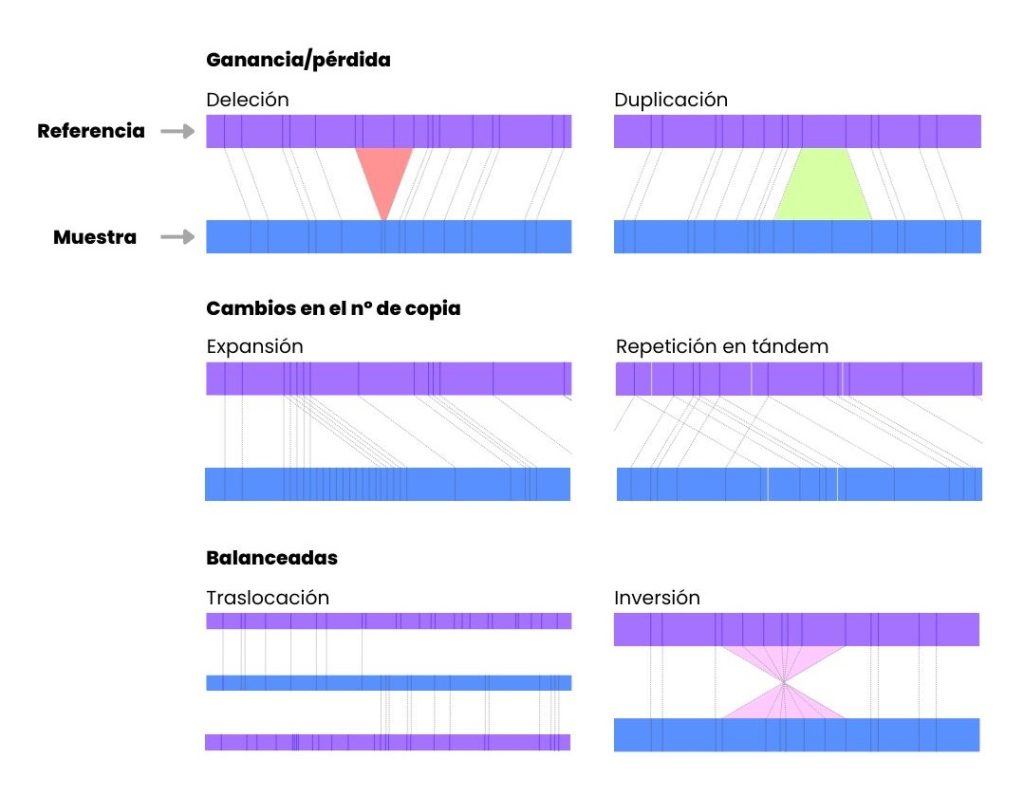
Posteriormente, el software compara las imágenes con una referencia del genoma, identificando variantes estructurales o numéricas. La Figura 2 muestra como la presencia, ausencia o cambio de etiquetas permite la identificación de las diferentes alteraciones en base a un genoma de referencia. El análisis se puede realizar mediante el software Bionano Access o VIA, ambos de Bionano Genomics (Why Structural Variation Matters, 2025).
APLICACIONES DEL MAPEO ÓPTICO DEL GENOMA
El mapeo óptico del genoma es una tecnología con gran aplicabilidad en la clínica, tanto en neoplasias como estudios constitucionales postnatales.
Diagnóstico genético en oncología.
En el caso de la oncología es de especial interés en oncohematología ya que las leucemias, linfomas y otras enfermedades hematológicas se caracterizan por la presencia de cambios genéticos específicos de cada entidad. El mapeo óptico del genoma evita así la aplicación de varias tecnologías (cariotipo, FISH y/o microarrays) para llegar al diagnóstico final.
Cada vez hay más artículos científicos que muestran una concordancia superior al 90% en los resultados de mapeo óptico del genoma con técnicas convencionales en diferentes neoplasias hematológicas (Neveling et al., 2021; Puiggros et al., 2022; Yang et al., 2022).
Estudios constitucionales postnatales
El mapeo óptico del genoma es especialmente útil en el caso de enfermedades genéticas que no se han podido resolver por otras técnicas como secuenciación del exoma o microarrays. Puede ser de gran utilidad para la identificación de reordenamientos complejos con o sin pérdida de material genético.
Un estudio reciente de Iqbal y cols. muestra como el mapeo óptico del genoma se puede usar como alternativa en el estudio de casos constitucionales posnatales (Iqbal et al., 2023).
LIMITACIONES DEL MAPEO ÓPTICO DEL GENOMA
Aunque el mapeo óptico del genoma es una técnica potente, presenta ciertas limitaciones y, por consiguiente, son desafíos a los que nos enfrentamos para su estandarización y su adopción más amplia:
· Estandarización de protocolos
Es necesario desarrollar protocolos más robustos y accesibles para la preparación de muestras. En el caso de la médula ósea, se requiere su procesamiento dentro de las 24 horas posteriores a la extracción.
La técnica requiere un mínimo de 1.5 millones de células para iniciar el protocolo.
En cuanto a calidad del ADN, la obtención de ADN de alta calidad y longitud puede ser difícil en algunas muestras, como tejidos fijados en parafina.
· Sensibilidad y clonalidad:
El mapeo óptico del genoma presenta una sensibilidad elevada (alrededor del 5%) pero para alteraciones de cambio en el número de copias (>500Kb) es del 15-20%.
Este hecho puede ser relevante en la identificación de alteraciones puntuales con valor pronóstico, como es el caso de la deleción de TP53 y de 13q en la leucemia linfática crónica (Puiggros et al., 2022).
· Identificación de regiones en homocigosis.
La importancia pronóstica de las regiones en homocigosis en las neoplasias hematológicas o enfermedades recesivas es ampliamente conocida. Actualmente, su detección es mediante microarrays de SNP.
El mapeo óptico del genoma permite la identificación mediante pipelines específicos, aunque presenta una sensibilidad y resolución limitada en comparación con otras técnicas.
· Coste elevado
El coste aun es elevado, alrededor de los 400€ (solo reactivos) pero si se compara con la aplicación de un conjunto de técnicas combinadas (cariotipo, FISH, microarrays) es económico.
Es una técnica costosa lo que hace que sea de difícil implementación en algunos países.
Es fundamental optimizar el proceso para hacerlo económicamente viable a gran escala.
· Dependencia de software.
La interpretación de datos depende en gran medida de algoritmos de análisis, lo que puede generar desafíos en la estandarización. Además, es una técnica muy nueva y, aunque el software ofrecido por Bionano Genomics ha mejorado mucho, aún hay margen de mejora para que sea más user friendly.
· Integración con tecnologías existentes
El mapeo óptico del genoma es una técnica muy completa pero no permite la detección de mutaciones puntuales, por lo que debe complementarse con técnicas como NGS para ofrecer un análisis genómico completo.
Como ya pasa con otras técnicas, como los microarrays o la secuenciación, pueden existir regiones genómicas de difícil acceso debido a la estructura molecular del ADN. Al ser una técnica nueva, hay pocas bases de datos específicas para diferentes poblaciones y especies.
· Falta de guías internacionales.
En la actualidad no se dispone de guías de recomendación sobre el uso de mapeo óptico del genoma frente las técnicas convencionales (cariotipo, FISH y microarrays).
CONCLUSIÓN
El mapeo óptico del genoma se presenta como una herramienta innovadora para la detección de variantes estructurales complejas, mejorando la resolución y sensibilidad respecto a técnicas convencionales.
A pesar de su gran potencial en oncología y estudios genéticos, su implementación aún enfrenta desafíos, como la estandarización de protocolos, la integración con otras tecnologías, la dependencia de software específico y el coste.
Con el desarrollo tecnológico y la validación clínica, el mapeo óptico del genoma podría convertirse en un estándar en el diagnóstico genético y la investigación biomédica. Sin embargo, es fundamental la formación de los usuarios finales para asegurar una correcta interpretación de los resultados, así como ser conscientes de las limitaciones intrínsecas a la técnica.
Declaración de conflicto de intereses
El autor declara la ausencia de conflicto de intereses.
