¡Hoy es un día especial! Un día como hoy, 13 de agosto, aunque en 1918, nació una gran figura de la genética que cambió la ciencia de mediados y finales del siglo XX. Él diseñó un sencillo método para determinar la estructura de las proteínas, pero es más conocido en la comunidad científica como el padre de la era genómica, por el desarrollo de un método de secuenciación de ADN realmente útil. ¿Sabes de quién hablo? Efectivamente, hablo del mismísimo Frederick Sanger, bioquímico británico que desarrolló la archiconocida secuenciación de Sanger. ¿Quieres saber más sobre él? ¡Sigue leyendo!

Los primeros pasos del padre de la genómica, Frederick Sanger
El día 13 de agosto del año 1918, nació en Rendcomb, un pueblecito inglés, el pequeño Frederick Sanger. El joven Sanger, que quería ser médico al igual que su padre, estudió hasta el año 1939 en el St. John’s College de Cambridge, donde obtuvo su bachiller en ciencias naturales. Durante sus estudios, comenzó a interesarse por la Bioquímica. Allí, en Cambridge, obtendría su doctorado más tarde, en 1943.
Después de doctorarse, Sanger se unió al plantel del Departamento de Bioquímica de la Universidad de Cambridge. En este mismo departamento, Sanger elaboró un método para determinar la estructura aminoacídica de la insulina. El método consistía en degradar la insulina en fragmentos y transferirlos a un papel vegetal. Después, gracias a una corriente eléctrica, los diferentes fragmentos se movían más o menos hacia un lado del papel, lo que le ayudó a deducir la secuencia de la insulina. El descubrimiento de la estructura aminoacídica de esta molécula tan importante le valió un Premio Nobel de Química, en 1958. Pero la cosa no quedó ahí, pues Sanger es una de las pocas personas que ha obtenido dos Premios Nobel en toda la historia de la humanidad. ¡Menudo estaba hecho!

El segundo Premio Nobel
Años más tarde de su primer Premio Nobel, en la década de los años 70, Sanger desarrolló uno de los primeros métodos de secuenciación del ADN. Actualmente esta técnica se conoce como método de Sanger o método de secuenciación de didesoxinucleótidos. Sanger utilizó este método para determinar el genoma del fago Phi-X174. Esto fue una hazaña enorme, ya que se trataba del primer genoma secuenciado completamente. En 1980, Sanger fue galardonado con el Premio Nobel de Química, tanto por el desarrollo de la secuenciación de didesoxinucleótidos como por su trabajo con Phi-X174. Este Premio Nobel lo compartió con los estadounidenses Walter Gilbert y Paul Berg.
El método de Frederick Sanger
El método de secuenciación de didesoxinucleótidos o método de Sanger es una forma de secuenciar una secuencia de ADN relativamente sencilla, basada en la acción normal de las polimerasas de ADN. Para desarrollar este método, Sanger utilizó:
- ADN a secuenciar
- ADN polimerasa
- Primer o cebador
- Nucleótidos (A, T, C y G) con Fosfatos radiactivos (Fósforo 32)
- Didesoxinucleótidos (ddA, ddT, ddG y ddC), desoxirribonucleótidos con una pequeña modificación en el grupo unido al carbono 3 de la ribosa. Para que os hagáis una idea, os lo muestro en el siguiente esquema.
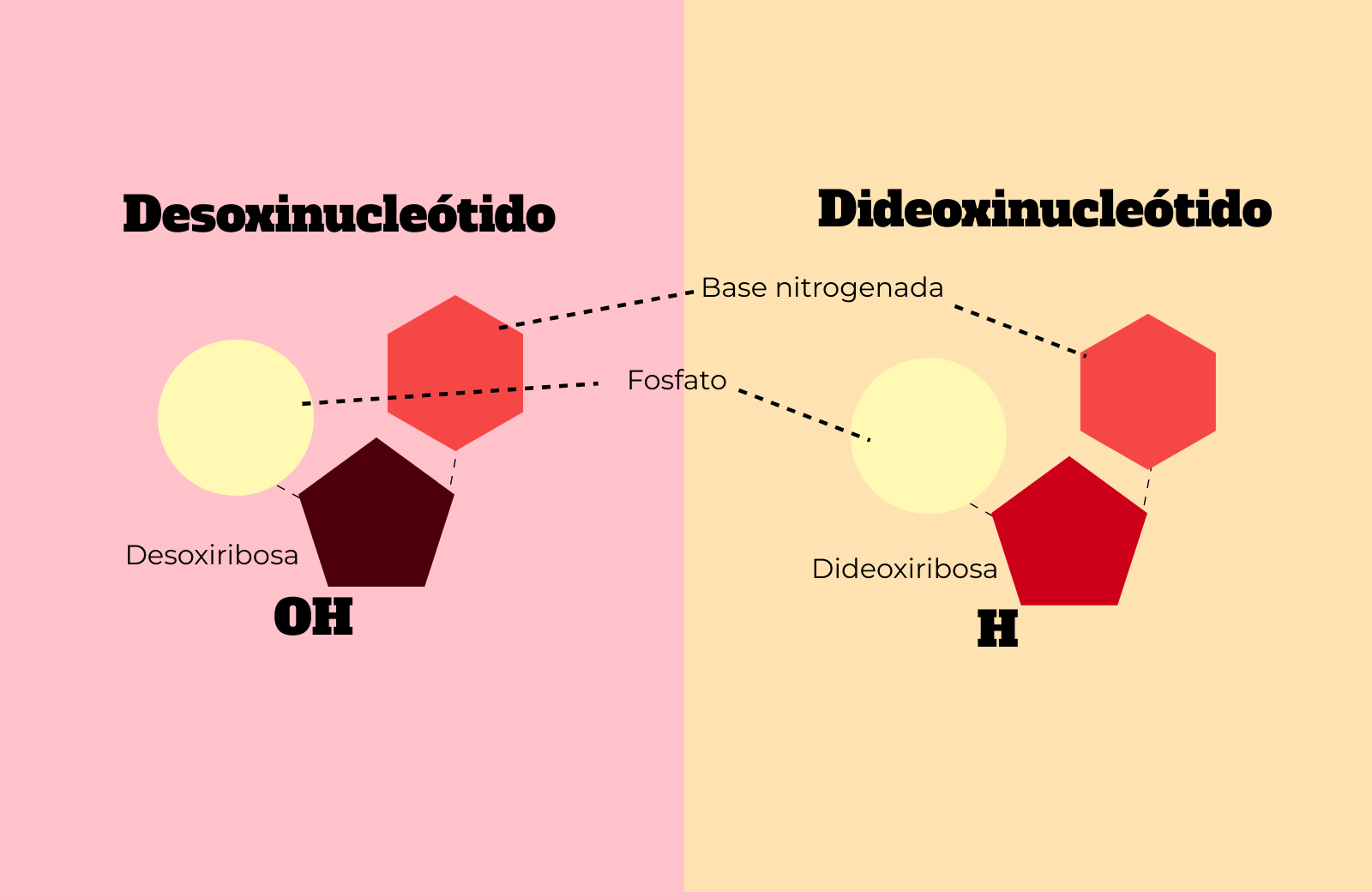
Como os decía, el método de secuenciación de Sanger se basa en la acción de las ADN polimerasas. Estos enzimas tan importantes para nuestras células son capaces de unirse a una cadena de ADN a la que se le haya unido un fragmento corto de ADN complementario (cebador) previamente e ir añadiendo nucleótidos. De este modo, a partir de una cadena simple, podemos obtener una doble cadena. Esto, de hecho, es lo que pasa en la secuenciación de Sanger, salvo por un ingenioso detalle, la presencia de didesoxinucleótidos. Cuando una polimerasa inserta un didesoxinucleótido, la síntesis de la nueva cadena se detiene.
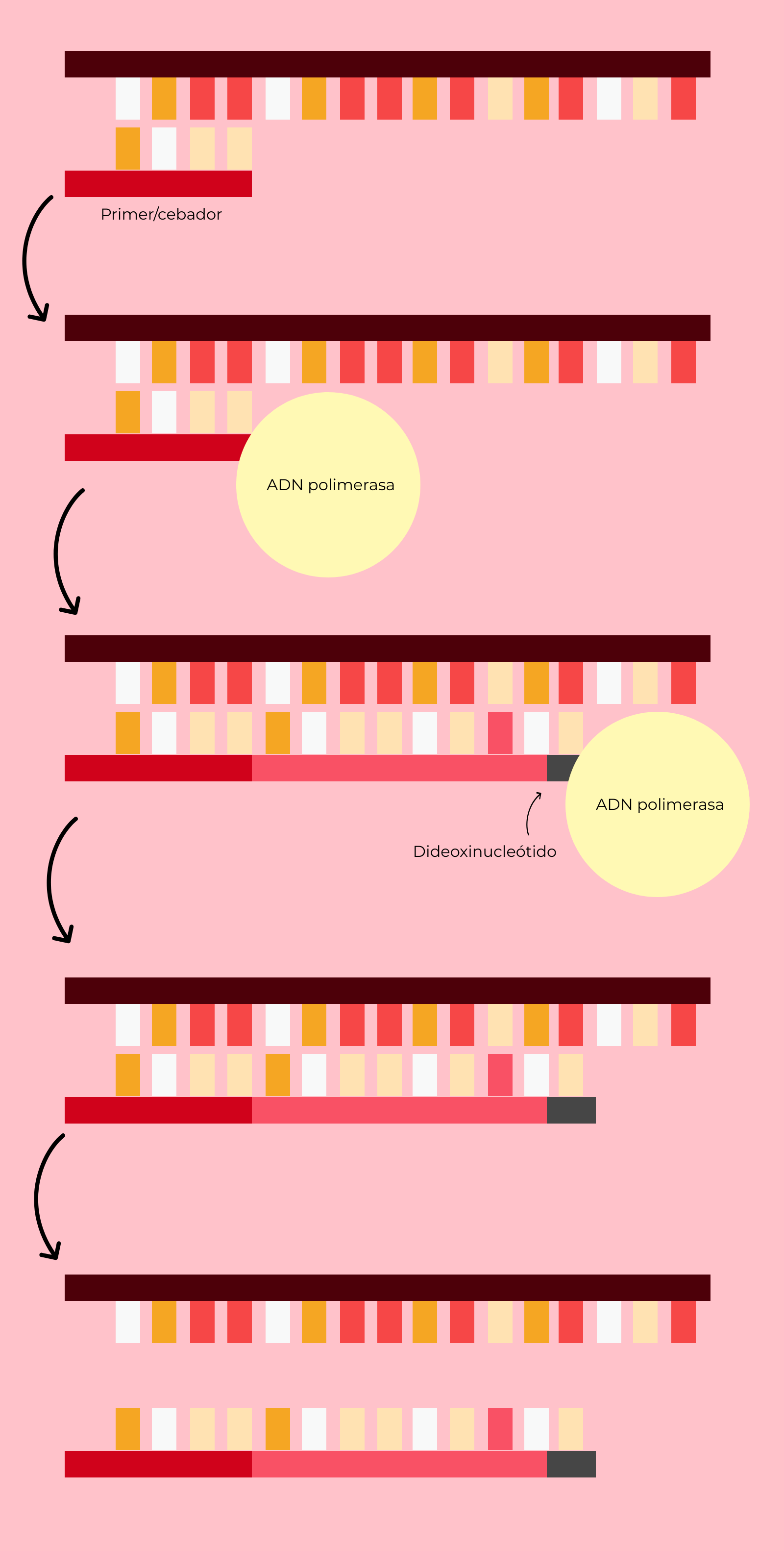
Para utilizar el método de Sanger, lo primero que hay que hacer es amplificar el ADN que queremos estudiar. En el caso de Sanger, fue el ADN del virus Phi-X174. Actualmente lo que se utiliza es la técnica de la PCR, pero en ese entonces no existía. Recordad que hasta 1983 no existía la PCR, así que Sanger seguramente clonase las cadenas de ADN de otro modo, por ejemplo dejando que el virus Phi-X174 se reprodujese en un cultivo celular.
El segundo paso es añadir este ADN a las 4 reacciones que se han de preparar, cada una de ellas con la ADN polimerasa (Sanger usó la de E. coli), un primer específico para la secuencia del virus Phi-X174 y uno de los cuatro tipos de nucleótidos con fosfatos radiactivos. En cada una de las disoluciones añadiremos una pequeña cantidad de un solo tipo de didesoxinucleótido.
Acto seguido, se aumenta la temperatura de las reacciones, para provocar la separación de las dos cadenas de ADN. Luego dejaremos enfriar para favorecer la unión de los primers o cebadores.
Una vez unido el cebador, comenzará la reacción de la ADN polimerasa, que irá añadiendo nucleótidos marcados, hasta que inserte un didesoxinucleótido, se detenga la síntesis y la nueva cadena se suelte. Entonces, la ADN polimerasa empieza de nuevo. De este modo, en cada una de las reacciones obtendremos fragmentos de diferentes tamaños, acabados en el didesoxinucleótido que hayamos empleado en ellas.
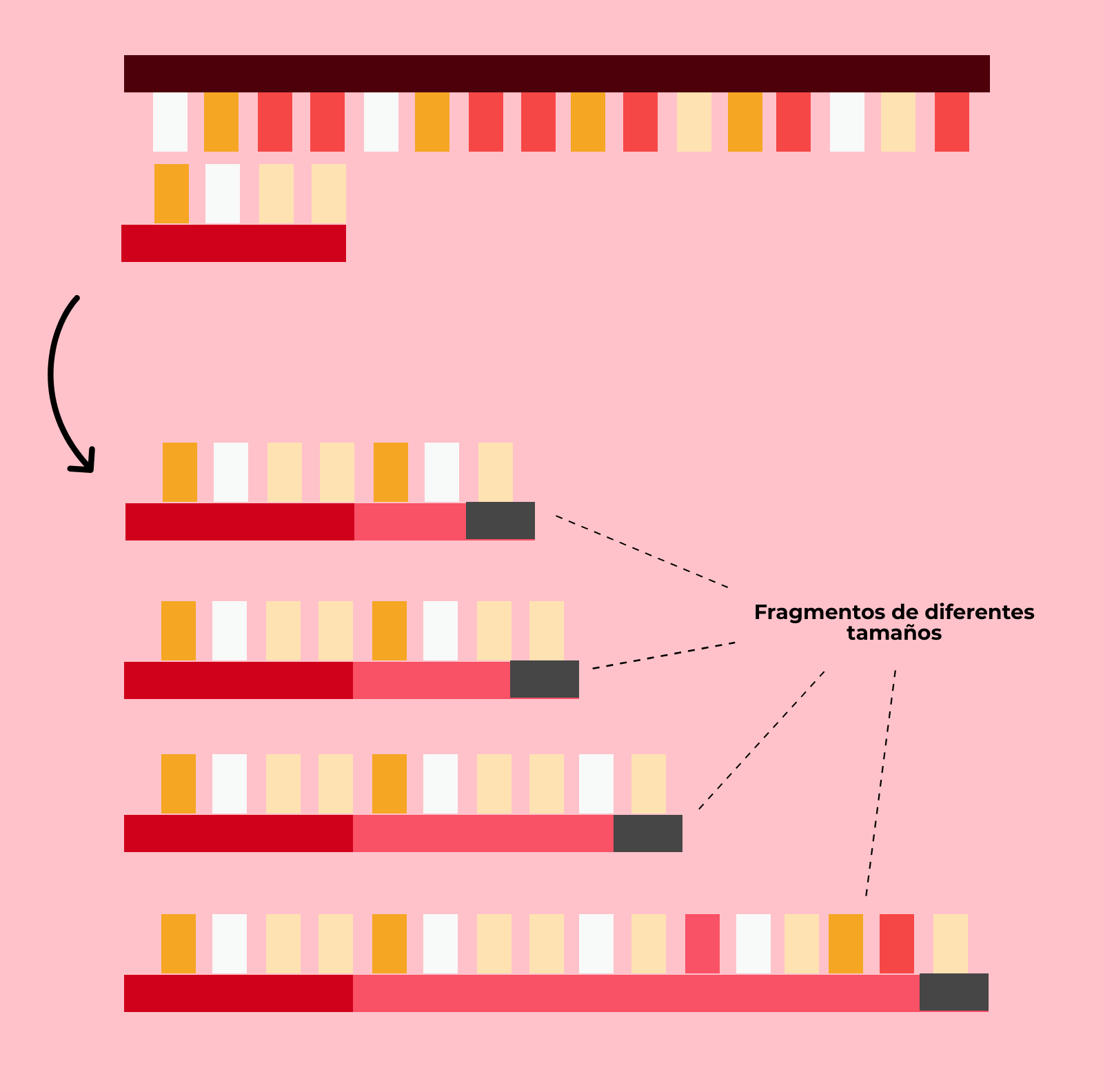
Posteriormente, el resultado de las cuatro reacciones se somete a una electroforesis. Para los que no conozcáis esta técnica, una electroforesis consiste en colocar una mezcla de moléculas con diferente tamaño y/o carga eléctrica en un “filtro” o soporte poroso, que puede ser un papel, en un gel o en acetato de celulosa. Este filtro se introduce en una solución y se somete a un campo eléctrico. De este modo, las moléculas se moverán por bandas más o menos rápido por el filtro según su carga y tamaño. Por ejemplo, el ADN, que tiene carga negativa, migrará hacia el polo positivo.
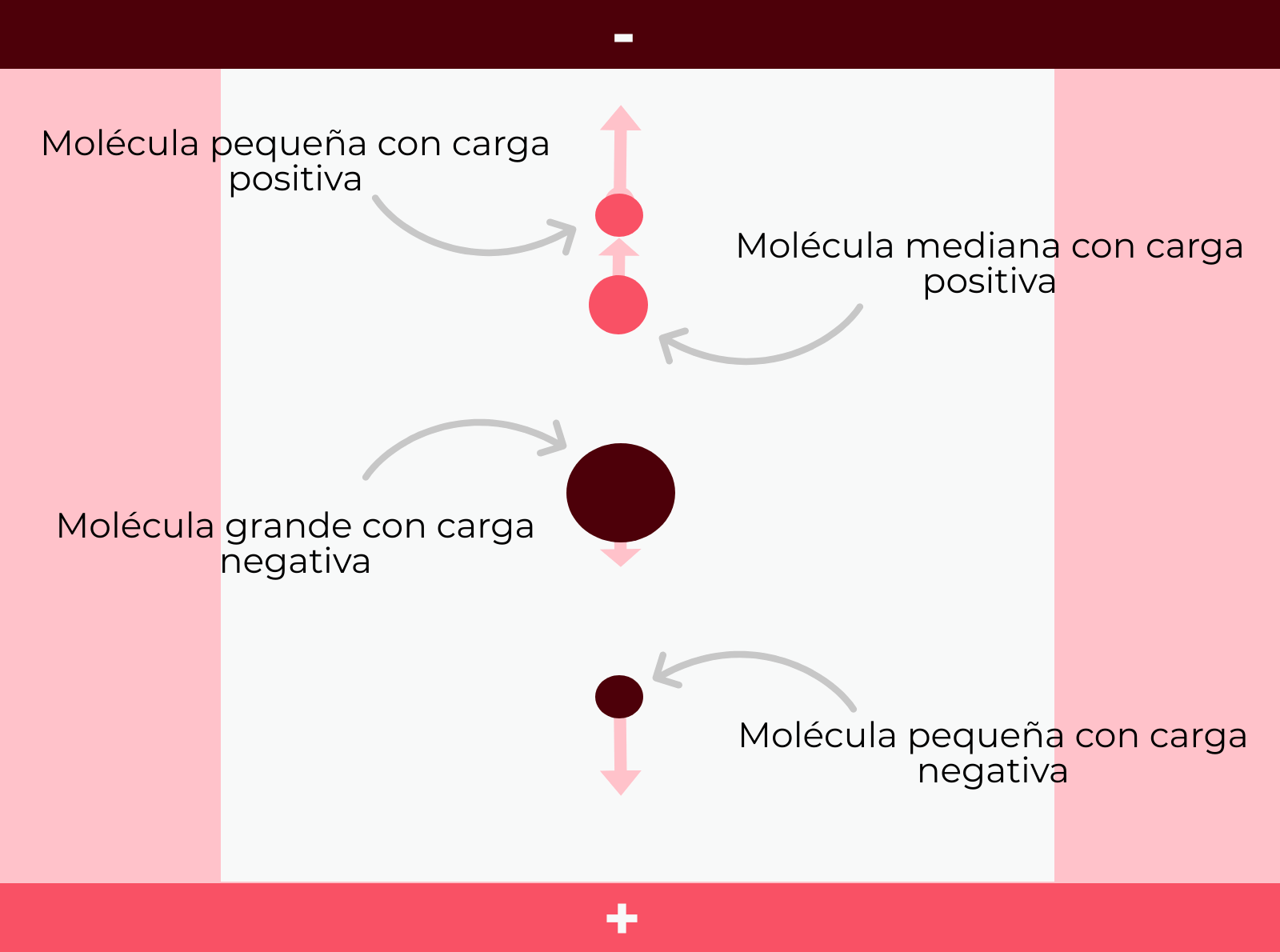
Volviendo al método de secuenciación de Sanger, veríamos algo similar a esto:
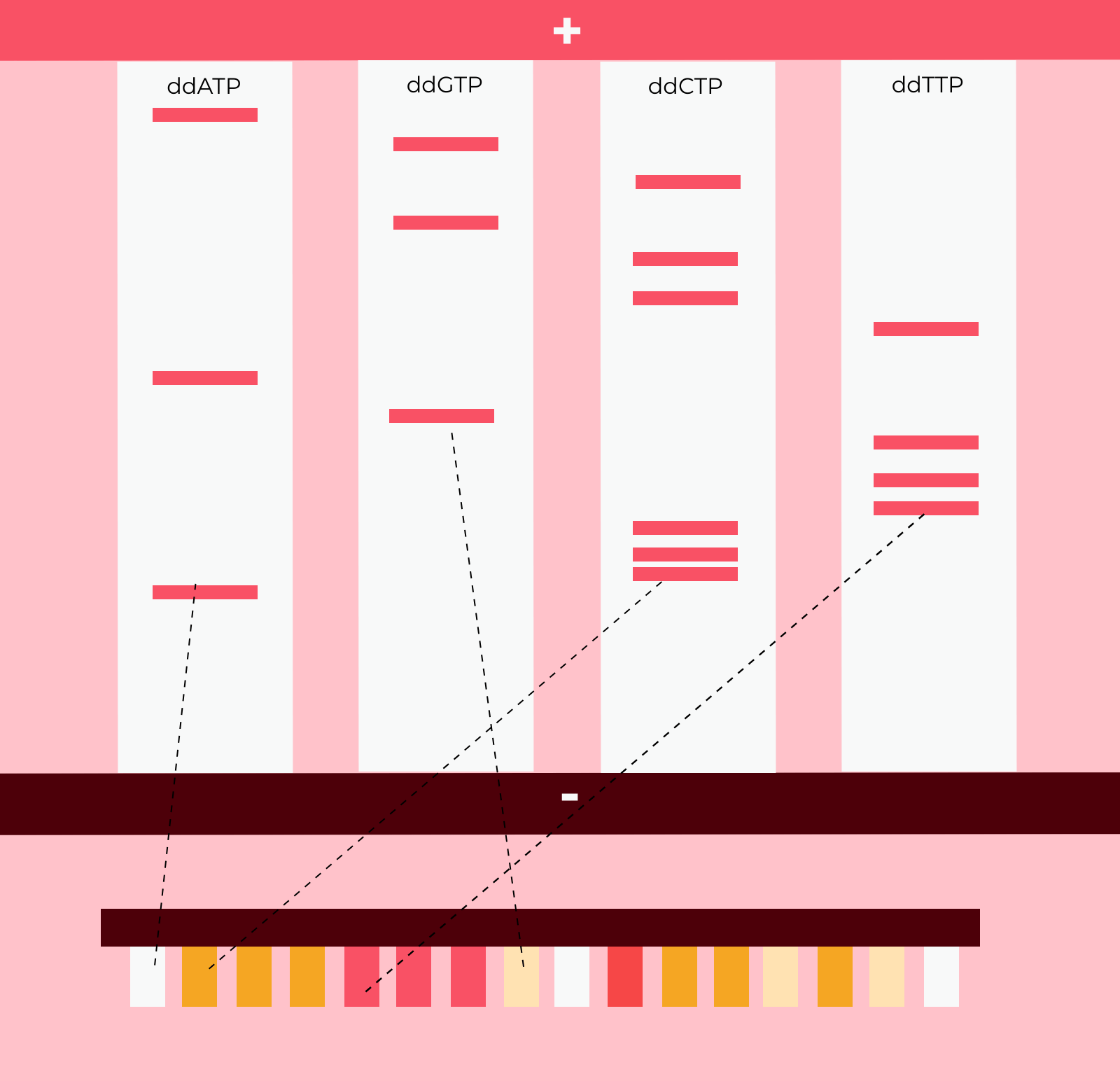
Combinando la información de las cuatro electroforesis, podemos establecer el orden de la cadena complementaria a la que estamos estudiando. Luego, solo hay que cambiar las bases por sus complementarias y ya tendríamos las dos. ¡Fantástico!
Secuenciación Sanger por electroforesis capilar
Frederick Sanger elaboró un método bastante bueno para secuenciar el ADN de un organismo. Sin embargo, todavía tenía muchas cosas por pulir. Eso de utilizar nucleótidos con isótopos radiactivos… Pues no era lo mejor, vaya. Afortunadamente, a finales del siglo XX se desarrollaron algunas tecnologías que hicieron este método mucho más sencillo. ¡Y pensar que Sanger lo hizo todo a mano!
Por un lado, el desarrollo de la PCR en los años 80 facilitó muchísimo el conseguir suficiente muestra de ADN a secuenciar. Por otro lado, las técnicas de electroforesis capilar y su automatización favorecieron modificaciones en el método de Sanger que perduran hasta el día de hoy: se dejó de utilizar nucleótidos radiactivos y se añadieron marcadores fluorescentes a los didesoxinucleótidos (cada uno de ellos de un color diferente). ¡Más visual!. De este modo, lo que antes veíamos como en la figura anterior, se puede ver como en esta figura. ¡Mucho más ordenado y vistoso!
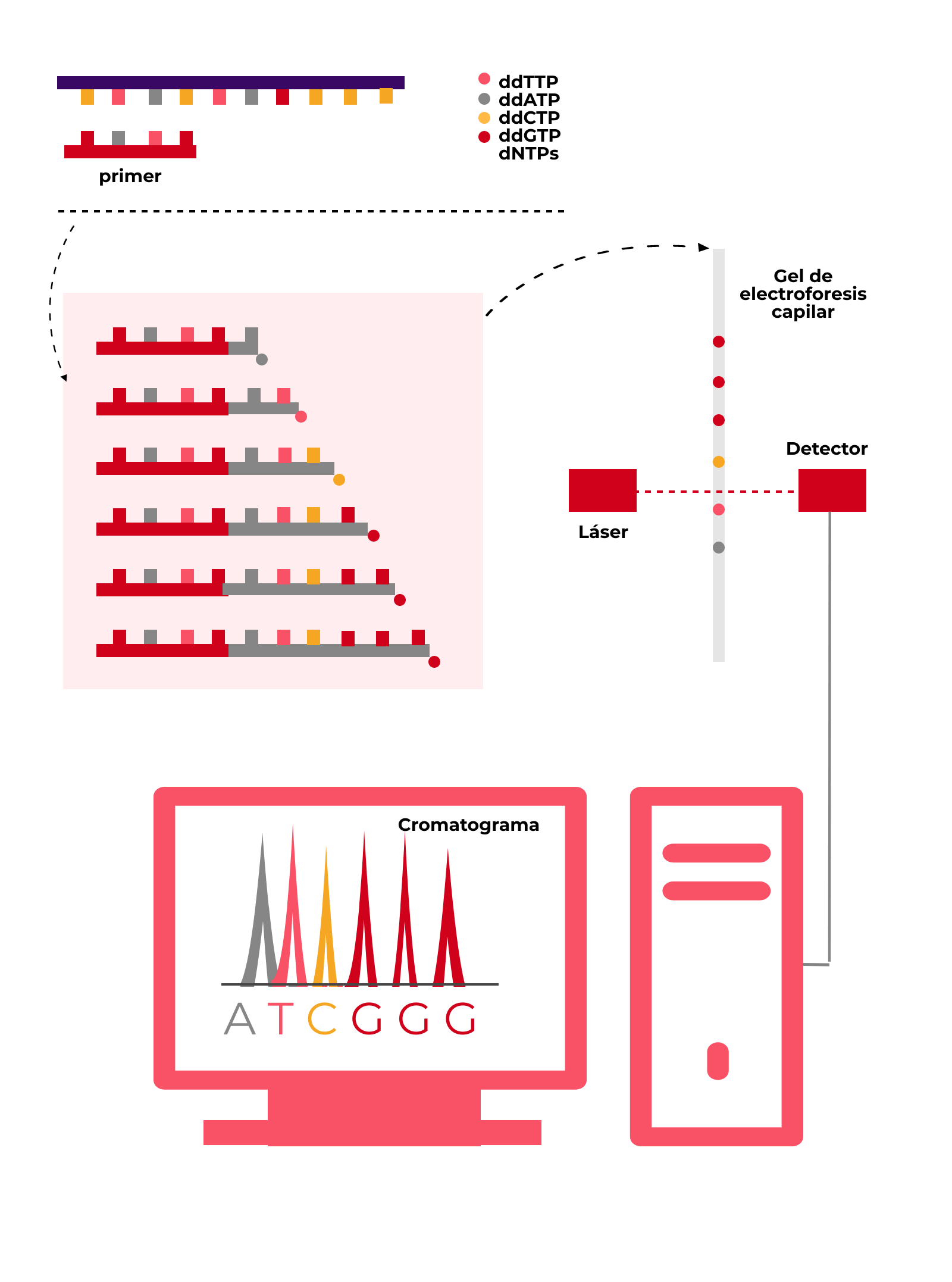
Aplicaciones de la Secuenciación de Sanger
La secuenciación de didesoxinucleótidos fue un bombazo científico en la década de los 70, que impulsó la creación de proyectos tan importantes como el Proyecto Genoma Humano, que empezaba a “cocinarse” como una posibilidad entre los científicos de todo el mundo. Después de unos años, en los años 90, comenzó formalmente este proyecto, gracias al avance en este tipo de técnicas de secuenciación y a la aparición de la PCR.
Actualmente la secuenciación de Sanger sigue utilizándose en los laboratorios, pero tiene sus limitaciones. Por ello, se han desarrollado diferentes técnicas de secuenciación de nueva generación, mucho más rápidas y económicas.
Y hasta aquí el blog de hoy. Ya conocéis la historia de Frederick Sanger, una de las pocas personas que ha ganado dos premios Nobel y el padre de la genómica. ¡Espero que os haya gustado! Nos leemos en el próximo post.