Hace poco publicamos en nuestro blog un nuevo post sobre la técnica bioquímica de la PCR, en el que explicamos en qué consiste. En este post os mostraré una de las variantes más utilizadas de la PCR, la RT-PCR cuantitativa, y cómo puede utilizarse para diagnosticar la infección por el coronavirus SARS-CoV-2.
Coronavirus SARS-CoV-2
El coronavirus SARS-CoV-2 es una especie de virus que apareció a finales de 2019 en el territorio de Wuhan, en China. En humanos este virus es capaz de causar diferentes afecciones respiratorias agudas y neumonías.
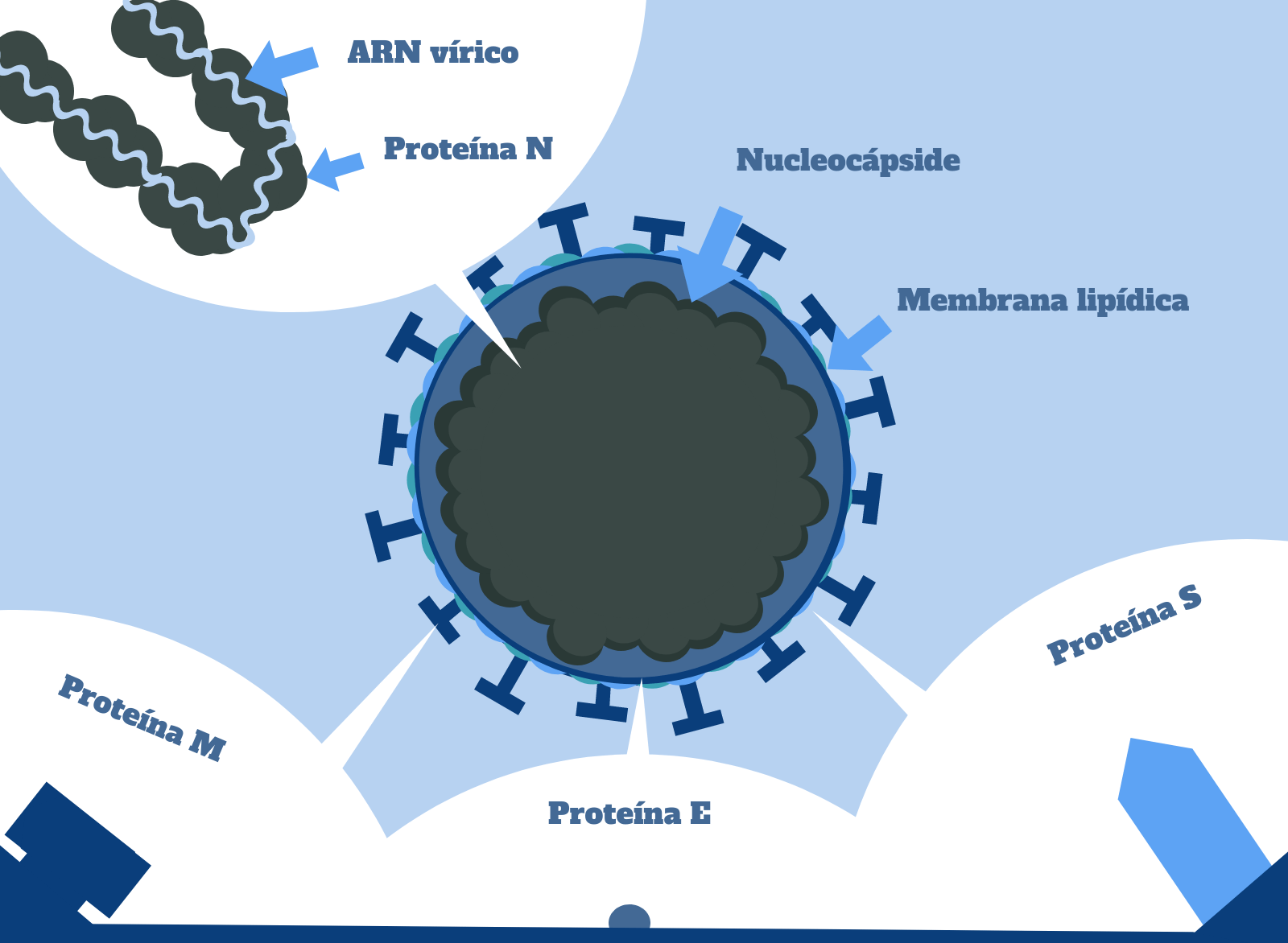
Los coronavirus son virus de ARN monocatenario positivo recubiertos por una estructura de glicoproteínas y lípidos. Eso quiere decir que, a diferencia de nosotros, los humanos, el SARS-CoV-2 tiene su material genético en forma de ARN. Pero… ¿la PCR sólo era capaz de amplificar el ADN, no? Cierto, las ADN polimerasas sólo pueden utilizar el ADN como molde y no el ARN. Entonces ¿cómo es posible utilizar la PCR para detectar la infección por coronavirus? Pues… utilizando una variante de la PCR estándar, la RT-PCR, que se vale de la ayuda de un enzima muy particular, la transcriptasa inversa.
Obtención de la muestra y conversión de ARN a ADN mediante Transcriptasa inversa
La transcriptasa inversa es una ADN polimerasa de origen vírico un tanto especial. Mientras el resto de ADN polimerasas sólo puede obtener ADN a partir de una cadena de ADN, la transcriptasa inversa puede sintetizar ADN a partir de una molécula de ARN. ¡Como el de SARS-CoV-2!
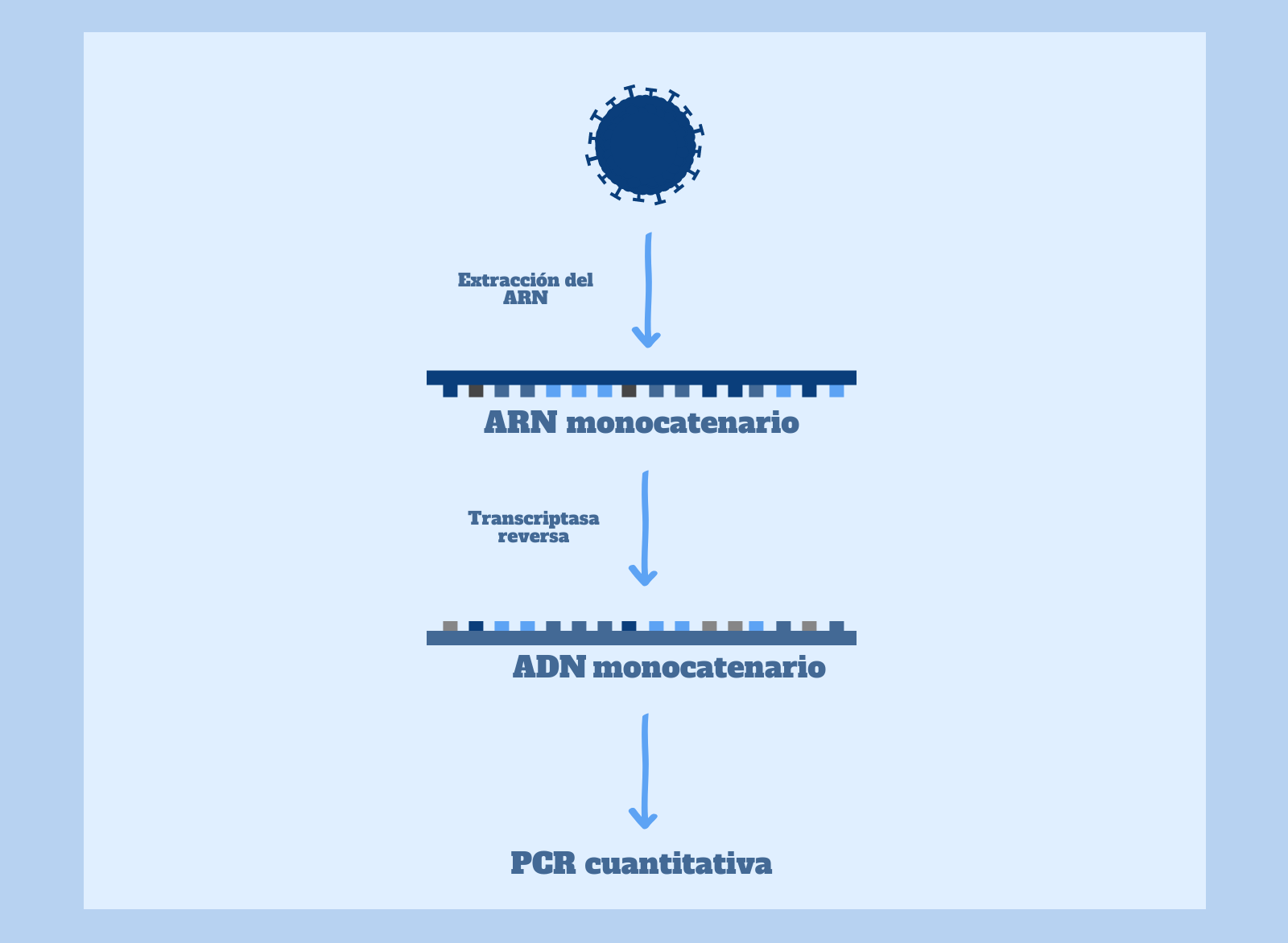
Por tanto, el primer paso para detectar la infección por SARS-CoV-2 mediante PCR es la conversión del ARN monocatenario viral en ADN . Para ello, en primer lugar, se obtiene el material genético del virus a partir de un frotis de nariz o garganta del paciente a diagnosticar y se purifica. Debemos tener en cuenta que en la muestra estamos recogiendo también ARN humano, ARN bacteriano e incluso ARN de otros virus. ¡Menudo lío!
Acto seguido, la muestra de ARN obtenida y purificada se mezcla con la transcriptasa inversa (y otros reactivos), para obtener cadenas de ADN, que podemos cuantificar mediante una PCR cuantitativa. Aquí habrá ADN de muchos orígenes (humano, vírico y bacteriano), pero no todo se amplificará en la PCR, ya que es una técnica dirigida a ciertas secuencias específicas (en este caso, secuencias del ADN retrotranscrito del virus).
PCR cuantitativa
Como os comentaba en el post anterior sobre la técnica de la PCR, la PCR cuantitativa es una variante de la PCR que nos permite medir en tiempo real la cantidad de fragmentos de ADN que se van produciendo. Para poder cuantificar la muestra de un paciente en este tipo de PCR, se añaden al tubo de ensayo sondas que se unen únicamente a secuencias específicas del ADN retrotranscrito del virus y emiten fluorescencia. Por tanto, a mayor fluorescencia en la muestra, mayor cantidad de copias del ADN obtenido mediante la retrotranscripción del virus SARS-CoV-2.
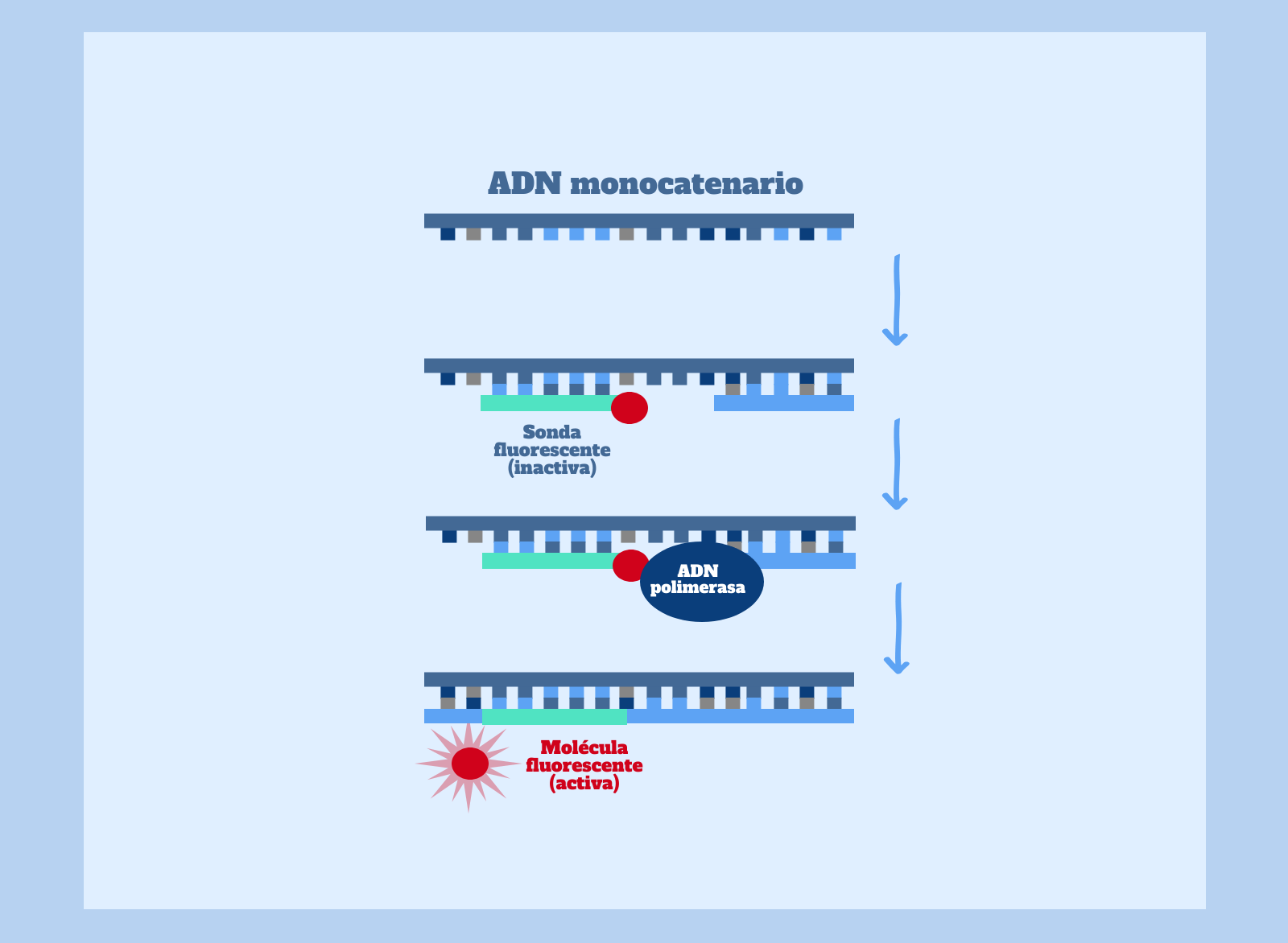
Análisis de Resultados
Una vez se está produciendo la reacción de la PCR cuantitativa de la muestra purificada y retrotranscrita del paciente, podemos obtener los siguientes resultados:
Presencia de fluorescencia en la PCR cuantitativa:
Si se detecta un aumento de la fluorescencia durante la reacción de PCR, estamos ante un claro indicio de la presencia de SARS–CoV-2 en el paciente. Recordad que, en esta prueba diagnóstica, la fluorescencia es producto de la amplificación del ADN que hemos obtenido de la retrotranscripción del ARN del virus.
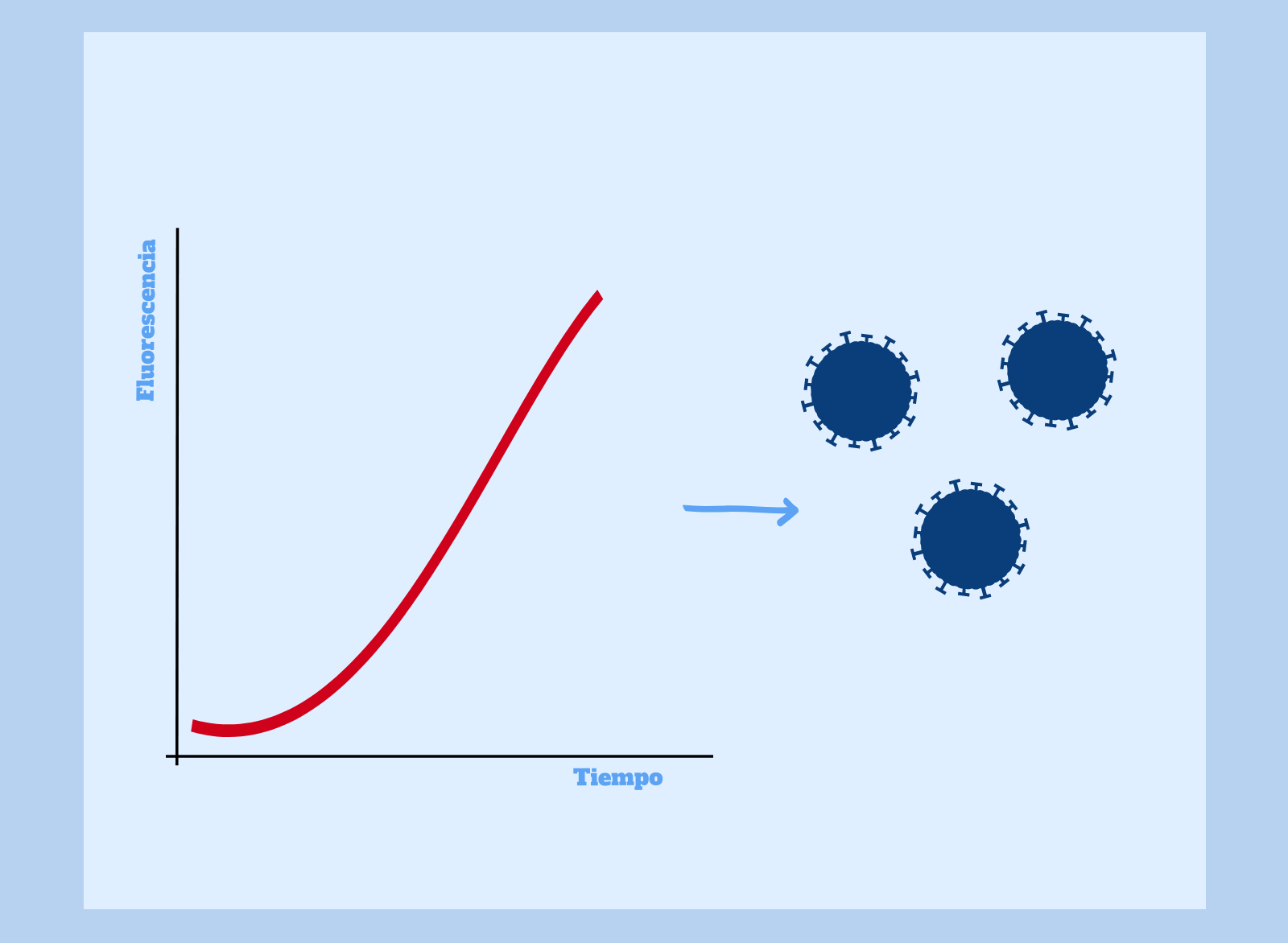
En este caso, diríamos que la prueba ha dado positivo, es decir, el paciente se encuentra infectado, en cierta medida, por SARS-CoV-2.
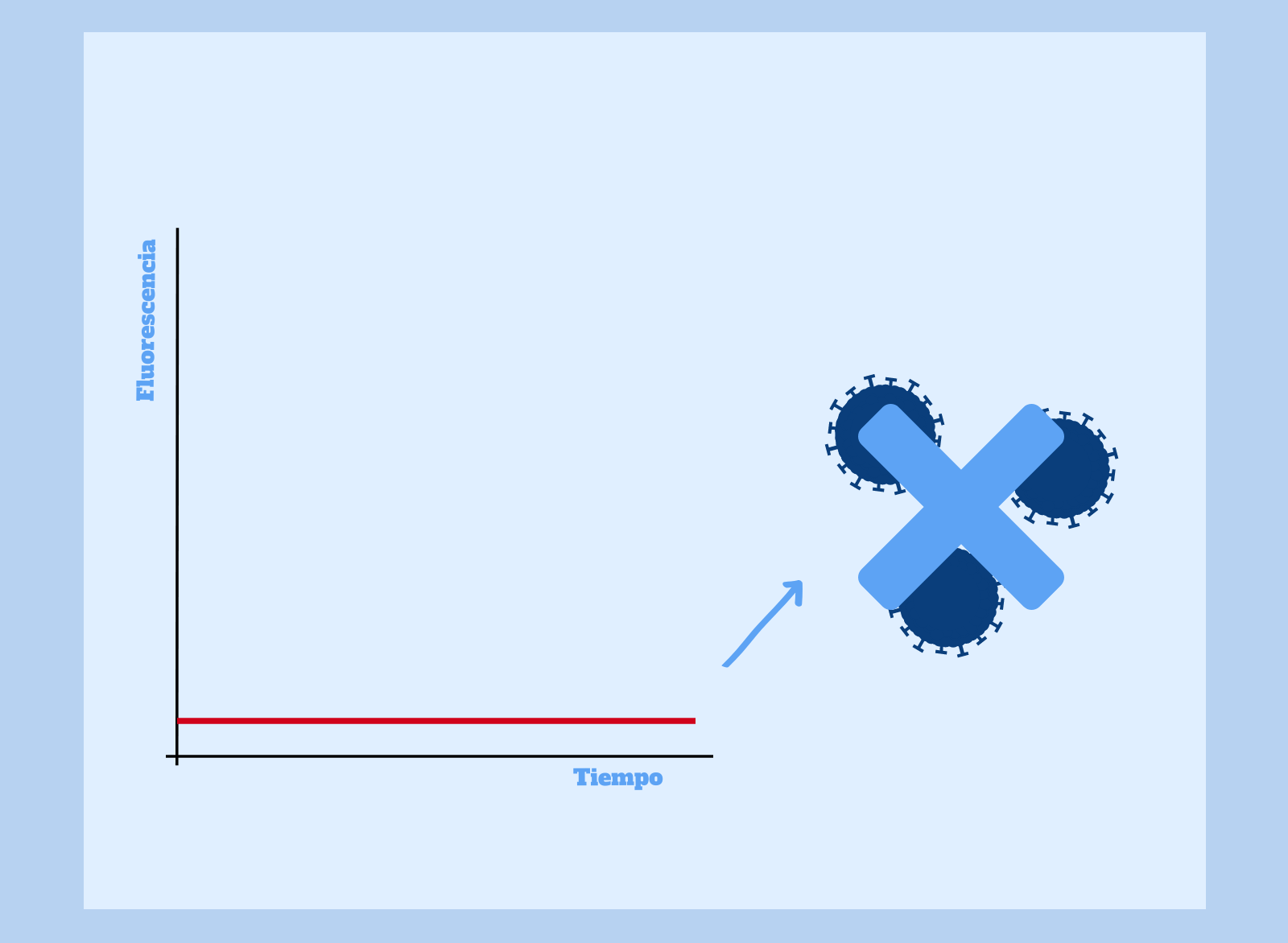
Ausencia de fluorescencia en la PCR cuantitativa:
Es posible que la prueba no detecte un aumento de la fluorescencia durante la reacción de PCR. En este caso, diríamos que la prueba ha resultado negativa y, por tanto, el paciente no se encuentra infectado por el virus SARS-CoV-2.
Limitaciones de la prueba mediante RT-PCR cuantitativa
Aunque la RT-PCR cuantitativa es una técnica muy interesante a la hora de detectar la infección por SARS-CoV-2 de los pacientes posiblemente infectados, presenta varias limitaciones asociadas que la hacen menos efectiva de lo que debería.
La primera de las limitaciones de las pruebas diagnósticas de SARS-CoV-2 mediante RT-PCR cuantitativa es que solo pueden determinar la infección por SARS-CoV-2 en el momento de la prueba. Esto quiere decir que, utilizando esta técnica, no podemos saber si un paciente estaba infectado días antes de la prueba.
Otra de las limitaciones de la RT-PCR cuantitativa es la velocidad a la que se lleva a cabo. Aunque, por lo general, la PCR es una técnica bastante rápida para amplificar muestras de ADN, se demora varias horas hasta poder establecer unos resultados. El diagnóstico mediante este método es, por tanto, lento en la situación actual, en la que se necesitan resultados rápidos para poder controlar a los pacientes infectados.
Por último, aunque la RT-PCR cuantitativa es una técnica relativamente fiable, se pueden producir falsos positivos o falsos negativos. Se denominan falsos negativos a todos aquellos resultados negativos de pacientes infectados por SARS-CoV-2, mientras que se considera falso positivo a un resultado positivo de un paciente no infectado. Estos errores en el diagnóstico pueden determinar incorrectamente el seguimiento de los pacientes.
Test alternativos para detectar SARS-CoV-2
Para suplir las limitaciones de la RT-PCR cuantitativa para el diagnóstico de infecciones de SARS-CoV-2, se están desarrollando diferentes técnicas de diagnóstico alternativas. Es el caso de los test de anticuerpos que se utilizan en el diagnóstico de SARS-CoV-2 o los test basados en CRISPR-Cas13 que se están desarrollando.
Test de anticuerpos
Los test de anticuerpos para la detección de SARS-CoV-2 son pruebas diagnósticas capaces de determinar si un paciente ha sido infectado y ya se encuentra inmunizado. Este tipo de pruebas consigue detectar los anticuerpos que el sistema inmunitario del paciente ha generado contra el virus. Una de las grandes ventajas de este test es el tiempo en el que se llevan a cabo, ya que solo se tardan unos 15 minutos en obtener resultados.
El problema principal de este tipo de test es que no pueden determinar una infección reciente del paciente. Es decir, que si el paciente acaba de ser infectado por SARS-CoV-2 puede dar un resultado falso negativo, ya que la persona todavía no ha tenido tiempo para desarrollar anticuerpos frente al virus. Por ello, se recomienda como prueba complementaria a la RT-PCR cuantitativa.
Test basados en CRISPR-Cas13
Hace unos años (exactamente en 2017) un equipo de investigadores publicó en la revista Science una herramienta excelente que utilizaba CRISPR para detectar ácidos nucleicos. Estos científicos llamaron a esta técnica “SHERLOCK”. Actualmente se está desarrollando un test de detección de SARS-CoV-2 a partir de SHERLOCK. Sin embargo, todavía no se ha probado mediante ensayos clínicos, por lo que no podríamos considerarlo todavía una técnica de diagnóstico.
En este tipo de pruebas, gracias al sistema CRISPR-Cas13, podemos detectar la infección por SARS-CoV-2 a partir de una muestra de una forma rápida y visual. En este caso, el sistema se encuentra en laminillas de papel que, al ser sumergidas en una muestra se colorea, indicando la presencia de moléculas de ARN de SARS-CoV-2. Para que os hagáis una idea, visualmente es similar a una prueba de embarazo.
Otros test
Además de los test anteriores, algunos centros europeos, entre los que figura el Instituto Catalán de Nanociencia y Nanotecnología, están trabajando en un nuevo dispositivo que se espera que detecte la infección por coronavirus en menos de 30 minutos. Este nuevo dispositivo, al que los autores han llamado “CoNVat” se basa en tecnologia de microchip y guías de onda interferométricas (nanotecnología óptica) para detectar el virus.
Si te interesan los virus y quieres conocer mejor las técnicas más comunes empleadas para la investigación básica de virus, te interesa nuestro programa «Virología Aplicada a la Investigación en Biomedicina«.
Bibliografia:
Enabling coronavirus detection using CRISPR-Cas13: Open-access SHERLOCK research protocols and design resources. Broad Institute: https://www.broadinstitute.org/news/enabling-coronavirus-detection-using-crispr-cas13-open-access-sherlock-research-protocols-and
Así son las pruebas PCR que se utilizan para detectar el coronavirus. Agencia SINC: https://www.agenciasinc.es/Noticias/Asi-son-las-pruebas-de-secuenciacion-que-se-utilizan-para-detectar-el-coronavirus