Los humanos compartimos el 99,9% de nuestro ADN entre nosotros y, sin embargo, todos no somos copias exactas de tu vecino Paco. De hecho, presentamos una gran variabilidad fenotípica; cada uno de nosotros tiene una altura y talla de pie diferente y una estructura facial distintiva. Incluso el color y textura de nuestro pelo y piel es diferente dependiendo de la persona. Estas pequeñas diferencias son debidas, en gran parte, a cambios en una sola letra de nuestro “manual genético”, es decir, lo que conocemos en genética como SNP. En este post, nos centraremos en este tipo de cambios genéticos, cuál es su relevancia clínica y cómo es el análisis de SNPs. ¿Te interesa? ¡Sigue leyendo!
¿Qué es un SNP en genética?
La palabra SNP proviene del inglés “Single Nucleotide Polymorphism”, que se traduce literalmente como “Polimorfismo de de un Solo Nucleótido”. Se trata de un término que se utiliza para hacer referencia a una variación en la secuencia del ADN que ocurre cuando un solo nucleótido (una de las “letras” que componen la secuencia del ADN) es diferente entre varios individuos de una especie. De hecho, para que un cambio de un nucleótido se considere SNP, tiene que suceder en, al menos, un 1% de toda la población que se esté estudiando. De lo contrario, no sería considerado SNP, sino una mutación puntual.

Diferencias entre SNP, indel y otras variantes genéticas
Aunque los SNPs son variantes genéticas, se trata de un concepto algo más complejo que el de otras variantes de inserción o deleción (indels). Mientras que en las inserciones y deleciones estamos hablando del tipo de variante (la adición o pérdida de secuencia), la definición de SNP nos habla, además del tipo de variante, de su frecuencia poblacional. Y es que una variante genética solamente se considera SNP si se trata una sustitución, inserción o deleción de un solo nucleótido del ADN y se encuentra en más del 1% de la población.
SNP: fundamentos del análisis
No todos los SNPs afectan de igual manera. Y es que su influencia en el fenotipo (y, por ende, su relevancia clínica) depende muchísimo de en qué región del genoma se encuentren y del tipo de SNP. Para entender esto, primero recomendamos repasar cómo funciona el código genético y la traducción del ARN mensajero.
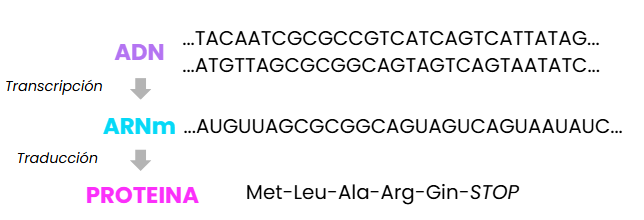
Tipos de SNPs según su localización y función
- SNPs sinónimos: el cambio de nucleótido no altera el aminoácido de la proteína resultante. Son generalmente “silenciosos”, aunque, en ocasiones, pueden afectar al splicing.
- SNPs no sinónimos (missense): estos SNPs cambian el aminoácido resultante tras la traducción del ARNm.
- SNPs terminadores (nonsense): el cambio de nucleótido provoca un codón de parada prematuro en la traducción del ARNm. Su efecto suele ser marcado
- SNPs en regiones no codificantes: SNPs que se producen en regiones reguladoras. En ocasiones pueden afectar negativamente a la regulación de los genes.
¿Cómo afecta la frecuencia alélica a la interpretación clínica?
Como comentábamos, la definición de SNP no solo nos habla del tipo de variación genética, sino también de su frecuencia dentro de la población. En el contexto de la interpretación de variantes, la frecuencia alélica es esencial, ya que si un SNP está muy presente en una población sana, suele ser indicativo de que se trata de una variante benigna o probablemente benigna. De hecho, es uno de los criterios que la American College of Medical Genetics and Genomics (ACMG) aconseja utilizar para la clasificación de variantes genéticas.

Concretamente, la ACMG considera que el hecho de que un SNP tenga una frecuencia alélica mayor del 5% ya es suficiente para considerarlo benigno (criterio BA1).
¿Para qué se utilizan los SNPs en medicina? Ejemplos de SNPs con relevancia clínica
No todos los SNPs son curiosidades evolutivas que afectan a aspectos tan banales como nuestro color de ojos. Algunos son especialmente relevantes porque pueden afectar a nuestra salud o incluso a nuestra respuesta a ciertos tratamientos. Veamos algunos ejemplos:
G1691A es un SNP ubicado en el gen F5 y que consiste en un cambio de guanina (G) a adenina (A). Este pequeño cambio provoca que el Factor V de la coagulación de la sangre sea resistente a su desactivación, lo que aumenta el riesgo de trombosis en pacientes que lo presentan.
C677T es un SNP que encontramos en el gen MTHFR y que consiste en un cambio de citosina (C) a timina (T). Las personas con este SNP producen un enzima metilentetrahidrofolato reductasa mucho menos eficiente, lo que resulta en una falta de folato (vitamina B9 activa) y un aumento en el riesgo de enfermedades cardiovasculares y defectos en el desarrollo fetal.
Algunos SNPs, como los que se han visto en los genes CYP2C9 y VKORC1 pueden afectar a la velocidad a la que una persona metaboliza fármacos como el anticoagulante warfarina. Cuando las personas con estos SNPs reciben la dosis estándar de warfarina, acumulan el fármaco en su cuerpo y presentan un riesgo grave de hemorragia.
¿Qué técnicas se utilizan para detectar SNPs?
Seguro que te estás preguntando cómo somos capaces de encontrar un cambio de una sola letra en un “manual” de más de 3.000 millones de pares de nucleótidos. Pues bueno, por suerte disponemos de diferentes herramientas que hacen el proceso mucho más sencillo que hace años.
Secuenciación de Sanger
Es el “Golden Standard” para el análisis de SNPs específicos de forma precisa. Es una técnica interesante de cara a confirmar ciertos SNPs, pero es costosa y lenta si queremos analizar miles de variantes de forma simultánea.
Secuenciación de Nueva Generación (NGS)
La joya de la corona. Las técnicas de NGS nos permiten analizar todo el genoma completo (o el exoma, dependiendo del caso) de una persona e identificar todos sus SNPs de forma rápida. Sin embargo, la tasa de error es algo más alta que en la secuenciación de Sanger y la cantidad de datos generados es, en ocasiones, difícil de manejar a nivel informático.
Arrays de genotipado y chips de SNPs
En casos en los que se busca un SNP concreto, muchas veces se utilizan los chips de SNPs. Estos “chips” son una especie de placas de cristal con millones de “anzuelos” capaces de capturar específicamente variantes ya conocidas. La parte positiva es que son muchísimo más baratos y rápidos que otras técnicas como la NGS o la secuenciación de Sanger. Sin embargo, son ciegos ante lo desconocido, es decir, no pueden detectar variantes que no estén contempladas en el diseño del chip.
Análisis: ¿Cómo interpretar un informe de SNPs?
Cuando un profesional recibe un informe en el que se indica que un paciente presenta un SNP concreto, es muy importante que evalúe ciertos aspectos relevantes alrededor de este SNP. En primer lugar, es interesante comprobar si este SNP está presente en una copia del gen o en las dos que tiene el paciente. Algunos SNPs sólo tienen relevancia clínica cuando se presentan en las dos copias (homocigosis).
Por otro lado, siempre es recomendable revisar la evidencia clínica. No todos los SNPs son peligrosos y es importante contrastar lo obtenido en el informe con datos de la comunidad científica de bases de datos como ClinVar o PharmKGB.
También es muy importante tener en cuenta el contexto del paciente. Muchos SNPs se relacionan con un riesgo elevado a desarrollar ciertas condiciones médicas. Sin embargo, no todo está escrito en nuestros genes y existen otros múltiples factores que pueden tanto contribuir al desarrollo de la enfermedad como proteger al paciente de ella. Por ejemplo, el informe puede decirnos que un paciente tiene un SNP de riesgo para el colesterol alto, pero si el paciente sigue una dieta saludable y hace deporte, este riesgo puede no llegar a manifestarse.
¿Cuáles son las limitaciones del análisis de SNPs?
El análisis de SNPs, especialmente en cuanto a arrays de SNPs se refiere, es una herramienta común en genómica por su bajo costo y eficiencia. Sin embargo, analizar únicamente los SNPs de una persona puede no ser suficiente para llegar a un diagnóstico.
En primer lugar, cuando utilizamos arrays de SNPs podemos detectar muchísimos SNPs patogénicos ya descritos, pero obviamos toda la variación que no está “cargada” en el diseño del chip. Si el origen de la enfermedad se encuentra en un SNP muy raro o no cargado en el chip, no será detectado.
En segundo lugar, analizar únicamente los SNPs obvia todo el resto de variaciones genéticas más grandes, como las CNV (variaciones en el número de copias), inserciones y deleciones de más de un nucleótido.Además, existe un importante sesgo de selección en los estudios de asociación del genoma completo (GWAS), que incluyen principalmente datos de personas que viven en ciudades europeas y del norte de América. Dado que estos estudios han aportado muchísima de la información disponible hasta el momento sobre SNP, los estudios de SNPs suelen ser menos eficaces en personas de ascendencia africana, asiática o sudamericana.

