INTRODUCCIÓN
Los avances en genómica y epigenómica están permitiendo la generación de nuevos test para el diagnóstico temprano, el pronóstico y la monitorización del efecto y la idoneidad de las terapias. En particular la epigenética está revolucionando la biomedicina y el diagnóstico clínico contribuyendo enormemente al desarrollo de la medicina de precisión al aportar biomarcadores con nuevas características de especial relevancia para el manejo clínico de las enfermedades.
A lo largo de este artículo manejaremos el concepto de biomarcador epigenético definido como cualquier marca epigenética o alteración de mecanismo epigenético que es estable y reproducible durante la toma y el procesamiento de la muestra biológica, ya sea fluido biológico o preparaciones de tejido fresco, congelado o fijado como muestras de tejido fijadas con formalina y embebidas es parafina (FFPE, formalin-fixed, paraffin-embedded) y que idealmente debería cumplir alguno de los siguientes requisitos: (i) define una enfermedad (diagnóstico), (ii) incluso antes de la aparición de los primeros síntomas (diagnóstico precoz); (iii) revela información sobre la historia natural de una enfermedad; (iv) predice la progresión y el desenlace de una enfermedad (pronóstico); (v) responde a la terapia (predictivo); (vi) permite el seguimiento de las respuestas a la terapia (monitorización de terapias), y (vii) permite el establecimiento de una terapia dirigida adecuada en función del diagnóstico (teragnosis) (García-Giménez, 2016a; Garcia- Gimenez, 2017). Las modificaciones epigenéticas a lo largo del genoma representan un sistema perfectamente orquestado que modula la organización de la cromatina y en último término la transcripción óptima del genoma en el tipo celular y el momento adecuado. En este sentido, la identificación de cambios aberrantes en las modificaciones o mecanismos epigenéticos se ha asociado a distintas enfermedades humanas (Toraño, 2012) y en consecuencia estas alteraciones epigenéticas proveen nuevos biomarcadores que pueden ayudar en la toma de decisiones clínicas (Hu, 2009; Balgkouranidou, 2016; Gao, 2016; Vrtačnik, 2014; Markopoulou, 2012; Heichman and Warren, 2012).
Existen varias ventajas de los biomarcadores epigenéticos en relación a otros biomarcadores. Aportan información relevante sobre la función adecuada de un gen en un tipo celular – por ejemplo sobre el estado de metilación, o la heterocromatinización de un promotor o una región reguladora – pero además pueden incorporar información sobre cómo el entorno, el estilo de vida y la alimentación de un individuo afectan, en último término, a la expresión de un gen o conjunto de genes que alteran rutas moleculares esenciales para la homeostasis celular (Relton, 2015; Andersen, 2015). Es por ello que los biomarcadores epigenéticos pueden considerarse bioarchivos que incluso pueden almacenar la información de la historia natural de una enfermedad. Otra ventaja es que por la propia naturaleza de algunos biomarcadores epigenéticos (como los microARN y algunas modificaciones postraduccionales de las histonas, PTM por sus siglas en inglés) son muy estables en fluidos como plasma, suero, orina, saliva, semen, secreciones vaginales, etc. o incluso en gota de sangre seca (Guthrie Cards) (Glinge, 2017; Park, 2009; Zubakov, 2010), pero también en preparaciones de tejidos, como por ejemplo en cultivos celulares, tejido fresco, congelado, y fijado (Patnaik, 2010; Peiró-Chova, 2013). En efecto, ya en uno de nuestros estudios demostramos que los microARN son extremadamente estables en muestras descongeladas y en tejidos parafinados (Patnaik, 2010; Peiró-Chova, 2013). En relación a la relativa estabilidad de algunas PTMs en las histonas, ello no supone una dificultad importante a la hora de su análisis, siempre que se tomen ciertas precauciones en los protocolos de extracción; por ejemplo, ciertas modificaciones específicas como la fosforilación requieren tener en cuenta ciertas precauciones metodológicas particulares en los protocolos de extracción, como añadir inhibidores de fosfatasas.
En otros trabajos también se ha demostrado que la determinación de la metilación del ADN en un panel de 22 genes estudiados no se veía alterada entre muestras de plasma EDTA conservadas a 4 ºC, -20 ºC y -80 ºC (Bulla, 2016). Por otro lado, Joo y cols., demostraron que en muestras de sangre seca (Guthrie Cards) era posible analizar la metilación del genoma mediante el ensayo Infinum Human Methylation 450 Beadchip (Joo, 2013).
Tras destacar la estabilidad de los biomarcadores epigenéticos y su presencia en distintos tipos de muestra, para su implantación a la clínica es necesario discutir sobre los métodos y procedimientos de detección y análisis. Podría pensarse que estos nuevos biomarcadores epigenéticos son fácilmente adaptables a las herramientas y los procedimientos rutinarios de un laboratorio clínico (Collinson, 2015). No obstante, existen ciertas limitaciones en relación a su implementación en un laboratorio clínico. Por mencionar algunos aspectos: en algunos casos los biomarcadores candidatos requieren de un procesado previo de la muestra antes de su análisis (por ejemplo, el tratamiento con bisulfito para el análisis de la metilación del ADN, o el aislamiento y separación de exosomas para el análisis de microARN circulantes). En algunos casos, se requiere adaptar tecnologías rutinarias a la medida de los nuevos biomarcadores, pero en otros casos se requiere adoptar tecnologías de alto rendimiento, como la secuenciación masiva (NGS), a ensayos que sean realmente asequibles, no solo en cuanto a su precio sino también al tiempo necesario para el análisis de los resultados. En relación a estos dos últimos puntos, las compañías de diagnóstico in vitro (IVD) están trabajando en el desarrollo de kits sencillos de manejar, que permitan tanto el procesamiento de la muestra como la purificación y la detección del biomarcador epigenético de interés para así acelerar el proceso de adopción de estas tecnologías por parte de los laboratorios clínicos.
MEJORAS EN LOS PROCESOS DE PURIFICACIÓN DE LOS BIOMARCADORES EPIGENÉTICOS
La biopsia líquida consiste en una nueva estrategia para identificar, de forma no invasiva y relativamente fácil, algunos componentes de la sangre y otros fluidos biológicos que pueden servir como biomarcadores de diagnóstico, como son las células tumorales circulantes (CTC), el ADN libre circulante (cell free DNA, cfDNA) y los exosomas (micro-vesículas que pueden transportar microARN, ARN no codificantes largos y ADN de doble cadena circulante). El análisis a partir de muestras de sangre periférica está tomando una gran relevancia para el diagnóstico dada la poca invasividad y facilidad de extracción que supone (Yap, 2014; Mabert, 2014; Kosaka, 2010a; Thakur, 2014). Además, también es posible aislar otro tipo de marcadores epigenéticos a partir de muestras de sangre como los microARN circulantes libres, histonas y nucleosomas.
Metilación del ADN
La extracción de ADN es una técnica habitual para múltiples aplicaciones de biología molecular, por lo que hay disponibles varios kits comerciales. Existen diferentes métodos, por lo que para su elección en clínica hay que tener en cuenta diferentes factores, como: a) la calidad, la concentración y pureza del ADN obtenido que debe ser adecuada para su posterior uso; b) la automatización y la simplicidad; y c) el precio.
Los diferentes métodos de extracción se basan en: a) extracción orgánica en la que las proteínas son precipitadas con fenol y cloroformo, y el ADN es posteriormente purificado con etanol o isopropanol; b) adsorción específica a membrana o partículas de sílice; c) reconocimiento específico del ADN por anticuerpos de unión al ADN o grupos funcionales específico; d) interacción específica entre los fosfatos cargados negativamente del ADN y las moléculas de superficie con carga positiva en el sustrato.
En relación al cfDNA, su aislamiento y cuantificación representa un reto debido a su baja cantidad y su naturaleza. Su fragmentación representa un problema adicional para la determinación del patrón de metilación cuando se procesa la muestra para su tratamiento con bisulfito. Por lo tanto, la purificación y cuantificación del cfDNA es un primer paso crítico para la obtención de resultados reales y, por ello el desarrollo de métodos estandarizados, reproducibles y que incrementen la eficiencia de los protocolos actuales es de vital importancia para la aplicación de estos métodos en la clínica (Devonshire, 2014). Actualmente existen distintos kits para la purificación de los ácidos nucleicos circulantes, aunque la mayoría se han diseñado para su uso en investigación y no para diagnóstico clínico. Algunos kits se han usado también para los estudios de metilación en cfDNA, tanto en forma de arrays como en análisis de metiloma completo ( Zhai, 2012; Legendre, 2015; Li, 2016), aunque en este último caso se requiere de pasos previos de purificación y tratamiento de la muestra. Por su parte, otros kits se han usado para preparar las librerías en los experimentos de secuenciación o para realizar experimentos de RT-qPCR [retrotranscripción (RT) y reacción en cadena de la polimerasa (PCR) cuantitativa (q)], llegando a obtenerse con alguno de ellos hasta 100 ng de cfDNA por mL de plasma, con un rendimiento suficiente de ADN en el rango de tamaños de 50-1000bp (Schmidt, 2005), que es adecuado para estudios de NGS.
Por el contrario, el ADN extraído de leucocitos de sangre periférica o sangre entera se puede obtener fácilmente y con mayor cantidad y calidad. Sin embargo, hay que tener en cuenta que los patrones de metilación identificados en leucocitos no siempre son representativos del patrón de metilación de otros tejidos, así por ejemplo puede diferir de los patrones de metilación observado en distintos tipos tumorales (Choi, 2009; Shen, 2017) y otras enfermedades como Alzheimer (Ozaki, 2017). No obstante, hay algún ejemplo en el que sí son informativos a este respecto, como el reciente trabajo publicado por Crujeiras y cols., donde encuentran varios sitios CpG con un patrón de metilación similar tanto en el tejido adiposo como en los leucocitos, estando ambos asociados con la obesidad (Crujeiras, 2017).
microARN circulantes
Los microARN pueden ser detectados en los fluidos biológicos, y dado que sus niveles aparecen alterados en situaciones patológicas, han generado un gran interés en los últimos años (García-Gimenez, 2015). Sin embargo, al igual que ocurre con el cfDNA la purificación de los microARN en fluidos biológicos suele ser poco eficiente (Kroh, 2010). Se pueden encontrar tanto como moléculas libres en la circulación, empaquetados en micropartículas (exosomas, microvesículas y cuerpos apoptóticos) (Kosaka, 2010b; Valadi, 2007; Zernecke, 2009), asociados a complejos lipoproteicos (lipoproteínas de alta densidad, high-density lipoprotein, HDL) (Vickers, 2011), como unidos a proteínas que participan en el silenciamiento de ARN para evitar su propia degradación (Arroyo, 2011). Dependiendo del protocolo que se utilice es posible aislar microARN circulantes libres, aquellos unidos a proteínas, solamente los que se encuentran asociados a microvesículas o bien los microARN totales presentes en la muestra de sangre (Moret, 2013).
Otro punto relevante a tener en cuenta cuando analizamos microARN circulantes es el tiempo en el que la muestra es procesada y el tiempo que ha sido almacenado el suero o el plasma. De hecho, es muy recomendable procesar los tubos de sangre con EDTA durante las 6 primeras horas después de su extracción (Tsui, 2002), ya que las células sanguíneas, incluyendo los eritrocitos, pueden liberar este tipo de moléculas, alterando de este modo la firma de microARN que debería ser utilizada como biomarcador (Duttagupta, 2011). No obstante, se han podido extraer microARN a partir de muestras de suero que han sido guardadas a -25ºC durante al menos 40 años (Rounge, 2015) e incluso a partir de muestras conservadas durante 2-4 años a -20ºC (Grasedieck, 2012). Por otro lado, y al igual que en el caso del cfADN, no existen kits de purificación de microARN específicamente diseñados para diagnóstico clínico, aunque sí distintos kits comerciales que permiten la purificación de microARN con un rendimiento adecuado, incluso para la construcción de librerías para la realización de smallRNA-seq mediante técnicas de NGS (Seco-Cervera, 2017). El kit PAXgene Blood RNA (Qiagen) es uno de los pocos kits aptos para IVD, cuando se utilizan los tubos PAXgene para ARN en sangre. El ARN tiene una buena calidad cuando la muestra de sangre se congela durante largos períodos de tiempo con este sistema (Debey-Pascher, 2011).
No obstante, las muestras utilizadas como fuente de microARN requieren pasar algunos controles de calidad para evitar la alta degradación del ARN o la contaminación por otros microARNs liberados por otras células o tejidos distintos de los requeridos para el diagnóstico. Por ejemplo, se sabe que la hemólisis es una fuente de variación significativa en los perfiles de microARN que puede afectar el perfil de biomarcadores propuestos. La hemólisis puede ocurrir en diferentes pasos del muestreo y procesamiento hasta que se eliminan el resto de los componentes de la sangre para separar el plasma o el suero. Es por ello que las muestras de suero / plasma deben analizarse previamente para detectar la hemólisis, bien por métodos espectrofotométricos (Kirschner, 2011), midiendo el ratio de hemólisis (Zanutto, 2014), el H-score (Appierto, 2014) o utilizando el método Harboe, que mide la concentración de hemoglobina (MacLellan, 2014), aunque el método más rápido para controlar la hemólisis es el primero. Además, un método alternativo para identificar supuestas muestras hemolisadas es usar microARNs que se sabe están enriquecidos en los eritrocitos, como miR-451, y aquellos que no están influenciados por hemólisis como miR-23a-3p (Blondal, 2013). Shah et al., compararon varios métodos para evaluar la hemólisis en muestras de plasma o suero para su posterior uso en el análisis de microARN, encontrando que la relación de miR-451a a miR-23a-3p propuesta por Blondal et al. (Blondal, 2013) resultó ser el método más sensible para detectar bajos niveles de hemólisis, y debería incluirse en el ensayo durante el análisis de microARNs como control de calidad (Shah, 2016).
Histonas y nucleosomas
El uso de las histonas como biomarcadores de enfermedad reside principalmente en dos posibilidades: bien en el análisis de sus PTM y sus variaciones en el contexto de la enfermedad, o bien en el análisis de la mera presencia de histonas en localizaciones extracelulares, principalmente el torrente sanguíneo. En este último caso, el análisis de las PTMs es a su vez una valiosa herramienta para el diagnóstico y/o pronóstico de ciertas enfermedades, como ha sido discutido anteriormente por García-Giménez (García-Gimenez, 2015) y Reddy (Reddy, 2017). La mayoría de kits y métodos de purificación de histonas son diseñados para células en cultivo, tejido, o sangre total y todos ellos requieren que pueda obtenerse una elevada densidad de células sea por homogeneización de tejidos o aislamiento de células sanguíneas. Sin embargo, la detección de histonas libres o circulantes, así como el análisis de PTMs en histonas en sangre, requiere exclusivamente del aislamiento de la fracción de plasma o suero. Escasean los métodos para purificar histonas a partir de fluidos biológicos, aunque se han utilizado algunas alternativas para concentrar esta fracción. Una de ellas es el uso de filtros de centrífuga Amicon-Ultra-15 10k para concentrar proteínas de bajo peso molecular, como las histonas, en muestras de orina (Bohmann, 2014). Más recientemente, Reddy et al., han desarrollado un método denominado Dual Acid Extraction protocol (DAE) para aumentar la eficiencia de la purificación en suero (Reddy, 2017). El método es, básicamente, el mismo que el descrito por Shechter et al. (Shechter, 2007), pero excluyendo el tampón de lisis hipotónico, que no se requiere para las muestras de suero.
MEJORA DE LOS MÉTODOS DE ANÁLISIS DE BIOMARCADORES EPIGENÉTICOS
La preparación de la muestra es uno de los pasos más críticos en la investigación epigenética y, por supuesto, se vuelve fundamental en el diagnóstico clínico. El aislamiento eficiente y de alta calidad de los ácidos nucleicos, la cromatina y las histonas para los posteriores análisis, asegurarán que se puedan obtener resultados de la más alta calidad a partir de muestras clínicas. Es por ello que los métodos de preparación de muestras para el análisis de biomarcadores epigenéticos y para su aplicación en diagnóstico clínico se están intentando mejorar continuamente.
Metilación del ADN
La conversión del ADN con bisulfito es un paso necesario en algunos de los procedimientos utilizados para análisis de metilación. El tratamiento del ADN con bisulfito convierte los residuos de citosina en uracilo, sin afectar a las citosinas metiladas. Sin embargo, este procedimiento presenta algunas limitaciones. En primer lugar, la cantidad de ADN convertido con bisulfito obtenido de algunas muestras biológicas es a menudo demasiado pequeña para permitir posteriormente el análisis de la metilación. El ADN convertido con bisulfito puede experimentar una degradación significativa durante la conversión (Tanaka and Okamoto, 2007) y durante el almacenamiento, disminuyendo la sensibilidad de los ensayos de metilación del ADN y haciendo que este no sea adecuado para un análisis posterior, si no se usa inmediatamente después del tratamiento con bisulfito (Wojdacz and Dobrovic, 2007). Además, existe riesgo de conversión incompleta, que daría lugar a errores de análisis, debido a que la conversión tan sólo se produce sobre ADN monocatenario siendo necesario asegurar la desnaturalización completa del ADN (Li and Tollefsbol, 2011). Otro factor a tener en cuenta es que el tratamiento con bisulfito no puede discriminar entre 5-metilcitosina y 5-hidroximetilcitosina, otra modificación del ADN (Huang, 2010).
Afortunadamente, en los últimos años se han comercializado varios kits que permiten mejorar la calidad de la conversión y la estabilidad del ADN tras su procesamiento con bisulfito. Holmes et al. estudiaron el rendimiento de diferentes kits para la conversión con bisulfito del ADN, evaluando a su vez diferentes bioespecímenes como FFPE, aspirados, lavados bronquiales, efusiones, plasma, suero y orina (Holmes, 2014). De especial relevancia es el hecho que las muestras de ADN tratadas con bisulfito fueron estables durante al menos 4 semanas cuando se almacenaron a una temperatura de -80 ° C a 4 ° C utilizando algunos de estos kits. Ese resultado está de acuerdo con los hallazgos previos de Dietrich et al., donde el ADN convertido con bisulfito fue estable más de 2 años a -20 °C (Dietrich, 2013). Más recientemente, Jung et al., detallaron varios métodos y protocolos para la preparación de muestras biológicas y su posterior conversión de bisulfito para varios tipos de muestras biológicas utilizadas en un entorno clínico (Jung, 2017).
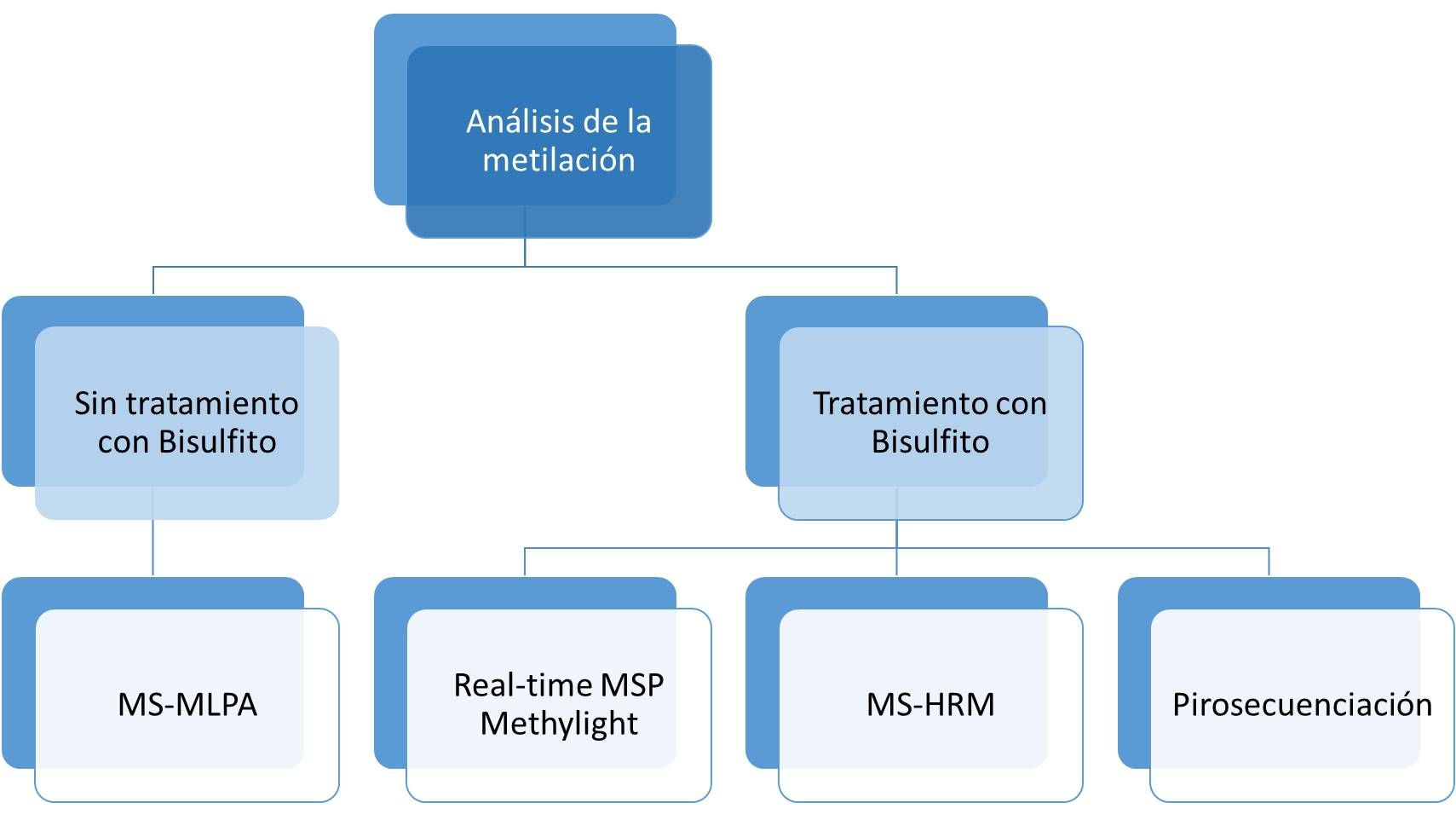
Los ensayos para el análisis de la metilación del ADN se han basado en el uso de enzimas de restricción o bien en el uso de técnicas que requieren del tratamiento con bisulfito (Figura 1). Este tipo de ensayos para el análisis de la metilación del ADN se han utilizado en la rutina de laboratorio clínico para el diagnóstico de algunos síndromes raros. Algunas de estas técnicas se basan en el uso de enzimas de restricción sensibles a la metilación. Sin embargo, uno de los principales problemas de dichas técnicas es que los fragmentos de restricción generados se analizan mediante métodos semicuantitativos, como el Southern-blot o PCR seguida de una electroforesis, y por lo tanto los resultados e interpretación van a depender de la inspección visual del gel, complicando la automatización. Esta desventaja ha propiciado el desarrollo de otras alternativas como los ensayos de amplificación de múltiples sondas dependientes de ligasa y específicas de metilación (MS-MLPA por sus siglas en inglés), las pruebas basadas en la conversión química de ADN usando tratamiento con bisulfito, las reacciones basadas en qPCR, las pruebas que analizan el perfil de fusión de alta resolución sensible a la metilación (MS-HRM por sus siglas en inglés) y, finalmente, la pirosecuenciación. La MS-HRM y la pirosecuenciación son las técnicas que tienen un mejor rendimiento, reproducibilidad y sensibilidad (Quillien, 2012; Migheli, 2013) y por ello son las que más aceptación están teniendo en el entorno clínico. La descripción detallada de cada una de estas técnicas se presenta en el libro Epigenetic Biomarkers and Diagnostics, editado por José Luis García-Giménez (García-Giménez, 2016b) y también en el libro Enfermedades de Impronta. Guías de buena práctica clínica, editado por Guiomar Pérez y Pablo Lapunzina (Pérez de Nanclares, 2016).
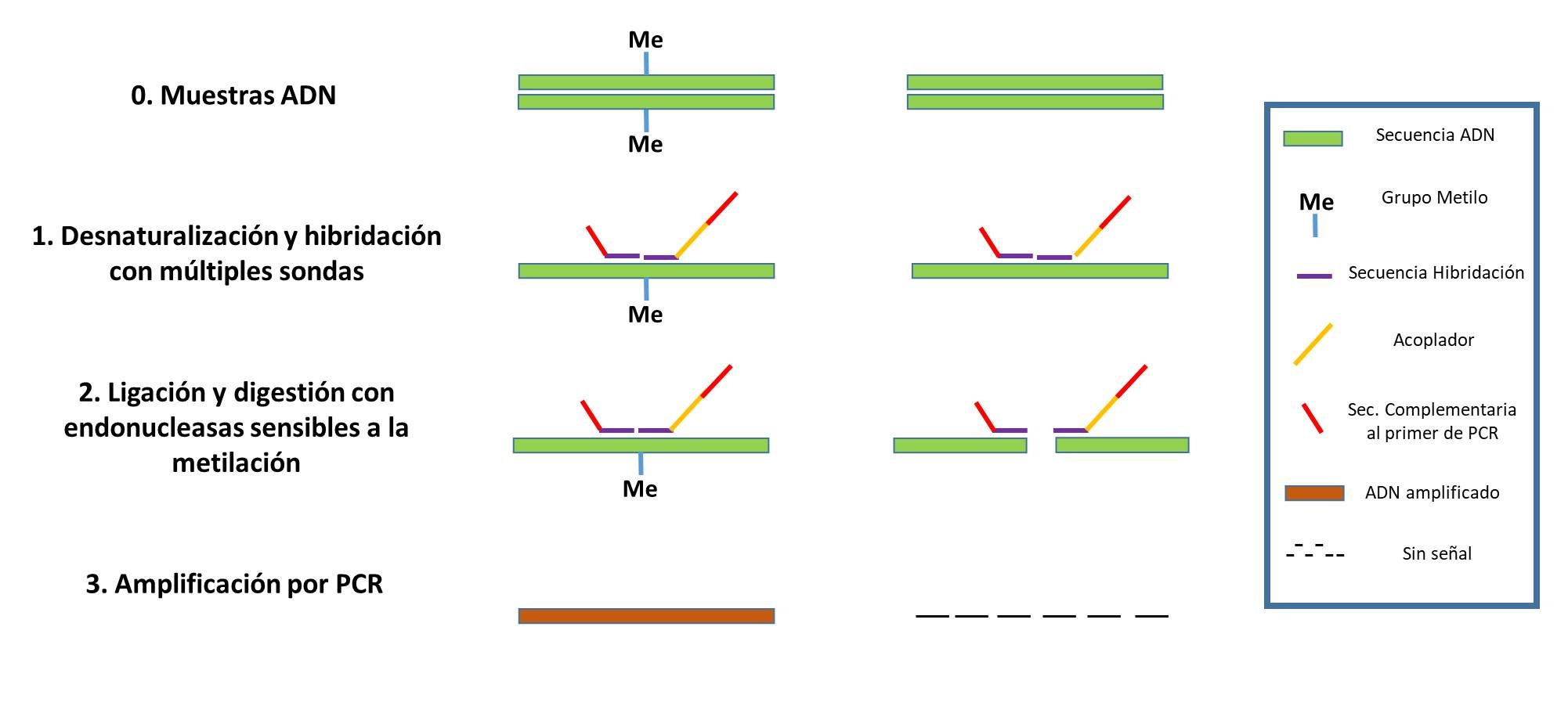
La técnica de MS-MLPA (Figura 2) es un método semicuantitativo que permite evaluar el perfil de metilación. Consiste en una variante de la técnica MLPA en la que la detección del número de copias se combina con el uso de una enzima de restricción sensible a la metilación. Los productos resultantes generalmente se analizan en un secuenciador automático (electroforesis capilar) (Nygren, 2005), y la dosis génica se calcula comparando las señales de las muestras no digeridas con los controles no digeridos. La comparación de las señales obtenidas permite determinar el nivel de metilación de las sondas que contienen el sitio de restricción HhaI. El análisis de los datos en bruto se puede llevar a cabo utilizando diferentes programas, algunos de ellos libres como el Coffalyser®, (MLPA-Holland, Países Bajos) o el desarrollado por el INGEMM (Instituto de Genética Médica y Molecular, Hospital Universitario La Paz de Madrid) (Romanelli, 2011).
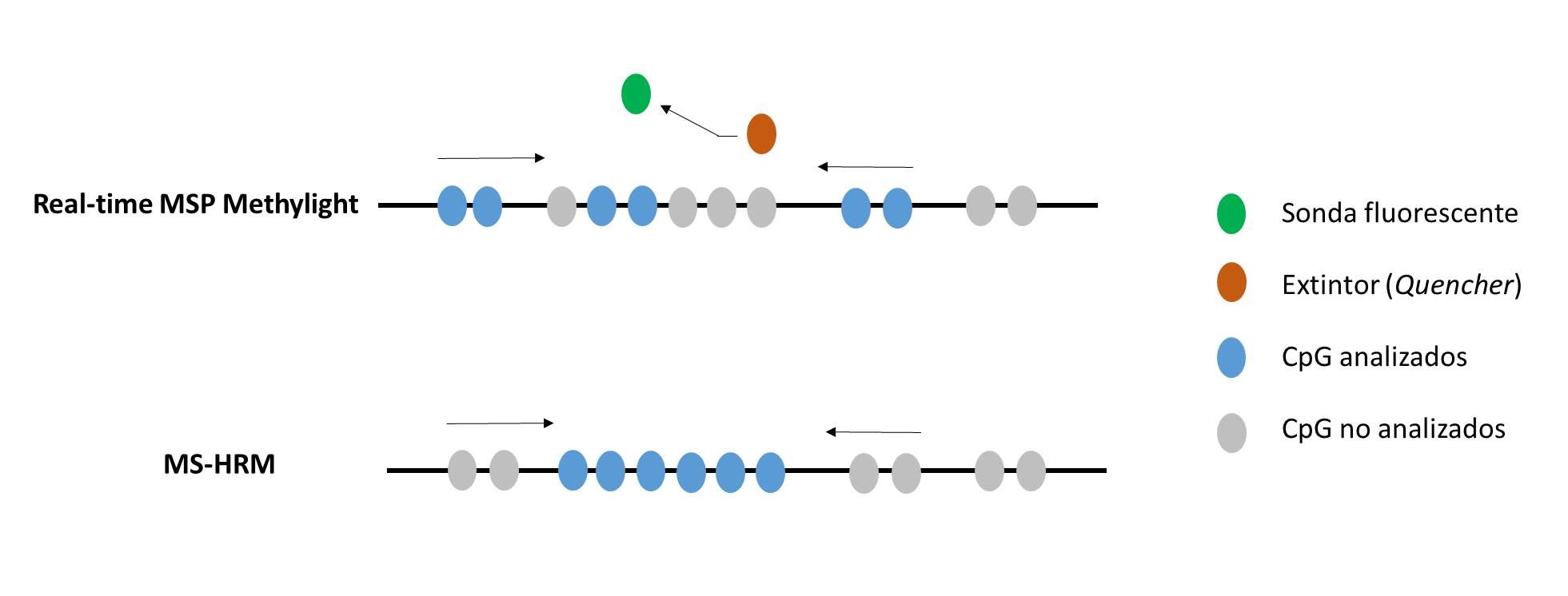
La técnica MethyLight o qRT-MS-PCR (Figura 3) fue desarrollada por primera vez por Eads et al. en 1999 (Eads, 2000), se basa en la tecnología estándar de PCR específica de metilación, en el que el ADN modificado por tratamiento con bisulfito es amplificado usando cebadores específicos y sondas fluorescentes TaqManTM. Dado que el poder discriminatorio del método se asienta en una qPCR, se puede lograr una alta sensibilidad (una resolución de hasta un nucleótido) manteniendo una alta especificidad (Zhao, 2015). Además, MethyLight se ha utilizado para analizar simultáneamente la metilación del ADN obtenido de FFPE (Liu, 2011; Olkhov-Mitsel, 2012), de cfDNA (Muller, 2003), así como de sedimentos de orina (Reinert, 2012).
En 2010, He et al., desarrollaron la técnica MethyLight multiplex para analizar simultáneamente la metilación de múltiples genes en la misma muestra de ADN (He, 2010). El uso del elemento ALU, como referencia y control interno, fue introducido en el procedimiento por el grupo de Bapat. Esta tecnología supera en precisión, reproducibilidad y en precio a la singleplex MethyLight, pues también reduce la cantidad de ADN de partida necesario para el análisis de múltiples genes. Esta técnica se ha aplicado con éxito para medir la metilación del ADN en tejido FFPE, tejido congelado fresco, biopsia de tejido y muestras de orina (Olkhov-Mitsel, 2014).
El análisis de temperatura de fusión de alta resolución (HRM) es una técnica que se basa en las características de disociación del ADN bicatenario durante el calentamiento. El MS-HRM (HRM sensible a metilación) mide la metilación del ADN sobre la base de que el ADN metilado y no metilado, al ser tratados con bisulfito, producen secuencias diferentes que generan productos de PCR con perfiles de temperatura de fusión marcadamente diferentes (Figura 3). La HRM puede estimar la proporción de metilación dentro de una muestra cuando se usan patrones de ADN convertidos en bisulfito (Wojdacz, 2012), pero la fiabilidad y sensibilidad de este método dependen de la captura de múltiples datos en cada ejecución y de una alta precisión en las evaluaciones pocillo a pocillo. Wojdacz y Dobrovic han optimizado protocolos de PCR para analizar la metilación del ADN por MS-HRM, favoreciendo la amplificación de la secuencia metilada (Wojdacz, 2008; Wojdacz, 2009; Wojdacz and Dobrovic, 2009). Considerando que el análisis de HRM requiere productos de PCR cortos, esta técnica puede ser aplicada al estudio de la metilación de muestras en las que el ADN suele estar de por sí degradado, como son las muestras de tejido FFPE. La técnica MS-HRM es adecuada para laboratorios clínicos, ya que es una herramienta rápida y económicamente viable (Worm, 2001). Aunque no es específica de locus, tiene amplias aplicaciones prácticas en el diagnóstico de enfermedades de impronta genética como PWS, AS (White, 2007) y BWS (Romanelli, 2011; Tenorio, 2016).
La pirosecuenciación se basa en el principio conocido como «secuenciación por síntesis», en el cual los nucleótidos se adicionan secuencialmente y su incorporación se detecta, en tiempo real, gracias a la señal luminosa que produce la liberación de pirofosfato inorgánico (Tost and Gut, 2007a; Tost and Gut, 2007b) Este pirofosfato es transformado por la ATP sulfurilasa en ATP, que se acopla a la luciferina, formando un complejo que será utilizado por una luciferasa para producir oxiluciferina y la señal luminosa. Dado que el orden de adición de nucleótidos es conocido se puede determinar la secuencia de ADN molde.
La pirosecuenciación tiene una amplia gama de aplicaciones que incluyen la detección de polimorfismos de un único nucléotido (siglas en ingés, SNPs,) inserción/deleciones, número de copias de genes y análisis de la metilación del ADN (Zhao, 2015). Algunas de las plataformas de pirosecuenciación que se encuentran en el mercado son PyroMark Q24, PyroMark Q24 Advanced o PyroMark Q96 ID de Qiagen, o el Secuenciador 454 de Roche, que es una plataforma de secuenciación de nueva generación y de alto rendimiento (Florea, 2015). Es una técnica fiable, rápida y de alto rendimiento que puede analizar simultáneamente, en aproximadamente 4 h, hasta 96 muestras de ADN modificadas con bisulfito. Esta técnica ha sido utilizada previamente en el análisis de la metilación del ADN en FFPE, sangre y cfDNA (Romanelli, 2011; Florea, 2015; Tang, 2016; Zmetakova, 2013).
microARN
El análisis por RT-qPCR se ha convertido en una de las herramientas más relevantes en diagnóstico molecular en los laboratorios clínicos. Es por ello que esta es una de las herramientas más habituales para analizar microARN en el diagnóstico clínico rutinario, ya que proporciona alta sensibilidad, es rentable, permite el análisis simultáneo de múltiples secuencias y una amplia variedad de aplicaciones.
La qPCR requiere de un paso de retrotranscripción previo, y para ello podemos encontrar distintas opciones comerciales. Una opción es crear una cola poli A mediante la incubación de los microARN con una poliadenilasa. Posteriormente, se emplea un cebador poli-T para iniciar la RT. Este método permite realizar la qPCR con un cebador específico complementario al microARN de interés y un cebador universal. Este sistema es en el que se basan Sigma, Qiagen, Agilent, TaqMan Advanced miRNA Assays (ThermoFisher) y Exiqon, aunque este último incorpora una tecnología propia LNA (Locked Nucleic Acid), que le confiere una conformación que hibrida con mayor selectividad con su base complementaria. Otra de las opciones es el uso de cebadores en bucle (stem loops) parcialmente complementario al extremo 3’ de un microARN usado por TaqMan™ MicroRNA Assays (ThermoFisher). O mediante la ligación de un oligonucleótido universal al extremo 3’ del microARN para una posterior RT empleando un cebador universal complementario al adaptador incorporado (miRStarTM miRNA First-Strand cDNA Synthesis Kit, Arraystar).
Sin embargo, a la hora de escoger el método apropiado es necesario conocer la vía de maduración de microARN, ya que estos métodos han de ser capaces de discriminar entre los precursores del microARN (pri- y pre-microARN) y del microARN maduro. Dado que la cantidad de muestras clínicas a menudo son escasas y solo se puede extraer una cantidad muy limitada de microARN, se requieren amplificaciones del material en muchos casos, tanto para la RT-PCR como para la NGS tras la construcción de las librerías.
Gran parte de los errores observados en los resultados de RT-PCR pueden ser debidos a errores en la incorporación de las bases por parte de la polimerasa durante la fase de amplificación, pero también se pueden deber a posibles errores en la ligación o hibridación de los cebadores.
Otra técnica para la detección de microARNs es la de micromatrices basada en hibridación. Esta tecnología se ha utilizado ampliamente para la detección de ARN mensajero y ADN. Pero la principal diferencia con ellos es el tamaño de los microARNs, y la posibilidad de que tanto los microARNs maduros como los pre-microARNs puedan hibridarse con las mismas sondas, lo que lleva a bajas especificidades y sensibilidades. Por último, podemos nombrar la NGS, que aporta una gran versatilidad en cuanto a la capacidad de identificar microARNs desconocidos previamente al estudio (Garcia-Gimenez, 2017).
El uso de microarrays o Small RNA-seq para el análisis de microARNs requiere de mucho tiempo y, generalmente resulta costoso, por lo que son herramientas muy útiles para la investigación biomédica cuando se exploran los mecanismos implicados en la enfermedad, pero de utilidad limitada para el análisis de uno o pocos microARNs como biomarcadores, aunque bien es cierto que en los próximos años la simplificación de los protocolos y del análisis bioinformático de los datos obtenidos a partir de estas técnicas permitirá que los laboratorios clínicos las adopten para el análisis de biomarcadores epigenéticos en el diagnóstico clínico. Sin embargo, los equipos de qPCR parecen ser los que mayor implementación clínica inmediata presentan en la actualidad. De hecho, muchos ensayos IVD basados en RT-qPCR ya han sido aprobados por la Agencia para el control de Medicamentos y alimentos de los Estados Unidos (siglas en inglés FDA). Un inconveniente de este tipo de técnicas para la cuantificación relativa de expresión de uno o varios microARNs es que requiere de varios controles (endógenos de referencia, de hemólisis en muestras de sangre…) para garantizar la calidad de los ensayos de diagnóstico de microARNs. La aparición de nuevos kits de IVD basados en la detección de microARNs utilizando esta tecnología facilitará su incorporación a los laboratorios clínicos (Garcia-Gimenez, 2017).
Por otro lado, los ensayos de RT-qPCR típicos requieren genes de referencia para normalizar los valores obtenidos para el gen diana, en este caso microARN. Varios autores han intentado encontrar los microARNs que pudieran servir como controles endógenos en el análisis de microARNs en biofluidos. En la Tabla 1 se muestran algunos ejemplos de microARNs usados como controles endógenos de referencia para la normalización de datos. En un futuro cercano, se espera que el desarrollo de nuevas técnicas que permitan la cuantificación absoluta, como la PCR digital (ddPCR), solucione este tipo de problemas y revolucione la aplicación de microARN como biomarcadores de enfermedades.
Tabla 1. microARNs propuestos como controles endógenos en ensayos de expresión relativa en RT-qPCR para detectar microARNs circulantes en biopsia líquida.
|
microARNs de referencia |
Biofluido |
Estudio en el que se ha usado el microARN(s) como gen de referencia |
Referencia |
| miR-146b-5p, miR-142-3p y miR-24 | Suero | Donantes sanos | (Marabita, 2016) |
| miR-17, miR-126, miR-484 | Suero | Infección por Hepatitis C | (Marabita, 2016) |
| miR-26a, miR-221 y miR-22 | Suero | Infección por virus de la Hepatitis B (HBV) | (Zhu, 2012) |
| miR-16 y miR-93 | Suero | Cáncer Gástrico | (Song, 2012) |
| miR-16 | Plasma | Ataxia de Friedreich | (Seco-Cervera, 2017) |
| Suero | Fibromialgia | (Masotti, 2016) | |
| miR-25-3p | Suero | Osteoporosis | (Chen, 2016) |
| miR-106b-5p, miR-93-5p y miR-25-3p | Suero | Cáncer Colorectal | (Niu, 2016) |
| miR-320a y miR-486-5p | Plasma | Sepsis | (Caserta, 2016) |
Histonas
El análisis de variantes de histonas y de PTMs en histonas se ha realizado tradicionalmente en investigación mediante una combinación de técnicas de biología molecular que mayormente incluyen el Western blot, la inmunoprecipitación de cromatina (ChIP, por sus siglas en inglés), la espectrometría de masas y los ensayos tipo ELISA. Generalmente, la combinación de diferentes técnicas proporciona la información más relevante sobre el código específico de variantes y modificaciones de histonas en un contexto celular concreto. No obstante, estas técnicas raramente se usan en la rutina clínica o tienen un escaso potencial diagnóstico/pronóstico. Debe considerarse que todos los métodos de purificación se dirigen al aislamiento de muestras de histonas o nucleosomas con propósito analítico, y se usan principalmente en investigación. Dichas técnicas están todavía lejos de producir muestras adecuadas para análisis de alto rendimiento, y una rápida aplicación a la rutina clínica. Sin embargo, se están produciendo avances en otras direcciones, principalmente aprovechando las potentes posibilidades de la tecnología de espectrometría de masas, como discutiremos más adelante.
La medida directa de las histonas en muestras de plasma, mediante inmunoensayos, es la manera estándar de detectar histonas circulantes, aunque empiezan a destacar nuevos métodos basados en la espectrometría de masas, como discutiremos en breve. En cuanto al análisis de ciertas PTM como biomarcadores de enfermedad, encontramos un ejemplo en el uso de la histona H3 citrulinada como biomarcador para enfermedades autoinmunes, como el lupus sistémico eritematoso o la artritis reumatoide (Frede, 2016). Otro ejemplo sería el uso de las histonas H3 y H4 acetiladas para monitorizar tratamientos en cáncer basados en inhibidores de desacetilasas de histonas (HDACi por sus siglas en inglés) (Rigby, 2012). Dado que otros modificadores de histonas, como las metiltransferasas y desmetilasas, están siendo incorporados en ensayos clínicos para terapia en cáncer (Morera, 2016), se precisan métodos para evaluar la eficiencia de dichos tratamientos. En este sentido, las limitaciones técnicas de los kits ELISA, o su baja reproducibilidad, son los principales cuellos de botella para otorgarles un valor de confianza como biomarcadores frente a los inmunoensayos. Por lo tanto, aunque la rutina clínica incluye varios kits ELISA para el diagnóstico diferencial de desórdenes inmunológicos que producen liberación de proteínas nucleares al torrente sanguíneo, se están desarrollando nuevos métodos que ostentan una especificidad mayor. Es el caso de las herramientas proteómicas de espectrometría de masas, que están siendo cada vez más utilizadas en las instalaciones clínicas, y cuya expansión se prevé para los próximos años (Vogeser y Seger, 2016). Esto se debe principalmente al hecho de que pueden reducir costes al reemplazar las técnicas basadas en inmunoensayos, que son más caras (Hetu, 2012). Estos métodos analíticos incrementarán su aplicación en el ámbito del laboratorio diagnóstico cuando se optimice el procesamiento de determinados bioespecímenes que por su complejidad son más difíciles de ser usados para el análisis de biomarcadores, como por ejemplo ocurre en los tejidos FFPE. Un trabajo reciente de Noberini et al., muestra que el análisis de PTM de histonas puede realizarse en tejidos FFPE, con una eficiencia similar a la obtenida mediante el análisis de muestras congeladas de tejido (Noberini, 2016a). En este contexto, las modificaciones de histonas podrían usarse como biomarcadores para el cáncer de mama, con los beneficios de usar muestras biológicas almacenadas a largo plazo en los archivos de anatomía patológica de los hospitales (Noberini, 2016b). El potencial de la espectrometría de masas puede aplicarse también a biopsias líquidas como fuente de histonas, como hemos mencionado antes; y están apareciendo nuevos métodos para mejorar la eficiencia del aislamiento de histonas a partir de suero, con vistas al análisis del perfil de sus PTMs en combinación con técnicas de espectrometría de masas (Reddy, 2017). Un buen ejemplo de la monitorización de tratamientos mediante este tipo de análisis lo encontramos en la evaluación de cambios en dicho perfil de modificaciones en células de leucemia sensibles a decitabina (Zhang, 2016). Otro ejemplo interesante es el uso que nuestro grupo ha hecho de la espectrometría de masas dirigida para detectar histonas circulantes (H3 y H2B) en muestras de plasma sin procesar, para utilizar estas histonas como biomarcadores para la predicción de un pronóstico fatal en pacientes de shock séptico (García-Giménez, 2017), constatándose su carácter de herramienta prometedora para ayudar a la toma de decisiones clínicas en el manejo de la sepsis, el cual todavía presenta numerosas dificultados en Unidades de Cuidados Intensivos (UCI) en todo el mundo (Reinhart, 2017). No obstante, los métodos basados en espectrometría de masas, aunque están bien establecidos en el ámbito de la investigación, todavía deben ajustarse rigurosamente y demostrar su potencial, para la mayoría de los usos clínicos que se describen en este artículo (Vogeser and Seger, 2016).
ANÁLISIS DE BIOMARCADORES EPIGENÉTICOS DISPONIBLES EN EL MERCADO
La continua investigación en mecanismos epigenéticos que contribuyen a las enfermedades está aportando un gran número de posibles biomarcadores epigenéticos (Ferlin and Foresta, 2014; van Dijk, 2015; Bakulski, 2016; García-Giménez, 2016b) para su potencial uso clínico. Sin embargo, el número de biomarcadores que han sido implementados en la rutina clínica es todavía escaso. A continuación, revisaremos algunos ejemplos concretos de kits o métodos que, centrándose en explotar las propiedades de este tipo de biomarcadores, han llegado a comercializarse o utilizarse en la rutina clínica. En la Tabla 2 se resumen las principales características de los ensayos que se utilizan actualmente en clínica y que utilizan biomarcadores epigenéticos.
Tabla 2. Ensayos epigenéticos basados en el análisis de la metilación del ADN y de microARNs.
| Ensayo/Compañía | Bioespecimen | Descripción | Sn/Sp | Referencia |
| Epi proColon® 2.0 CE / Epigenomics Inc | Sangre periférica | Metilación del gen de septina9 en muestras de sangre periférica. | Sn 75-81%
Sp 96-99% |
(Lamb and Dhillon, 2017) |
| Cologuard® / Exact Sciences | Sangre periférica | Niveles de metilación de los genes N-Myc 4 downstream regulado (NDRG4) y la proteína morfogénica ósea 3 (BMP3). | Sn 92,3%
Sp 86,6% |
(van Lanschot, 2017). |
| EPICUP™/ Ferrer | tejido fresco congelado/ FFPE | Compara el perfil de metilación (450K CpGs) específico del tumor, asignando un origen primario a la muestra de tumor desconocido. | Sn 97,7%
Sp 99,6% |
(Moran, 2016) |
| ThyraMIRTM/ Interpace® Diagnostic Inc | Aspirados | Determinación de 10 microARN (miR-29b-1–5p, miR-31–5p, miR-138–1-3p, miR-139–5p, miR-146b-5p, miR-155, miR-204–5p, miR-222–3p, miR-375, and miR-551b-3p) para el diagnóstico del cáncer de tiroides. | Sn 89%
Sp 85% |
(Kitano, 2012). |
| Rosetta GX Cancer Origin/ Rosetta Genomics | FFPE | Análisis del perfil de expresión de 64 microARNs analizados mediante plataforma de microArrays de Agilent y su comparación con el perfil de microARNs de 42 tumores distintos para determinar el origen del tumor. | Precisión del 85% | (Meiri, 2012). |
| Rosetta GX MI-lung™/ Rosseta Genomics Inc. | FFPE, biopsia con aguja fina, raspado bronquial y lavado bronquial | Análisis de una firma de 8 miARN mediante el uso de un análisis RT-qPCR para diferenciar el cáncer de pulmón de células pequeñas (CPCP), el cáncer de pulmón de células no pequeñas (CPCNP) y el carcinoma escamoso de pulmón no microcítico (CPCNP) no escamoso. | Sn 93,7%
Sp 90% |
(Gilad, 2012) |
| Rosetta GX MI-KidneyTM/ Rosetta Genomics Inc. | FFPE | Análisis de una firma de 24 miARNs que permite diferenciar los 4 principales tipos histológicos de tumores renales primarios; oncocitoma benigno y los 3 subtipos más comunes de carcinomas de células renales (células claras, papilares y cromófobos). | Precisión del 95% | (Spector, 2013). |
Kits IVD basados en biomarcadores epigenéticos aprobados por la FDA
Epi proColon® 2.0
Epi proColon® 2.0 CE (Epigenomics AG, Alemania) es una prueba que utiliza la técnica de qPCR para detectar el nivel de metilación del gen de septina 9 en muestras de sangre periférica. Está diseñado para la detección temprana del cáncer colorrectal (CRC). Sin embargo, un resultado positivo obtenido en la prueba debe verificarse siempre mediante otros procedimientos de diagnóstico invasivos, como la colonoscopia o sigmoidoscopia (Lamb and Dhillon, 2017). En todos los estudios, esta prueba discriminó entre pacientes con CRC y controles sanos con una sensibilidad del 75-81% y con una especificidad para el CRC versus individuos sanos del 96-99%. Dos de los estudios de casos control, también compararon el rendimiento de EpiproColon® 2.0 CE con el de la prueba inmunoquímica fecal (FIT) (Jin, 2015) o la prueba del antígeno carcinoembrionario (CEA) en sangre periférica (que no es recomendable para cribado en el CRC), y con la prueba de sangre oculta en heces (gFOBT) (Toth, 2012).
En los distintos estudios clínicos realizados la sensibilidad mostrada por la prueba varía dependiendo de la etapa de progresión del CRC. De hecho, la sensibilidad fue menor para los estadios I que para los estadios II, III o IV. Las directrices en Europa en el screening de CRC (European Colorectal Cancer Screening Guidelines Working, 2013) y en Asia-Pacífico (Sung, 2015) no incluyen todavía EpiproColon 2.0 CE, y recomiendan el uso de gFOBT o FIT cuantitativo como screening no invasivo. La colonoscopia se considera todavía el método estándar para detectar el cáncer colorrectal y aún no se recomiendan las nuevas tecnologías de detección.
Test Cologuard® basado en el análisis de ADN en heces
Las ventajas del kit Cologuard® (Cologuard®, Exact Sciences, Madison, WI, EE. UU.) estriban en la simplicidad de su uso y la capacidad de detección, con una sensibilidad del 92,3% y una especificidad del 86,6% (MDXHealth, 2014). Debido a que la exfoliación celular y la liberación del ADN en las heces se produce de forma continua, es posible detectar marcadores moleculares derivados de las células neoplásicas en el colon, lo que hace que la detección de CRC sea más precisa (Quintero, 2012; Morikawa, 2005).
Cologuard® consiste en varios abordajes de análisis que consisten en la determinación de la metilación específica del ADN y también la detección de hemoglobina en las heces (van Lanschot, 2017). Esta prueba mide específicamente los niveles de metilación de los genes N-Myc 4 downstream regulado (NDRG4) y la proteína morfogénica ósea 3 (BMP3).
Además, Cologuard® detecta la posible presencia de hasta 7 mutaciones en el exón 2 del gen KRAS (Imperiale, 2014), y los analiza a través de un algoritmo que proporciona el resultado final. Recientemente, la FDA aprobó el ensayo de metilación de NDRG4 y BMP3 como prueba de detección de CRC, puesto que ha demostrado una elevada sensibilidad y especificidad, pudiendo ser usado como una alternativa fiable a la colonoscopia (van Lanschot, 2017).
Test de diagnóstico epigenético en los laboratorios de diagnóstico genético
Existen otras pruebas comerciales basadas en el análisis de la impronta genómica que están disponibles para los laboratorios clínicos. En particular, existen pruebas para el síndrome de Prader-Willi (PWS/AS), síndrome de Angelman y síndrome de Beckwith-Wiedemann (BWS). Existen kits comerciales para detectar el estado de metilación de loci implicados en varios síndromes como PWS/AS, BWS/Silver Russell, osteodistrofia hereditaria de Albright, pseudohipoparatiroidismo, disomía uniparental de la región 6q24, 7p12, 7q32 y 14q32, diabetes mellitus neonatal transitoria, gliomas y retinoblastoma. Estos kits son comercializados por diferentes compañías como MRC-Holland (Amsterdam, Países Bajos), Prevention Genetics Inc. (Marshfield, Wisconsin, EE. UU.), Premier Biosoft (Palo Alto, California, EE. UU.) y Eurofins Biomnis (Dublín, Irlanda), entre otros.
Otra aplicación que está captando el interés de los laboratorios de diagnóstico clínico es la prueba prenatal no invasiva (NIPT), también conocida como ADN prenatal libre (pcfDNA), para detectar ciertas anormalidades cromosómicas en un feto. Durante este análisis, el ADN prenatal libre se extrae de una muestra de sangre materna para detectar la probabilidad de que el feto pueda tener alguna alteración cromosómica, como en el síndrome de Down, síndrome de Patau y el síndrome de Edwards. Este tipo de análisis también puede proporcionar información sobre el sexo del feto y el grupo sanguíneo, junto con el Rh. En relación a este tipo de análisis NIPT, recientemente se están usando regiones metiladas para diferenciar el cfDNA materno del fetal (Lim, 2014a; Lim, 2014b; Lee, 2013); por ejemplo, evaluando la metilación de los genes HLCS, RASSF1A (Lim, 2014a; Lim, 2014b) y SERPIN5B5 (Lee, 2013).
En general, la mayoría de las técnicas utilizadas en los laboratorios clínicos para diagnosticar estos trastornos se basan en enzimas de restricción sensibles a la metilación y en el tratamiento químico del ADN con bisulfito, para PCR específica de metilación.
En cambio, EPICUP™ está basado en la tecnología de micromatrices e identifica el perfil de metilación específico del tumor, asignando un origen primario a la muestra de tumor desconocido a partir de tejido fresco congelado e incluso FFPE (Moran, 2016), y que ofrece Ferrer Hospitales como servicio de análisis bajo la certificación CLIA (Clinical Laboratory Improvement Amendments), que asegura la fiabilidad, la exactitud y la reproducibilidad de un ensayo en un laboratorio clínico (Kaul, 2017). Un estudio realizado con 10.481 muestras tumorales de origen conocido indicó que la especificidad de EPICUP para determinar el tipo de tumor era del 99,6% y su sensibilidad del 97,7%.
Por otro lado, existen algunos test IVD basados en microARNs con certificación CLIA, por citar algunos ejemplos comercializados: ThyGenX® Oncogene Panel y ThyraMIRTM, desarrollados por Interpace® Diagnostic Inc, (Parsippany, NJ, USA), y el test miRView desarrollado por Rosetta® Genomics (Rehovot, Israel). El primero se desarrolló para el diagnóstico del cáncer de tiroides (Ferraz, 2011; Giordano, 2014), a partir de aspirados de punciones (Kitano, 2012) analizando la expresión de un panel de 10 microARN (miR-29b-1–5p, miR-31–5p, miR-138–1-3p, miR-139–5p, miR-146b-5p, miR-155, miR-204–5p, miR-222–3p, miR-375, and miR-551b-3p) implementados en un algoritmo propio que permite ofrecer un diagnóstico. En un estudio prospectivo realizado en pacientes con nódulos tiroideos con una citología indeterminada, la realización de las pruebas combinadas con ThyGenX® y ThyraMIR™ ofreció resultados con una buena sensibilidad del 89% (95% intervalo de confianza [CI], 73–97%) y una especificidad del 85% (95% CI, 75–92%) (Kitano, 2012).
Existen distintos test desarrollados por Rosetta Genomics, el Rosetta GX Cancer Origin, por ejemplo es un ensayo que analiza un perfil de microARNs para el diagnóstico de cánceres de origen desconocido. Por medio de una tecnología patentada se extraen 64 microARNs, a partir de muestras FFPE, y estos son analizados mediante una plataforma de micromatrices de Agilent Technologies (Rosenfeld, 2008; Meiri, 2012) usando una serie de clasificadores asignados a 42 tipos de tumores distintos que permiten proponer el origen del tumor primario. En un estudio de validación en una cohorte independiente, la precisión global del ensayo fue alta (85%), y en el 82% de las muestras analizadas fue posible establecer un único origen del tumor primario con un 90% de precisión (Meiri, 2012).
Por otra parte, el Rosetta GX MI-lung™ es un test diseñado para clasificar y diagnosticar pacientes con cáncer de pulmón. En esta prueba, se pueden utilizar muestras de FFPE, tejido fresco de biopsia, que incluyen biopsia FNA y lavado bronquial para determinar el nivel de expresión de 8 microARNs mediante un análisis RT-qPCR. Estos biomarcadores se utilizan para diferenciar el cáncer de pulmón de células pequeñas (CPCP), cáncer escamoso de pulmón no microcítico (CPNM) y del cáncer de pulmón no escamoso. La validación demostró una sensibilidad del 93.7% (95% CI, 90.8% to 95.8%) y una especificidad superior al 90%, para la identificación de los distintos subtipos histológicos del cáncer de pulmón (Gilad, 2012).
Finalmente, Rosetta GX MI-KidneyTM es un test que evalúa una firma de 24 microARN en un array de Agilent Technologies diseñado para proporcionar un diagnóstico diferencial rápido de cáncer renal a partir de muestra FFPE del tumor. Esta prueba de diagnóstico molecular in vitro permite diferenciar los 4 principales tipos histológicos de tumores renales primarios: oncocitoma benigno y los 3 subtipos más comunes de carcinomas de células renales (células claras, papilares y cromófobos) con una precisión global del 95%, evitando así cirugías innecesarias (Spector, 2013).
En cuanto a las histonas, no existen métodos de diagnóstico aprobados por la FDA basados en la detección de histonas circulantes ni de PTM de histonas específicas detectadas en tejidos. Sin embargo, existe un kit de Histonas basado en un ELISA (http://www.rapidtest.com) que se ha diseñado para el diagnóstico de enfermedades autoinmunes. En particular, el kit permite la semicuantificación de autoanticuerpos contra histonas en suero humano. Dado que la presencia de histonas puede usarse en el diagnóstico diferencial entre el lupus eritematoso sistémico y otros desórdenes raros autoinmunes que también muestran proteínas nucleares en el suero como la esclerodermia u otras enfermedades del tejido conjuntivo, el potencial del kit depende de su combinación con otras herramientas diagnósticas.
FUTUROS RETOS EN EL ANÁLISIS DE LOS BIOMARCADORES EPIGENÉTICOS EN MUESTRAS CLÍNICAS
Las pruebas médicas que se realizan en los laboratorios clínicos son generalmente parte de un proceso médico más complejo: sus resultados complementan a la anamnesis y el estudio de los signos y síntomas, ayudando a orientar el diagnóstico, las acciones médicas y la toma de decisiones terapéuticas. El desarrollo de la tecnología facilita la determinación de nuevos biomarcadores, pero no todas las tecnologías llegarán a implementarse con el mismo éxito en los laboratorios clínicos debido a su complejidad, coste o dificultad en el manejo de los datos.
Como hemos visto en esta revisión, se han producido enormes avances en los últimos años en cuanto al desarrollo e implantación de los biomarcadores epigenéticos en la rutina clínica, pero todavía queda un camino largo por recorrer, así como algunos puntos clave por solucionar, como la necesidad de adoptar nuevas metodologías de trabajo (por ejemplo, tratamiento con bisulfito del ADN, o de aislamiento de exosomas para la detección de microARN). Uno de los mayores retos está relacionado con las particularidades de los mecanismos epigenéticos, y es que todavía se necesita, en muchos casos, comprender en profundidad los procesos que regulan tanto a nivel molecular, como fisiológico y patológico. Este es el caso de los microARN, los cuales pueden regular multitud de genes y muchas veces su función se comprende en el contexto de un tejido biológico particular y no en otro, como por ejemplo al aparecer en un fluido biológico.
Otro reto adicional está relacionado con la adopción de los biomarcadores epigenéticos en un entorno clínico, donde no solo el personal del laboratorio debe conocer las tecnologías y metodologías para la determinación de este tipo de biomarcadores, sino que también el propio personal médico debe incrementar su conocimiento en relación al potencial de este tipo de biomarcadores y cómo implementarlos en su rutina de diagnóstico. Por supuesto se requiere de un esfuerzo común por parte de los laboratorios clínicos, la investigación clínica, y la industria del diagnóstico in vitro para la generación no solo de nuevos biomarcadores epigenéticos, sino también para el desarrollo y la adopción de las metodologías más manejables y sencillas posibles para adaptar los procedimientos a la rutina de un laboratorio de diagnóstico clínico. Por supuesto, todavía existen algunos cuellos de botella como el hecho de que no todos los hospitales y laboratorios clínicos pueden disponer de equipos de alto rendimiento, como el requerido para NGS, o un espectrómetro de masas. Sin embargo, este no será un problema en un futuro cercano, ya que se están abaratando este tipo de tecnologías y además la industria tiene gran interés en introducir nuevas aplicaciones y equipamientos en el entorno clínico.
La epigenética contribuirá decisivamente a la mejora de la medicina de precisión y la implementación de la teragnosis, mejorando la seguridad y la eficacia de los tratamientos. En este sentido, un gran número de iniciativas han sido anunciadas tanto en Europa como en los Estados Unidos. Por supuesto, las tecnologías epigenómicas y los biomarcadores epigenéticos empoderarán la medicina al refinar y mejorar la toma de decisiones clínicas, pero para ello también la revisión de las directivas y regulaciones sobre los dispositivos médicos de diagnóstico in vitro debe prepararse acorde con estos avances tecnológicos (Garcia-Gimenez, 2017).
AGRADECIMIENTOS
J.L.G-G agradece al INCLIVA y a la GVA la financiación de los proyectos emergentes (GV/2014/132), así como a la Acción Estratégica en Salud AES2016 (ISCIII) por la financiación en un proyecto FIS número PI16/01036, co-financiado por los Fondos de Desarrollo Regional Europeos (FEDER). J.L.G-G agradece también su soporte al Consorcio Centro de Investigación en Red del Instituto de Salud Carlos III, CIBER-ISCIII. G P. M agradece al Ministerio de Economía y Competitividad la Ayuda Torres Quevedo (PTQ15-078899). J. B. G agradece a la GVA la ayuda otorgada dentro del Programa Contratación de Personal Técnico de Transferencia Tecnológica (APOTIP/2017/012). E. B. agradece al Ministerio de Economía y Competitividad la ayuda otorgada en el marco del Programa de Doctorado Industrial (DI16-08917). E.G.L agradece a la GVA la ayuda otorgada dentro del Programa Contratación de Doctores en Empresas de la Comunitat Valenciana (AEST2016/026). C.R-M agradece a la Universitat de València y a la Fundación INCLIVA la ayuda obtenida en el programa VLC‐BIOCLINIC 2017. S.M.M. agradece a la GVA la financiación del proyecto emergente (GV/2016/109)

