El cáncer de ovario es un grupo heterogéneo de enfermedades que presentan divergencias a distintos niveles, desde su origen celular hasta sus características clínico-patológicas y moleculares, impactando sobre el pronóstico y la evolución del mismo. Junto con el avance de las ciencias biomédicas y las tecnologías de alto rendimiento, el escenario molecular del cáncer de ovario se ha transformado drásticamente en los últimos años.
En este contexto, la inestabilidad genómica (IG) y la vía de reparación del ADN por recombinación homóloga (RH) han tenido un papel crítico. La IG, conocida como uno de los hallmarks del cáncer, es la tendencia a acumular alteraciones genómicas de manera progresiva, lo que le confiere a la célula tumoral una ventaja para promover el desarrollo y la progresión tumoral (Hanahan, 2022). A pesar de ser una característica inherente a la célula tumoral, el grado de inestabilidad varia dramáticamente entre los distintos tipos de tumores. Ciertos subtipos, como el cáncer de ovario seroso de alto grado (COSAG), presentan fenotipos altamente inestables (Hanahan y Weinberg, 2011).
En el campo de la oncología existe un interés creciente por la medicina personalizada, cuyo objetivo es mejorar los resultados del tratamiento haciendo coincidir las características biológicas específicas de un tumor con la opción terapéutica más adecuada. La presencia de una alta IG y su causa subyacente abre una ventana de nuevas oportunidades al respecto (Zhao et al., 2019). Así, la detección de la IG tiene una implicación directa sobre el pronóstico y manejo clínico del paciente, específicamente en cuanto a la elección del tratamiento (Ngoi y Tan, 2021).
En particular, los inhibidores de PARP (iPARP) se han incorporado con éxito en la rutina clínica para el tratamiento del COSAG con alteraciones en la vía RH, presente en alrededor del 50% de los casos (Lheureux et al., 2019). Los iPARP más destacables son Olaparib (primer fármaco aprobado con esta actividad), Rucaparib y Niraparib (Lheureux et al., 2019, Coleman et al., 2019, González-Martín et al., 2019).
APROXIMACIONES COMERCIALES PARA LA DETERMINACIÓN DE LA INESTABILIDAD GENÓMICA
Teniendo en cuenta su importancia en el diagnóstico y la selección de pacientes para tratamientos dirigidos, se han llevado a cabo diversos estudios con el objetivo de obtener el mejor subrogado para detectar las deficiencias en la vía de RH.
Descifrar los perfiles de IG como subrogado de la deficiencia de recombinación homóloga (HRD) requiere el estudio, de modo que se objetiven las alteraciones en diferentes niveles. En este sentido, hasta el momento se han descrito una enorme cantidad de herramientas, sin que ninguna de ellas alcance la categoría de gold standard. Desde la secuenciación de genes como BRCA1/2 o paneles multi-génicos de RH, enfoques más simples y dirigidos, a la secuenciación del genoma completo (WGS) basada en la detección de CNV para detectar cicatrices genómicas. Aunque la opción elegida para ser incorporada a la rutina clínica es, en la mayoría de los casos, los paneles dirigidos, por su menor complejidad y precio, tienen como defecto una menor sensibilidad. Por ello, metodologías más complejas están ganando cada vez más fuerza en este campo (Ngoi y Tan, 2021).
En este sentido, en 2012 se publicaron tres firmas, cada una de las cuales medía un tipo específico de cicatriz genómica (Marquard et al., 2015), todas basadas en datos de array de single-nucleotide polimorphism (SNP). Su presencia ponía de manifiesto la deficiencia del sistema de reparación del ADN por RH. Las tres alteraciones que se describieron fueron: Telomeric Allelic Imbalance (TAI), aberraciones cromosómicas que implicaban la presencia de regiones sub-teloméricas con desequilibrio alélico, que comienza más allá del centrómero, extendiéndose hasta el telómero (Birkbak et al., 2012); Transiciones de gran escala (LST), rupturas cromosómicas entre regiones adyacentes de al menos 10 Mb (Popova et al., 2012); y HRD score, basada en la detección de eventos de pérdida de heterozigosidad (LOH) mayores de 15 Mb pero inferiores al cromosoma completo (Abkevich et al., 2012). Mientras que cada una de ellas ya presentaba un valor pronóstico en sí misma, se vio que la combinación de las tres mejoraba el rendimiento del biomarcador (Marquard et al., 2015). Estos hallazgos pronto promovieron la aparición de soluciones comerciales. Las dos más aceptadas en uso clínico son de myChoice®CDx de Myriad Genetics, FoundationOne®CDx de Foundation Medicine y SOPHiA DDM™ Homologous Recombination Deficiency (HRD) Solution, de Sophia Genetics.
La solución de Myriad Genetics, myChoice®CDx, determina el estado de la vía de RH mediante la detección de alteraciones en BRCA1 y BRCA2 y la evaluación de las tres cicatrices genómicas. Foundation Medicine, por su parte, desarrolló FoundationOne®CDx, una prueba que detecta y cuantifica eventos de LOH superiores a 15 Mb, además de TMB y estado mutacional de un amplio panel de genes. Por último, la solución comercial de Sophia Genetics, mediante la utilización de su algoritmo GIInger, establece el índice de integridad genómica a partir de una secuenciación de baja cobertura y las mutaciones puntuales en genes involucrados en RH.
DETERMINACIÓN IN-HOUSE DE LA INESTABILIDAD GENÓMICA: SCARFACE SCORE
El desarrollo de un biomarcador propio, que consiga igualar al gold standard, para estratificar a los pacientes que albergan defectos en la vía de RH y, por lo tanto, presentan una mayor sensibilidad a los agentes que dañan el ADN, es un paso clave en el campo de las terapias dirigidas. Sin embargo, dada la complejidad del fenotipo de IG causado por la presencia de defectos en la vía de RH, un biomarcador a un único nivel puede ser limitante. La combinación, mediante un modelo, de aproximaciones que evalúen estos defectos a distintos niveles es recomendable (Hoppe et al., 2018, Ladan et al., 2021).
Por ello, nuestro grupo construyó y ajustó tres modelos de fuente única a partir de tres tipos de datos; expresión génica, SNPs y parámetros de IG. A partir de estos tres modelos individuales, se ajustó el Scarface Score, un modelo integrativo con valor pronóstico y predictivo de respuesta a agentes que dañan el ADN, en particular la quimioterapia basada en platinos e iPARP. El modelo comprende 8 SNPs, 28 parámetros de IG y la expresión de 7 genes, alcanzando una precisión de 0,9615 y un índice kappa de 0,9128 (Fernandez-Serra et al., 2023) (Figura 1).
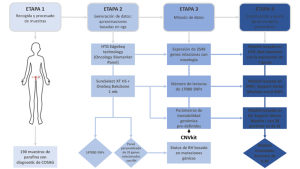
Así, mientras cada capa del modelo presenta sus propias limitaciones, cada una de ellas suple las de las demás, consiguiendo mejorar el resultado obtenido de los modelos de fuente única. Este diseño abarca los distintos mecanismos en los que se produce la deficiencia en la vía de RH, intentando imitar el complejo contexto biológico (genómico, transcriptómico, epigenético, etc…) mediante un enfoque multiómico(Stevens et al., 2020).
Sin embargo, aunque el rendimiento del Scarface Score es superior a los modelos de fuente única, siguen siendo una herramienta adecuada y eficiente que podría ser empleada en entorno clínico real, ayudando a guiar el manejo clínico de los pacientes, simplificando el enfoque y disminuyendo los costes.
VENTAJAS Y LIMITACIONES DE LA APROXIMACIÓN
A pesar de los buenos resultados de las soluciones comerciales, su uso sigue estando limitado, no siendo una opción real en el contexto clínico de la mayoría de los pacientes.
Las soluciones comerciales tienen dos problemas principales, por un lado, la externalización del procedimiento ocasiona una falta de control sobre el procesado y el análisis de las muestras tumorales, así como los datos generados. Por otro lado, los elevados precios de las determinaciones no son asumibles para la mayoría de los laboratorios clínicos. Por ello, la disponibilidad de una herramienta académica adecuada y simplificada que pueda llevarse a cabo en los laboratorios clínicos es una necesidad.
El modelo Scarface Score, así como los modelos de fuente única, cumplen con estos requisitos. La posibilidad de realizar tanto el procesamiento como el análisis en los propios laboratorios facilita y abarata el procedimiento, además de dar acceso a todos los datos generados para su explotación. Por otra parte, el modelo integrativo permite estudiar el fenómeno de la IG en su conjunto, tanto a nivel genómico, cromosómico, como transcriptómico.
En conclusión, el Scarface Score podría constituir una herramienta académica útil para predecir la respuesta a agentes que dañan el ADN en el escenario clínico del COSAG y, potencialmente, de otros tumores con deficiencias en la vía de RH. Este algoritmo cubre las necesidades que no cubren las soluciones comerciales disponibles y validadas, abordando el IG y la biología molecular del tumor desde un punto de vista más completo y a un precio más asumible.
DIRECCIONES FUTURAS
Debido a que este modelo aborda la IG desde diferentes puntos de vista, parece una estrategia plausible calibrar el modelo para predecir la respuesta con diferentes puntos de corte en otros tumores en los que la vía de RH desempeñe un papel clave, como el cáncer de próstata avanzado con mutaciones BRCA, el cáncer de páncreas o el cáncer de mama triple negativo. De manera análoga, el modelo podría ajustarse a otros fármacos similares (Ngoi y Tan, 2021).
Conflicto de Intereses
Los autores declaran la ausencia de conflictos de intereses.
Agradecimientos
Los autores agradecen al biobanco de la Fundación Instituto Valenciano de Oncología por la cesión de las muestras biológicas utilizadas para el análisis y al Grupo Español de Investigaciones en Cáncer de Ovario (GEICO) y a sus centros por la participación en el proyecto. El proyecto para el desarrollo del “Scarface Score” fue parcialmente financiado por las subvenciones de la GVA “Subvencions per a la realització de projectes d’i+d+i desenvolupats per grups d’investigació emergents (GV/2020/158)” y “Ayudas para la contratación de personal investigador en formación de carácter predoctoral” (ACIF/2016/008) y la “Beca de investigación traslacional Andrés Poveda 2020” del grupo GEICO. Este proyecto recibió el Premio “Antonio Llombart Rodriguez-FINCIVO 2020” de la Real Academia de Medicina de la Comunidad Valenciana.


