En anteriores posts hemos hablado de cómo el desarrollo de las tecnologías de secuenciación de nueva generación (NGS) ha sido crucial para el avance de la Medicina Moderna. De hecho, su desarrollo ha propiciado la mejora del diagnóstico genético, el avance de la investigación biomédica y el auge de la medicina de precisión. En esta ocasión, queremos poner el foco en una de estas técnicas de análisis genómico masivo: la secuenciación de fragmentos largos.
Esta forma de secuenciar está demostrando ser una alternativa viable y una herramienta complementaria muy útil en múltiples contextos, como, por ejemplo, el diagnóstico de enfermedades raras. ¿Quieres saber más? ¡Sigue leyendo!
¿Qué es la secuenciación de fragmentos largos y cómo funciona?
La secuenciación de fragmentos largos (long-read sequencing, en inglés) es un método para secuenciar paralelamente y de forma masiva largas cadenas de ADN (o ARN) de hasta 20 megabases, sin necesidad de fragmentarlas en pequeños trozos. Este primer “paso” de reducción del tamaño de los fragmentos a secuenciar es común en otras técnicas de secuenciación masiva, como la secuenciación por terminación cíclica reversible o la secuenciación por ión conductor.
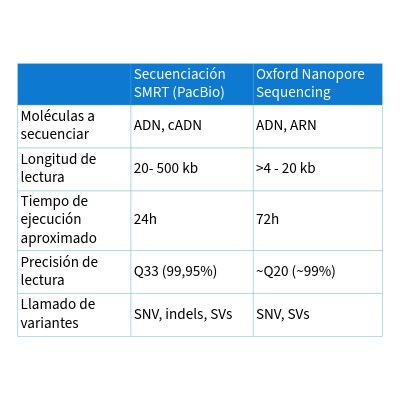
En cuanto a su funcionamiento, actualmente existen dos estrategias principales para la secuenciación de fragmentos largos: la secuenciación en tiempo real de una sola molécula de ADN (secuenciación SMRT, por sus siglas en inglés) y la secuenciación por nanoporos.
Secuenciación en tiempo real de una sola molécula de ADN (SMRT sequencing)
Este tipo de secuenciación se basa en la detección de la fluorescencia emitida por la adición de nucleótidos marcados durante la síntesis de ADN. Para ello, el ADN a secuenciar se divide en fragmentos largos, de hasta decenas de kilobases, y se circulariza, es decir, se unen sus extremos 3’ y 5’.
Tras la circularización de las cadenas largas de ADN muestra, se añade cada fragmento largo a un pocillo ZMW (zero-mode waveguide), que contiene una enzima ADN polimerasa unida a su base. Durante la secuenciación la polimerasa va copiando la cadena molde e incorporando nucleótidos marcados (cada tipo con un marcador diferente) a la nueva cadena, lo que provoca que se emita fluorescencia. El sistema va detectando la fluorescencia de cada tipo de nucleótido marcado y, así, se va determinando la secuencia del ADN muestra.
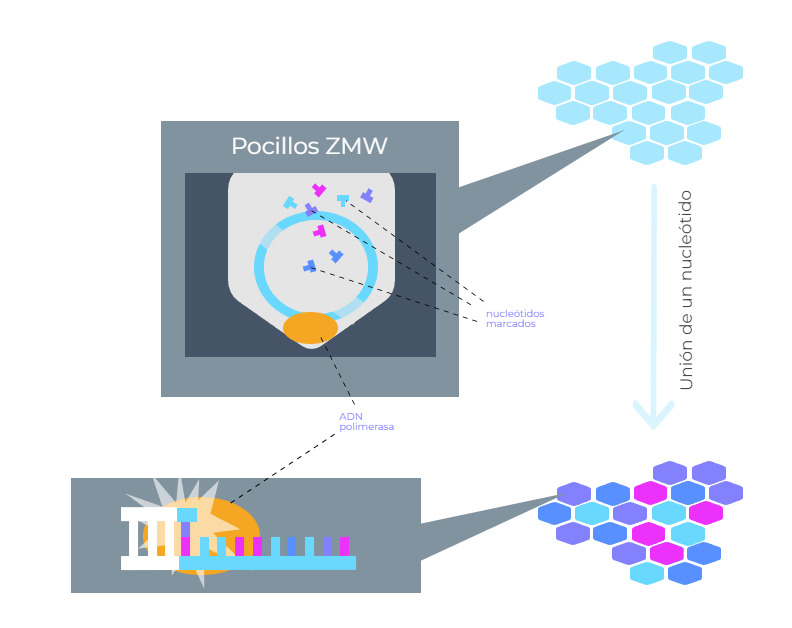
Este proceso de secuenciación se produce en miles de pocillos, lo que permite secuenciar miles de fragmentos largos de forma simultánea. Además, como se han circularizado las moléculas, la síntesis del nuevo fragmento no finaliza y cada fragmento largo puede ser secuenciado múltiples veces, lo que mejora la precisión.
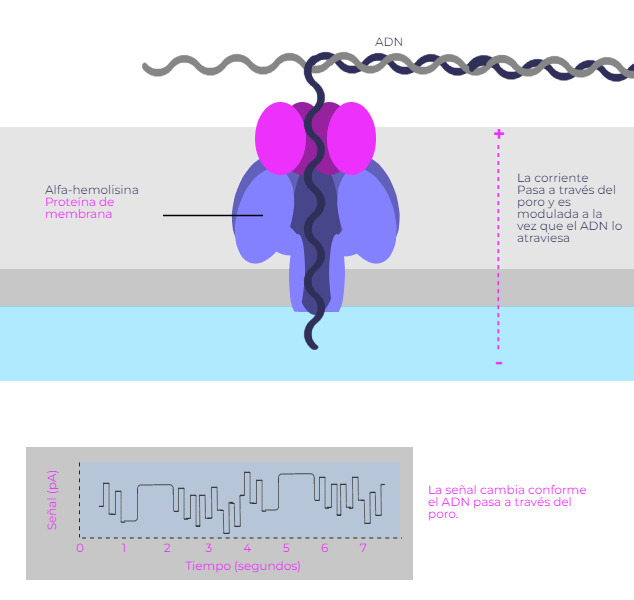
Secuenciación por nanoporos
En el método de secuenciación por nanoporos, los fragmentos largos de ADN a secuenciar pasan a través del nanoporo de una proteína de membrana, provocando cambios en la corriente eléctrica. Un algoritmo analiza los cambios en la corriente eléctrica causada por el paso de la molécula a través del nanoporo, que son diferentes dependiendo del nucleótido que pase, y va obteniendo la secuencia de nucleótidos.
Ventajas y limitaciones
La secuenciación de fragmentos largos es una opción con un gran potencial para el rendimiento diagnóstico. Diferentes estudios han demostrado que estos métodos de secuenciación NGS pueden reducir el tiempo de obtención de resultados y acelerar significativamente el diagnóstico en enfermedades raras. Esto es debido a que el análisis de fragmentos largos puede ayudar a identificar anomalías genéticas que pueden pasar desapercibidas por otros métodos, como reordenamientos complejos o variantes en regiones repetitivas.
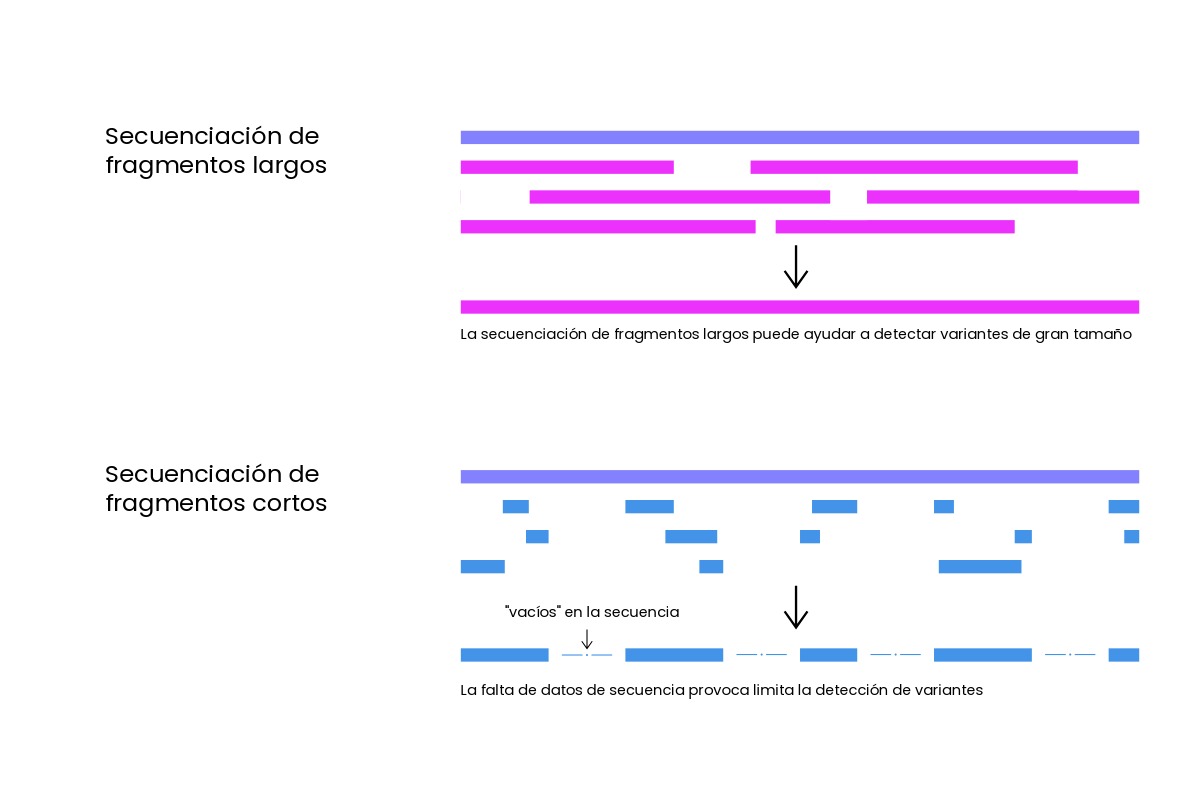
Sin embargo, este tipo de secuenciación también tiene sus limitaciones, lo que dificulta que se esté utilizando actualmente en la práctica clínica. Una de ellas es su precisión, ya que tiene una mayor tasa de error por cada lectura de la secuencia que en la secuenciación de fragmentos cortos (short-read sequencing). Otra limitación es el coste económico: la secuenciación de fragmentos largos es más costosa en comparación con la secuenciación de fragmentos cortos. Además, requiere de muestras de ADN de mayor calidad y de procesamientos computacionales más complejos que otros métodos de secuenciación.
Aplicaciones clínicas de la secuenciación de fragmentos largos
Pese a sus limitaciones, actualmente se está explorando la utilidad de la secuenciación de fragmentos largos en entornos clínicos. Por ejemplo, en el contexto de las enfermedades raras, la secuenciación de fragmentos largos puede ayudar a identificar variantes estructurales y expansiones repetitivas que se escapen en métodos de secuenciación basados en secuencias cortas.
Otra aplicación clínica de este tipo de secuenciación es en el ámbito de la Oncología. Y es que este tipo de secuenciación puede ayudar a detectar reordenamientos de gran tamaño que puedan jugar un papel importante en el desarrollo y la proliferación del cáncer.
También se está explorando el potencial de la secuenciación de fragmentos largos en el diagnóstico de enfermedades infecciosas, ya que permitiría la identificación y diagnóstico de enfermedades causadas por patógenos infecciosos de forma rápida. Esto podría ser clave para estudiar y evitar la propagación de enfermedades causadas por virus, hongos o bacterias patógenas.
Estas son solo algunas de las potenciales aplicaciones clínicas de la secuenciación de fragmentos largos. No obstante, cada vez más, esta técnica de secuenciación está demostrando ser una alternativa viable para muchos otros contextos. Si te interesa conocer mejor esta técnica de secuenciación masiva, te recomendamos el Curso Superior Universitario en Técnicas de Análisis Genómico o nuestro Experto Universitario en Genética Médica y Genómica.
Enlaces y artículos de interés:
Oehler JB, Wright H, Stark Z, Mallett AJ, Schmitz U. The application of long-read sequencing in clinical settings. Hum Genomics. 2023 Aug 8;17(1):73. doi: 10.1186/s40246-023-00522-3
Rodríguez-Santiago B, Armengol L. Tecnologías de secuenciación de nueva generación en diagnóstico genético pre- y postnatal. Diagn Prenat. 2012 Apr-Jun; 23(2):56-66. doi: 10.1016/j.diapre.2012.02.001
Long-read sequencing. NHS England. https://www.genomicseducation.hee.nhs.uk/genotes/knowledge-hub/long-read-sequencing/#synthetic-long-reads

