¡OMG! Hay siglas que se te quedan grabadas en la cabeza, que son realmente difíciles de ignorar por la gran repercusión que tienen en nuestro día a día. Los términos “CIA”, “EEUU” o “OMS” nos acompañan a todas horas en diferentes ámbitos de nuestras vidas y todo el mundo sabe qué significan. Los entendemos con total naturalidad. En el ámbito de la genética ocurre algo parecido con NGS, una sigla que aparece constantemente en investigación y diagnóstico.
En el ámbito de la genética existen muchísimas siglas (de verdad, el glosario es extremadamente extenso), pero pocas tienen la misma importancia que tiene el término “NGS”. ¿Quieres saber qué significan estas tres letras? ¡Sigue leyendo!
¿Qué significa NGS?
“NGS” es un término que proviene del inglés y que significa “Next Generation Sequencing” (en castellano, Secuenciación de Nueva Generación). Se trata de un término genérico, que surgió para englobar una gran cantidad de técnicas de secuenciación de gran utilidad para la práctica clínica.
¿Por qué se desarrolló la secuenciación de nueva generación (NGS)?
La finalización del primer borrador del Proyecto Genoma Humano a principios de siglo marcó un antes y un después en el contexto de la Genómica: se consiguió secuenciar (parcialmente) un genoma humano de referencia. El problema es que, pese a sus esfuerzos por acelerar el proceso, los investigadores tardaron muchos años en conseguirlo. Por eso, la meta en ese momento pasó de “secuenciar el genoma humano” a “secuenciar el genoma humano de una forma más eficiente, barata y rápida”.
Y entonces, en 2005, nació un nuevo tipo de tecnología de secuenciación: la NGS. Esto supuso una revolución gigantesca en el ámbito de la investigación en Genómica, tanto básica como la más aplicada. Todo ello, gracias a la capacidad de generar una mayor cantidad de lecturas del genoma que permitieran obtener un resultado más sólido, a precios más asequibles y en un menor período de tiempo.
¿Qué tipos de tecnologías de NGS existen?
Desde que se desarrollaron las primeras tecnologías de NGS a principios de siglo, científicos de todo el mundo han dedicado su trabajo a mejorarlas y a desarrollar otras nuevas y más eficientes. Actualmente, las técnicas más conocidas y utilizadas son la secuenciación por ión conductor, la secuenciación por terminación cíclica reversible, la secuenciación de una sola molécula de ADN y la secuenciación por nanoporos.
Antes de explicaros cada una de ellas, me gustaría aclarar que solamente voy a mencionar cómo funciona el proceso de secuenciación del ADN como tal en cada plataforma NGS. No obstante, existen pasos previos y posteriores que son esenciales para seguir estos métodos de secuenciación.
Secuenciación por síntesis o polimerización
Se trata de sistemas de NGS basados en la acción de una ADN polimerasa sobre una cadena de ADN que actúa como molde. Entre estas tecnologías, encontramos los secuenciadores semiconductores, los sistemas de terminación cíclica reversible y la secuenciación SMRT.
Las características propias de este tipo de estrategias de NGS hacen que no puedan utilizarse para secuenciar secuencias grandes, como genomas completos. Sin embargo, se trata de técnicas relativamente baratas y rápidas, así como bastante precisas, por lo que se suelen utilizar en laboratorios de diagnóstico para secuenciar paneles de genes pequeños.
Secuenciación por Ion Conductor (Ion Torrent Sequencing)
Se trata de una estrategia que se basa en la detección de las modificaciones en el pH que se producen en la síntesis de ADN. Para ello, se van incorporando nucleótidos a una cadena de ADN, provocando que se libere un protón (H+) en la reacción y, por tanto, que se vea modificado el pH. Para poder diferenciar cuál de los cuatro tipos de nucleótidos se ha introducido en cada posición de la secuencia, se repiten varios ciclos, cada uno de ellos, con la adición de un único tipo de nucleótido.
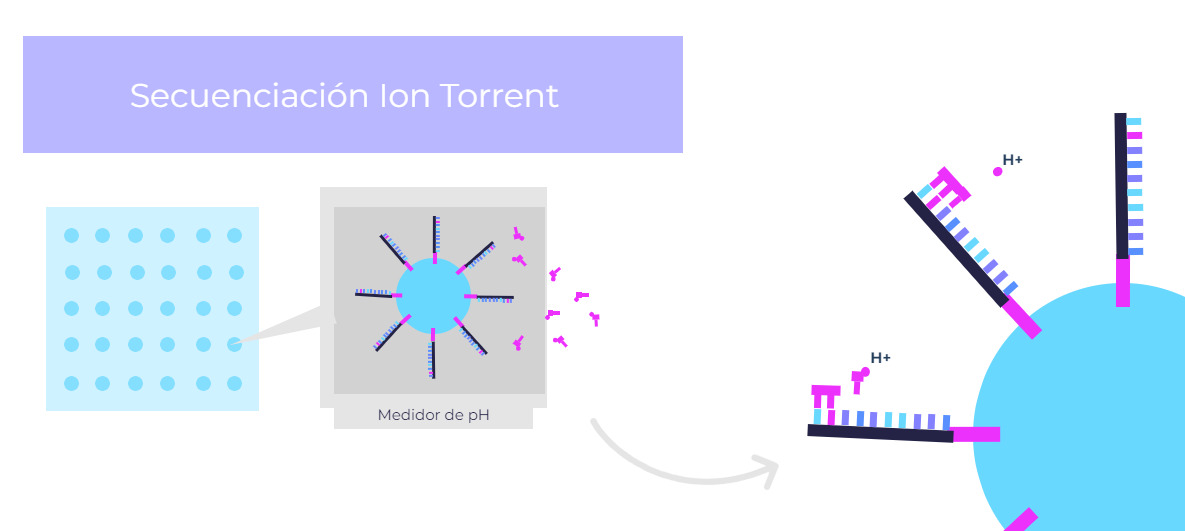
La secuenciación por ión conductor es la técnica de secuenciación utilizada en las tecnologías NGS de la empresa estadounidense Thermo Fisher Scientific.
Secuenciación por terminación cíclica reversible
Esta metodología se basa en la utilización de nucleótidos marcados con fluoróforos en una reacción de síntesis de ADN. Cada vez que uno de estos nucleótidos se incorpora a la cadena, el sistema toma una captura y registra de qué tipo de nucleótido se trata. Una vez tomada la captura, se eliminan los fluoróforos de los nucleótidos que se han incorporado y se continúa la síntesis de la cadena con nuevos nucleótidos marcados.
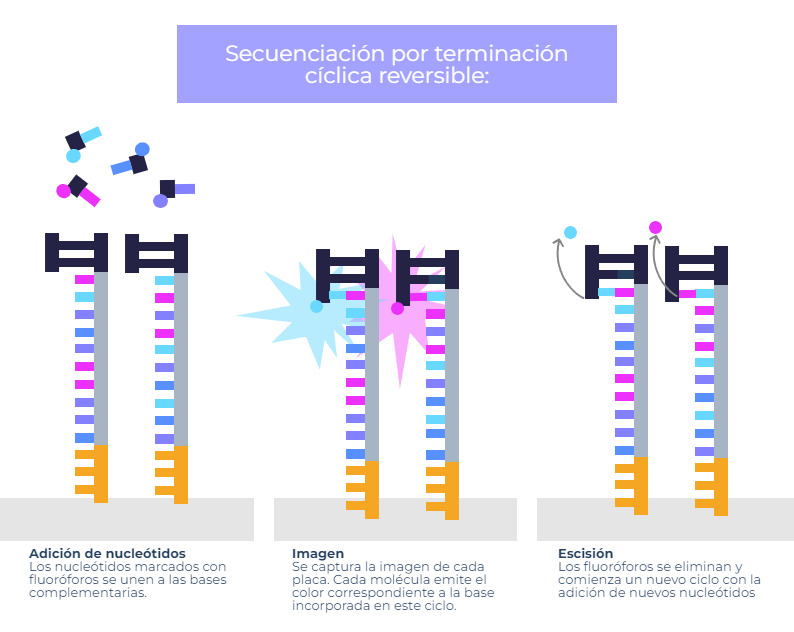
Este tipo de secuenciación es el que utilizan algunas de las tecnologías de secuenciación de Illumina, una de las compañías desarrolladoras de sistemas integrados para el análisis de moléculas de ADN más reconocidas del planeta.
Secuenciación en tiempo real
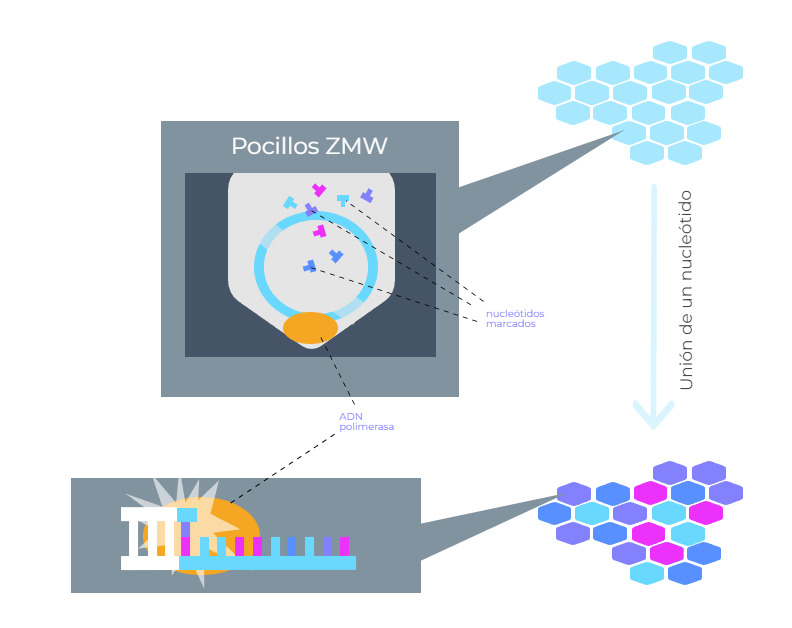
Secuenciación de una sola molécula de ADN (SMRT Sequencing)
Esta estrategia de secuenciación desarrollada por Pacific Biosciences se basa en la utilización de nucleótidos marcados que se incorporan a una nueva cadena de ADN en un proceso de síntesis. Sin embargo, en la secuenciación SMRT se utiliza un soporte especial, unos pequeños pocillos denominados “pocillos ZMW”.
En la secuenciación SMRT, las ADN polimerasas se anclan a la base de los pocillos ZMW. El ADN a secuenciar se divide en varios fragmentos y se unen sus extremos, lo que resulta en moléculas de una sola cadena de ADN “circular”. Luego, se añade uno de estos fragmentos a cada pocillo. En cada pocillo, se van incorporando nucleótidos marcados y el sistema va detectando la fluorescencia y obteniendo la secuencia de cada fragmento.
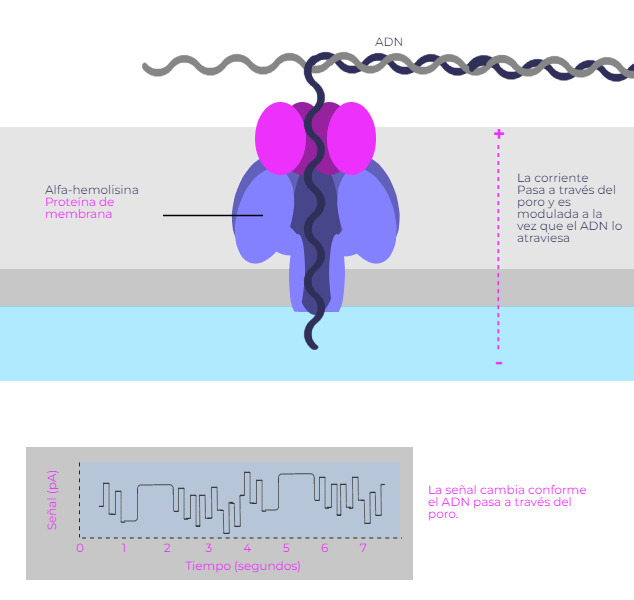
Secuenciación basada en nanoporos
En esta estrategia de secuenciación, diseñada por Oxford Nanopore Technologies, se realiza un análisis de los cambios en la corriente eléctrica que produce el paso de una molécula de ADN a través de una proteína de membrana. Un algoritmo analiza los cambios en la corriente eléctrica causada por el paso de la molécula a través del nanoporo de la proteína de membrana y va obteniendo la secuencia de nucleótidos.
Existen, además, otros tipos de NGS, como la secuenciación por ligación o la pirosecuenciación, que actualmente no son muy utilizadas en los laboratorios. Esto es debido al desarrollo de técnicas más sofisticadas y eficientes, como la presentada recientemente por Ultima Genomics, que han ido sustituyendo estas técnicas.
Aplicaciones de las NGS en Salud
En los últimos años, las tecnologías NGS se han ido mejorando en términos de rapidez y precisión, así como en cuanto a precio. Esto las ha convertido en unas interesantísimas herramientas tanto en diagnóstico como en investigación de enfermedades genéticas.
Por ejemplo, en casos de enfermedades genéticas complejas que presentan cuadros clínicos poco característicos, la secuenciación del exoma es una herramienta súper útil para emitir un diagnóstico final.
Otro ejemplo es en el campo de la Oncología. En este ámbito, la utilización de la secuenciación masiva es interesante de cara a la detección de mutaciones genéticas relacionadas con el desarrollo del cáncer. En este mismo contexto, las NGS se han vuelto unas tecnologías de especial relevancia a la hora de determinar si un paciente es susceptible a recibir un tratamiento u otro en base a las mutaciones de sus células cancerosas.
Y bien, hasta aquí el post de hoy. Espero que hayáis aprendido muchísimo acerca de las tecnologías NGS más importantes en la actualidad. Si queréis profundizar más en este tipo de estrategias y sus aplicaciones en diferentes ámbitos de la Salud Humana, no podéis perderos nuestros programas formativos de especialización. Y si queréis ir más allá y convertiros en todos unos expertos, tenéis que echar un vistazo a nuestra formación universitaria. ¡Nos leemos en el siguiente post!
Enlaces de interés:
A beginner’s guide to NGS. Illumina.
What is Next-Generation Sequencing (NGS)? ThermoFisher.
Secuenciación de nueva generación (NGS) de ADN: presente y futuro en la práctica clínica
Goodwin S, McPherson JD, McCombie WR. Coming of age: ten years of next-generation sequencing technologies. Nat Rev Genet. 2016 May 17;17(6):333-51. doi: 10.1038/nrg.2016.49


