INTRODUCCIÓN
Aquel controversial experimento realizado por el investigador He Jiankui marcó inexorablemente un antes y un después en el entramado evolutivo de los seres humanos, así como iluminó el advenimiento de las múltiples quimeras que derivarán de los mismos. Es así como las gemelas Lulu y Nana representan un punto de inflexión en la edición de genomas de seres humanos y sus descendencias representarán las primeras ramas evolutivas editadas a voluntad por el hombre.
Cerca de cumplirse un año de la noticia (Jiankui, 2018; Regalado, 2018), un grupo de jóvenes estudiantes de la carrera de Ingeniería en Biotecnología de Ikiam, una pujante Universidad enclavada en el corazón de la Amazonía ecuatoriana, tomó la iniciativa de discernir sobre algunas inconsistencias técnicas del experimento realizado por He. Todo comenzó en los hallazgos obtenidos de un video subido a la plataforma de YouTube de la ambigua presentación que realizó He durante la 2da Cumbre Internacional sobre Edición del Genoma Humano. Fotos extraídas de los videos revelaron trágicamente que el ARN guía (ARNg) diseñado y empleado por He para editar a las bebés provoca una anomalía genómica con efectos desconocidos y sin precedentes. Sin embargo, He, a pesar de conocer esta información antes de modificar a las gemelas, continuó adelante con sus experimentos. Lamentablemente, las niñas nacidas son mosaicos, con la posibilidad de llevar múltiples variantes alélicas cada una de ellas, incluida la variante original (que seguiría permitiendo la entrada del virus). Al final, además de no haber logrado conquistar su deseo de crear los primeros seres humanos resistentes al HIV, He fue incapaz de detener a tiempo la crónica de una muerte anunciada.
Aspectos generales sobre la técnica CRISPR/Cas9 para la edición de genomas
La técnica de edición genética CRISPR/Cas9 está formada básicamente por dos componentes: una endonucleasa asociada a CRISPR (en este caso la proteína Cas9) y un ARNg, tal como se puede apreciar en la Figura 1. La proteína Cas9 fue aislada originalmente de la bacteria Streptococcus pyogenes y tiene actividad endonucleasa debido a sus dos distintos dominios: un dominio de nucleasa tipo HNH, que permite escindir la cadena de ADN complementaria a la secuencia del ARN guía, y un dominio de nucleasa tipo RuvC, responsable de escindir el ADN de cadena opuesta a la complementaria (hebra no objetivo) (Chen et al., 2014). El segundo elemento, el ARNg, por su naturaleza sintética, está compuesto por una secuencia de andamiaje (scaffold) y un espaciador de alrededor de 20 nucleótidos que define el objetivo genético a modificar (Jinek et al., 2012).
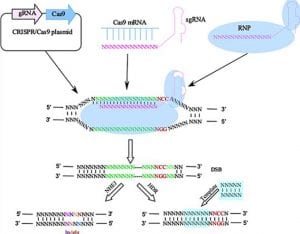
Una vez enlazados ambos elementos, se forma un complejo de ribonucleoproteína (RNP) a través de interacciones del scaffold del ARNg con la superficie cargada positivamente de la proteína Cas9. Esta unión provoca un cambio conformacional en Cas9, activándola y permitiendo la unión al ADN, donde el ARNg queda libre para la búsqueda del ADN objetivo o diana mediante complementariedad (Jinek et al., 2014). La unión de la proteína Cas9 y la secuencia diana ocurre a través de una señal de unión, que es mediada por un motivo adyacente al protoespaciador (PAM, por sus siglas en inglés, Protospacer Adjacent Motif). La longitud de la secuencia PAM está conservada, es de alrededor de 3 a 5 nucleótidos y depende de qué proteína Cas9 se utilice (Gasiunas et al., 2012). Una vez reconocido este motivo adyacente, la proteína Cas9 sufre un segundo cambio conformacional permitiendo unir los dominios de la endonucleasa al ADN objetivo y posteriormente realizar el corte. El producto de la escisión del complejo CRISPR/Cas9 es una ruptura de doble cadena del ADN diana (Nishimatsu et al., 2014).
La ruptura de doble cadena de ADN es restaurada espontáneamente por mecanismos de reparación, los cuales están altamente conservados entre eucariotas (Haber, 2000). La unión de extremos del ADN no homólogo (NHEJ, por sus siglas en inglés Non-homologous end joining) y la reparación dirigida por homología (HDR, por sus siglas en inglés Homology Directed Repair) son las dos vías principales de reparación del ADN (Figura 1). El HDR es una vía que utiliza un fragmento de ADN como molde, como una copia cromosómica hermana intacta, por lo tanto, puede generar una reparación perfecta. Sin embargo, este mecanismo depende de ciertos factores, tales como el tipo de especie a editar, el tipo de célula y la etapa del ciclo celular (Downs & Jackson, 2004). El mecanismo NHEJ se ha considerado como la vía principal de reparación del ADN en mamíferos. Este es un proceso imperfecto que frecuentemente produce inserciones, deleciones (INDELs) o sustituciones de nucleótidos en los extremos del sitio de escisión (Guirouilh et al., 2004), provocando mutaciones y la consecuente inactivación de genes.
La herramienta CRISPR/Cas9 presenta muchas ventajas ya que permite la modificación de genes a voluntad, debido a su versatilidad y al hecho de ser programable. Sin embargo, presenta inconvenientes como la precisión, ya que en determinadas ocasiones puede escindir secuencias nucleotídicas muy parecidas, causando alteraciones no deseadas en genes similares que no deberían ser modificados, tal como ocurrió con las gemelas Lulu y Nana. Esta es una limitación que puede reducirse al mínimo si se diseñan cada vez mejores ARN guías y se seleccionan tijeras moleculares (endonucleasas) con mayor precisión.
CONTRADICCIONES TÉCNICAS EN EL DISEÑO DEL ARNg O CRÓNICA DE UNA MUERTE ANUNCIADA
El sistema CRISPR/Cas9 es una técnica que ha ganado adeptos en la comunidad científica debido a su simplicidad, velocidad y eficiencia para modificar genes en cualquier organismo, además de ser programable para escindir cualquier secuencia (Rodríguez et al., 2019). La especificidad de esta técnica depende del segmento de ADN diana dentro del gen que se desea interrumpir, la nucleasa utilizada y el ARNg con su respectiva secuencia PAM. El ARNg depende de la diana utilizada, donde los objetivos dentro de la secuencia diana se denominan on-targets, mientras que los objetivos fuera de lugar son denominados off-targets (Ma et al., 2014). Cuando se diseña el ARNg a utilizar en cualquier experimento de edición genómica, se requiere que el número de off-targets sea cero o el menor posible (Ma et al., 2014). Actualmente, existe poca información sobre las consecuencias de las modificaciones fuera del objetivo, por ello se precisa más estudios que permitan cuantificar y elucidar los efectos secundarios biológicos en la edición de genes, para así poder preverlos (Rodríguez et al., 2019). Por esta razón, la correcta escisión, entre otros aspectos técnicos, va a depender del ARNg utilizado.
Los hallazgos obtenidos extrayendo información de un conjunto de fotografías tomadas durante la corta y ambigua presentación de He durante el 2do Congreso de Edición de Genoma Humano celebrado en Hong Kong en el 2018, han podido mostrar que el ARNg diseñado y utilizado por He para la edición genómica de Lulu y Nana provoca una gran cantidad de off-targets y un concomitante alto riesgo de mosaicismo. Es de esperar que esta información la conocía He antes de editar a las gemelas, sin embargo, continuó adelante con sus experimentos.
La Tabla 1 muestra los resultados obtenidos de la evaluación de especificidad del ARNg utilizado por He, donde se cuantifican los off-targets de acuerdo al número de errores. En este caso, los resultados obtenidos mostraron que el ARNg diseñado y empleado por He en sus experimentos presentan un gran número de off-targets, además de que ninguno de los algoritmos utilizados recomendó el uso del mismo. La utilización de un ARNg con varios objetivos fuera de lugar puede ocasionar mosaicismo genético en el organismo a editar. Esta anomalía, se define como la presencia de más de un genotipo en un mismo individuo, es decir, que las poblaciones celulares tienen distintas modificaciones que las hacen distintas entre sí (Mehravar et al., 2019). El mosaicismo tiene efectos desconocidos en un organismo y van a depender de la modificación genética que se haya realizado.
Tabla 1. Evaluación del ARNg diseñado y utilizado por He Jiankui en sus experimentos.
|
ARNg: 5’…CAGAATTGATACTGACTGTATGG…3’ (-) |
|||||||
|
Nombre |
Off-targets* |
Sitio Web |
Referencia |
||||
| 0 | 1 | 2 | 3 | 4 | |||
| Breaking-Cas | 0 | 0 | 1 | 8 | 134 | http://bioinfogp.cnb.csic.es/tools/ breakingcas |
Oliveros et al., 2016 |
| Cas-OFFinder | 0 | 0 | 1 | 8 | 134 | http://www.rgenome.net/cas-offinder/ | Bae, Park, & Kim, 2014 |
| CCTop | 0 | 0 | 1 | 5 | 65 | https://crispr.cos.uni-heidelberg.de/ | Stemmer et al., 2015 |
| CRISPOR | 0 | 1 | 1 | 13 | 136 | http://crispor.tefor.net/ | Haeussler et al., 2016 |
| Off-Spotter | 0 | 0 | 1 | 3 | 30 | https://cm.jefferson.edu/Off-Spotter/ | Pliatsika & Rigoutsos, 2015 |
| IDT | 0 | 0 | 3 | 39 | 57 | https://www.idtdna.com/site/order/ designtool/index/CRISPR_CUSTOM |
Integrated DNA Technologies, Inc. |
| CHOPCHOP | 0 | 0 | 1 | 9 | S/I | http://chopchop.cbu.uib.no | Montague et al., 2014 |
| GT-Scan | 0 | 0 | 1 | 8 | S/I | https://gt-scan.csiro.au/gt-scan | O’Brien & Bailey, 2014 |
| COSMID | 3 | 9 | 1 | 9 | S/I | https://crispr.bme.gatech.edu/ | Cradick et al., 2014 |
S/I: Sin información
*Número de errores de las secuencias off-targets
Según indicó He en la charla realizada durante el 2do Congreso de Edición de Genoma Humano, él había realizado un ensayo en ratones donde afirmaba haber obtenido un 93% de eficiencia utilizando su ARNg (Figura 2B). Sin embargo, como ya se demostró (Tabla 1), este resultado no pudo haber sido cierto debido al alto número de off-targets obtenidos con el ARNg que empleó para editar a Lulu y Nana. Además, He declaró que solamente una de las niñas presentaba mosaicismo, al tiempo que afirmaba que estos cambios en su genoma no representan riesgo alguno para su integridad física. Dicha aseveración es imposible que sea cierta, ya que los resultados muestran que es inevitable que ambas gemelas presenten numerosos poliformismos en sus respectivos genomas, lo cual solamente podría determinarse con la secuenciación de los mismos y mediante sólidos estudios de genómica comparativa. A pesar de que He indicó en su ponencia que había secuenciado los genomas de las gemelas, no es posible corroborar esta afirmación, ya que no existe ninguna publicación o información científica fidedigna que lo pueda confirmar. ¿Se tratará éste polémico experimento de una “fábula” o de una “barbarie”? En este sentido, todos esperamos que la sublime intriga sea despejada con la primera respuesta.