Amparo Tolosa, Genotipia
Las herramientas de edición del genoma basadas en CRISPR podrían incorporar pronto un nuevo miembro en su equipo. Investigadores de la Universidad de California Berkeley describen en un artículo recientemente publicado en Science, el sistema CRISPR-CasΦ, detectado en grandes virus bacteriófagos, que incluye un nuevo tipo de nucleasa Cas con importantes ventajas para las diferentes aplicaciones en biotecnología o medicina.
Los autores del trabajo presentan el sistema CRISPR- CasΦ como una potencial herramienta compacta de edición genómica, que ha mostrado resultados preliminares prometedores en células humanas y vegetales. Además, plantean que la existencia inesperada de un sistema CRISPR en virus podría representar un sistema para eliminar otros fagos competidores.
De mecanismo de defensa bacteriano a sistema para modificar el genoma
Hace apenas ocho años, la introducción del sistema CRISPR-Cas9 como técnica para introducir cambios específicos en el genoma de un organismo representó un punto de inflexión en el campo de la biotecnología y el estudio de las enfermedades genéticas, además de abrir una vía hacia el desarrollo de potenciales terapias para múltiples enfermedades.
La versión original de CRISPR-Cas, caracterizada en diversos organismos procariotas, es un mecanismo de defensa –descrito por primera vez por el investigador español Francis Mojica– que utiliza ARNs no codificantes y nucleasas para eliminar el material hereditario de agentes patógenos para los microorganismos. Los ARNs no codificantes derivan de las secuencias CRISPR, secuencias repetitivas intercaladas por fragmentos de secuencias de virus o plásmidos que han infectado previamente al microrganismo. Una vez generados, estos ARNs no codificantes permanecen en el interior de la bacteria como una memoria molecular de las amenazas anteriores, preparados para posibles nuevos contactos. El segundo componente del sistema, la nucleasa, interviene en el procesamiento de los ARNs y los utiliza como guías de reconocimiento para atacar el ADN de los patógenos cuando la bacteria vuelve a estar expuesta a ellos.
Desde la descripción de la primera herramienta de edición CRISPR se han ido identificando diferentes sistemas CRISPR y nucleasas asociadas (conocidas como Cas), con características variadas, que han ampliado el catálogo de aplicaciones posibles con las herramientas CRISPR. La última incorporación, sin embargo, no procede de un microorganismo, sino de un nuevo tipo de virus bacteriófago.
Virus con su propio antivirus
Los megafagos gigantes o Biggiephages son virus de gran tamaño (en comparación con otros virus) presentes en múltiples ambientes, desde suelos húmedos, a lagos, océanos o el microbioma de múltiples animales, incluidos los humanos. Pese a su ubicuidad, su descubrimiento es reciente, y procede, no de su purificación y cultivo en laboratorio, sino de análisis metagenómicos. Este tipo de estudios obtiene información de las poblaciones de microorganismos de una muestra a partir del análisis de todo el ADN que se detecta en la misma.
Una de las características de los megafagos gigantes es que su genoma puede ser hasta 15 veces mayor que el de los bacteriófagos normales. No obstante, lo más llamativo (e inesperado) para los investigadores ha sido la identificación de un sistema CRISPR como el que tienen las bacterias para defenderse de otros virus.
¿Para qué necesita un virus su propio sistema CRISPR de defensa frente a otros virus? Los investigadores plantean que se trata de una estrategia para evitar la competencia. Cuando los megafagos introducen su ADN en el interior de la bacteria incluyen sus secuencias CRISPR con fragmentos de otros virus y las instrucciones para producir sus nucleases específicas denominadas CasΦ (Cas-phi). Gracias a los fragmentos de ARN que se producen a partir del ADN de las secuencias CRISPR del fago, la nucleasa puede reconocer y eliminar el ADN de potenciales virus competidores. De esta forma el megafago gigante retiene para él los recursos de la bacteria. Esta propiedad habría sido adquirida por los megafagos de las bacterias.
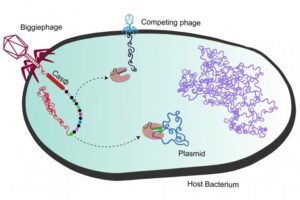
Los investigadores plantean que la existencia inesperada de un sistema CRISPR en virus podría representar un sistema para eliminar otros fagos competidores. Imagen: UC Berkeley image by Basem Al-Shayeb and Patrick Pausch.
La nucleasa Casφ: pequeña pero poderosa
Cuando caracterizaron el sistema CRISPR- Cas presente en los megafagos gigantes, los investigadores encontraron diversas características que podrían ser de gran utilidad en el contexto de la edición genómica.
En primer lugar, el tamaño de la enzimas CasΦ identificadas en los megafagos gigantes es mucho menor que el de las otras Cas (cerca de la mitad que Cas9), lo que se traduce en que las instrucciones necesarias para producirlas (la secuencia de ADN) son más cortas. Esta característica es una gran ventaja a la hora de diseñar un sistema de edición del genoma, ya que los vectores que se suelen utilizar para introducir las herramientas de edición en el interior de las células tienen limitaciones de espacio. Además, utiliza el mismo centro activo para procesar el ARN y para cortar el ADN. Otra ventaja es que CasΦ corta la doble cadena de ADN y puede dirigirse a una mayor variedad de secuencias diana que Cas9.
“Lo que hizo que realmente me interesara por estudiar esta proteína específicamente es que todos los sistemas CRISPR-Cas habían sido descubiertos originalmente en bacterias y arqueas como medio para defenderse de virus, pero esta era la única vez que se encontraba un tipo de sistema CRISPR completamente nuevo, el único encontrado hasta la fecha en genoma viral”, destaca Basem Al-Shayev, investigador doctoral en el Instituto de Innovación Genómica de la Universidad de California Berkeley. Al-Shayev firma el trabajo como primer autor, posición compartida con el investigador postdoctoral Patrick Pausch.
De momento, los investigadores han evaluado si las nucleasas de tipo CasΦ puede adaptarse a un sistema de edición genómica en dos escenarios: células humanas y vegetales. En el primer caso, el equipo observó que una de las nucleasas CasΦ, combinada con un ARN individual, fue capaz de editar hasta un 33% de las células. Estos resultados son similares a los primeros obtenidos con otras enzimas Cas, lo que resulta muy prometedor. Respecto a la prueba en células vegetales, los investigadores consiguieron introducir deleciones en el gen diana tanto en células in vitro como en protoplastos de plantas.
“Este estudio muestra que esta proteína CRISPR-Cas codificada en un virus es realmente muy buena en lo que hace, aunque sea mucho más pequeña”, señala Jennifer Doudna, profesora de la Universidad de Berkeley y directora del Instituto de Innovación Genómica de la misma universidad, además de investigadora senior del artículo. Doudna, ampliamente conocida por su labor en el desarrollo de la tecnología CRISPR de edición del genoma, destaca la importancia del reducido tamaño de CasΦ para hacer llegar los componentes del sistema CRISPR a las células: “Cuando pensamos en cómo podría ser aplicado CRISPR en el futuro, la administración es uno de nuestros principales cuellos de botella en el campo ahora mismo. Pensamos que este pequeño sistema CRISPR-Cas9 codificado en un virus podría ser una forma de romper esa barrera”.
Estudios futuros deberán analizar si la precisión y especificidad de CasΦ son lo suficientemente elevadas para las diferentes aplicaciones biotecnológicas. En cualquier caso los resultados del trabajo presentan un sistema CRISPR compacto que podría contribuir al desarrollo de herramientas de edición genómica más eficientes y versátiles. “Proteínas tan compactas podrían ser susceptibles de modificarse y evolucionar en el laboratorio para crear nuevas funcionalidades en la manipulación de genomas”, destacan los investigadores.
Por otra parte, el reciente descubrimiento de los megafagos y la identificación del sistema CRISPR- Cas viral, resaltan que, entre lo mucho que se desconoce sobre el mundo microscópico, todavía pueden existir múltiples herramientas moleculares con gran potencial para aplicaciones biotecnológicas.
Referencias:
Megaphages harbor mini-Cas proteins ideal for gene editing. https://news.berkeley.edu/2020/07/16/megaphages-harbor-mini-cas-proteins-ideal-for-gene-editing/
Pausch P, et al. CRISPR-CasΦ from huge phages is a hypercompact genome editor. Science. 2020. DOI: http://dx.doi.org/10.1126/science.abb1400
Si te ha gustado esta noticia y quieres aprender más sobre Genética en Medicina, te interesan nuestros cursos y formación universitaria.