Eduardo Eyras
Universitat Pompeu Fabra e Institución Catalana de Investigación y Estudios Avanzados
El transcriptoma y la genómica del cáncer
El cáncer se origina a partir alteraciones genéticas que interfieren con mecanismos esenciales para el ciclo vital de las células, como son la reparación del ADN, el control de la duplicación y la activación de la muerte celular o apoptosis. Dichas alteraciones pueden tomar forma de mutaciones en posiciones específicas del genoma, o bien la eliminación o la generación de múltiples copias de una región determinada. Las nuevas técnicas de secuenciación han facilitado la detección de alteraciones en el genoma de múltiples tumores y han puesto de manifiesto la variabilidad genética del tumor entre diferentes individuos y dentro de cada individuo. Dichos estudios también han permitido identificar aquellas alteraciones más probablemente relevantes, llamadas drivers, que dirigirían las transformaciones del tumor, y diferenciarlas de mutaciones secundarias, llamadas passengers. Hasta ahora, la mayoría de los estudios de genómica del cáncer se han centrado en el análisis de alteraciones que afectan a regiones que codifican para proteína, las cuales están distribuidas en unos 20.000 genes, y en cómo estos cambios afectarían a la función de dichas proteínas. Sin embargo, otros procesos relacionados con la transmisión de la información genética hasta llegar a la síntesis de proteínas han sido menos explorados.
La información genética, antes de dar lugar a proteínas, genera moléculas de ARN mediante la transcripción y el splicing. El splicing es el mecanismo por el cual la información codificada en exones a lo largo del genoma, e interrumpida por los llamados intrones, elimina dichos intrones para dar lugar a las moléculas de ARN maduro. La mayoría de los genes dan lugar a múltiples moléculas de ARN mediante el proceso del splicing alternativo, y por tanto pueden producir a partir de un único gen diferentes proteínas con funciones potencialmente muy diversas, o moléculas de ARN no codificantes. El conjunto de moléculas de ARN en la célula se denomina transcriptoma, y el splicing es esencial para definir y entender el transcriptoma. El splicing está controlado por múltiples complejos moleculares, compuestos principalmente por un gran número de proteínas, entre las que destacan aquellas que interaccionan con los ARNs (RBPs – RNA binding proteins). Dichos complejos se unen al ARN en distintos lugares, o motivos de unión a ARN, para mediar su procesamiento. La molécula de ARN resultante es el producto de un equilibrio entre las concentraciones y las eficiencias de los diferentes complejos actuando sobre el ARN. Cambios en dichos complejos dan lugar al splicing alternativo, el cual es un mecanismo esencial en determinados procesos biológicos como la diferenciación celular.
El splicing y la terapia en cáncer
Aunque el cáncer se origina a partir de alteraciones en el ADN, éstas tienen un impacto en el transcriptoma, el cual puede inducir y mantener diferentes mecanismos vinculados al desarrollo del cáncer. De hecho, al ser el transcriptoma el que define la capacidad funcional de una célula, se puede considerar que es el transcriptoma el que de una forma u otra da lugar al estado tumoral. A lo largo de los últimos años, múltiples trabajos científicos han relacionando las alteraciones del splicing alternativo con el cáncer, y recientemente se han descubierto que dichas alteraciones son también relevantes desde un punto de vista clínico. Este es el caso de un cambio de splicing detectado en el gen MET en pacientes con adenocarcinoma de pulmón. Dicho cambio consiste en la omisión de exón en el ARN maduro y da lugar a la eliminación de una región de la proteína que actúa como represora de la actividad catalítica (Ma et al. 2003). Sorprendentemente, los tumores que muestran dicha omisión del exón en MET, sin ninguna otra alteración en el gen MET, responden a las terapias dirigidas contra MET, originalmente desarrolladas para actuar contra mutaciones que afectan a la proteína (Paik et al. 2015, Frampton et al 2015). Este resultado sugiere que las alteraciones del splicing pueden ser consideradas como posibles dianas de terapia en cáncer. Hasta ahora, el cambio de splicing de MET se ha podido explicar como consecuencia de mutaciones en las regiones no codificantes que controlan el splicing de dicho exón. Sin embargo, cabe pensar que el mismo cambio de splicing podría tener lugar debido a otros mecanismos aún por descubrir. El cambio de splicing en MET, independientemente de su origen, se convertiría en un evento con información clínica relevante o como se suele llamar, en una alteración accionable.
El transcriptoma no sólo permite la identificación de posibles estrategias terapéuticas nuevas. Recientemente se ha observado que las alteraciones en el splicing alternativo también son esenciales para entender la resistencia a fármacos. Este es el caso de la terapia dirigida contra el gen BRAF, que aparece mutado en varios tumores, y especialmente en melanoma. Aunque existe un tratamiento específico dirigido a tumores con mutaciones en BRAF, se ha observado que un número de pacientes con dichas mutaciones BRAF no responden a tratamiento. Un análisis del transcriptoma de dichos pacientes ha descubierto que en pacientes resistentes a la terapia, el ARN de BRAF carece de varios exones (Poulikakos et al. 2011). Aunque estos exones no se expresan en el ARN, su secuencia genómica está aún presente en las células del tumor, por lo que se trata de un caso de splicing alternativo, que además ha sido asociado a mutaciones en un intrón del gen BRAF (Salton et al. 2015). También se ha demostrado que cuando se revierte el patrón de splicing con moléculas moduladoras del splicing, las células tumorales recuperan su sensibilidad a la terapia (Salton et al. 2015). Un mecanismo similar se ha observado en relación a terapias inmunes en leucemias linfoblásticas agudas de células B. En pacientes resistentes a terapia se ha detectado que el ARN del gen diana CD19, tiene un cambio de splicing que no ocurre en tumores no resistentes (Sotillo et al. 2015). En este caso, dicha alteración es debida a un cambio en la expresión de una RBP que controlaría el splicing de CD19. Estos mecanismos de resistencia indican que alteraciones específicas en el splicing pueden proporcionar una ventaja selectiva a los tumores, y sugieren que algunos de estos cambios de splicing podrían incluso considerarse drivers (AS-driver – alternative splicing driver) del cáncer.
La determinación del transcriptoma en tumores se convierte por tanto en un recurso esencial para entender las propiedades del tumor y las posibles estrategias para tratarlo. Esto adquiere especial relevancia en tumores que carecen de dianas terapéuticas conocidas o mutaciones en drivers conocidos que permitan establecer una estrategia terapéutica concreta. Dichos tumores son denominados pan-negativos, y los pacientes en estos casos no pueden beneficiarse de las terapias disponibles. Las alteraciones en el splicing alternativo podrían jugar un papel fundamental para el desarrollo de nuevas estrategias terapéuticas que permitan mejorar la supervivencia de los pacientes pan-negativos.
Tras los mecanismos de splicing alternativo en cáncer
Las alteraciones en el splicing que confieren ventaja selectiva a las células tumorales pueden ser causadas por mutaciones en las secuencias reguladoras del splicing o por alteraciones en la actividad de los factores involucrados en dicha regulación. Para los síndromes mielodisplásicos, leucemias linfoides y adenocarcinomas de pulmón se han descrito mutaciones frecuentes en varios factores de splicing (Yoshida et al. 2011, Brooks et al. 2014), pero se desconoce la relevancia de estas alteraciones en otros tipos de tumor. Los cambios en la expresión de los factores de splicing también pueden desencadenar procesos tumorales, y diversos factores se han definido como oncogénicos (Karni et al. 2007) o supresores (Wang et al. 2014) de tumor, dependiendo de su efecto. Tanto las mutaciones como los cambios de expresión pueden dar lugar a cambios de splicing en genes, los cuales a su vez pueden recapitular sustancialmente fenotipos asociados con el cáncer. Este es el caso del gen NUMB, para el que un cambio de splicing se ha relacionado con un incremento de la proliferación celular (Misquitta-Ali et al. 2011). Los mecanismos causantes del cambio de splicing pueden llegar a ser complejos, ya que podría estar controlado por múltiples factores. Por ejemplo, el de splicing en NUMB se ha descrito como consecuencia de mutaciones en el factor RBM10 (Bechara et al. 2013) o por la alteración de la expresión del factor QKI (Zong et al. 2014). Por lo tanto, los cambios del splicing que caracterizan y contribuyen a la fisiopatología del cáncer vienen determinados por las alteraciones de una red compleja de proteínas de unión a ARN. Esta red de regulación está aún por determinar y muchos de los cambios de splicing con relevancia funcional en cáncer están por describir.
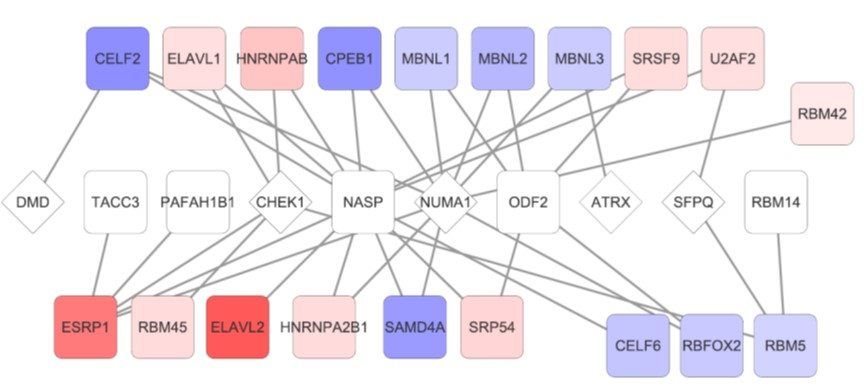
En nuestro laboratorio hemos llevado a cabo recientemente un estudio de las alteraciones en cáncer en los factores que se unen a ARN y los cambios de splicing asociados con el fin de dilucidar dicha red de regulación (Sebestyén et al. 2016). Utilizado las mismas técnicas de secuenciación del ADN, los proyectos de genómica de cáncer han podido medir el ARN presente en tumores. Hemos aprovechado el libre acceso a estos datos para determinar la relevancia del transcriptoma en cáncer. Así, usando datos del proyecto TCGA (The Cancer Genome Atlas) para más de 4.000 muestras de 11 tumores sólidos, hemos analizado las mutaciones, alteraciones en el número de copias y cambios de expresión de unos 1348 genes que codifican para proteínas que presentan evidencia de unión a ARNs (RBPs), además de los cambios de splicing asociados a dichas alteraciones. La mayoría de las RBPs presentan cambios de expresión en algún tipo de tumor comparado con muestras normales y muchos de estos cambios están asociados a duplicaciones de las regiones genómicas del gen. En contraste, las RBPs están poco mutadas, y en general menos frecuentemente que las que ya se han observado anteriormente en otros tumores. Con el fin de estudiar el impacto de las alteraciones en estas RBPs en el transcriptoma, medimos los cambios de splicing potencialmente debidos a las mutaciones o los cambios de expresión de las RBPs. Este análisis mostró que tienen lugar muchos más cambios en el splicing, posiblemente debidos a los cambios de expresión. Con el fin de determinar con mayor precisión la relación entre las RBPs y los cambios de splicing observados, calculamos los posibles sitios de unión al ARN de las RBPs en las regiones variables de los genes, también llamados eventos, y la posible asociación mediante la correlación de la expresión de las RBPs y el patrón de splicing de cada evento. Esto nos permitió reconstruir una red de regulación en la que para cada gen que cambia splicing en cáncer obtenemos las RBPs que lo controlarían en cada tumor. Esta red nos proporciona hipótesis mecanísticas concretas que se pueden validar experimentalmente. En dicho contexto, nos interesa examinar mecanismos que involucran a genes que participan en funciones celulares que juegan un papel importante en cáncer. Nuestro análisis predijo que la RBP MBNL1, un factor de splicing asociado a la diferenciación celular (Han et al. 2013), controla el splicing alternativo de varios genes involucrados en el ciclo celular, incluido el gen NUMA1, en tumores de mama de tipo luminal. En colaboración con el laboratorio de Juan Valcárcel (CRG, Barcelona) validamos esta hipótesis experimentalmente. Usando células mamarias, observamos que al modular la expresión de la RBP MBNL1, el splicing de NUMA1 cambia tal y como habíamos predicho.
Para determinar el impacto funcional asociado al cambio de splicing de NUMA1, diseñamos oligonucleótidos antisentido (AONs) que se unen a la secuencia del exón variable del gen NUMA1 y bloquean su reconocimiento por la maquinaria de splicing. Con estos AONs logramos por tanto inducir en células normales la forma de splicing observada en tumores. Células con este cambio inducido presentaron un incremento en la capacidad proliferativa, indicando que sólo el cambio de splicing puede cambiar las propiedades celulares.
Ya que la función del gen NUMA1 está relacionada con la formación de estructuras celulares esenciales para la separación de los cromosomas durante la división celular, en colaboración con el grupo de Miguel Angel Pujana (ICO, Barcelona) llevamos a cabo un ensayo funcional para medir el impacto del cambio de splicing en NUMA1 sobre dichas estructuras. Observamos un incremento de la inestabilidad genómica asociada a la duplicación de centriolos en células con el cambio de splicing tumoral de NUMA1 en comparación con células de control en las que NUMA1 no tiene ese cambio. Es importante remarcar que en este trabajo también mostramos que los efectos observados no estaban ligados a un cambio de expresión del gen NUMA1. Es decir, el impacto funcional es específico del cambio de splicing, independientemente de la expresión total del gen. Tampoco observamos que ninguna de las muestras analizadas contenía mutación alguna que pudiera explicar los cambios de splicing en NUMA1. Por lo tanto, el cambio de splicing de NUMA1 representa un nuevo caso de alteración del transcriptoma que contribuye a la transformación oncogénica y que sin embargo no son visibles a muchos de los análisis que se llevan a cabo actualmente con datos genómicos de cáncer. Nuestro análisis también describe un nuevo rol en cáncer para muchas RBPs, y en particular para MBNL1, y nos permite aventurar que puede haber muchas más alteraciones relevantes en cáncer por descubrir y que están relacionas con el splicing.
Desafíos actuales en el estudio del splicing alternativo en cáncer
Aunque los resultados obtenidos son muy prometedores, el estudio del splicing alternativo en cáncer no está exento de múltiples dificultades. Muchos genes presentan variabilidad en sus patrones de splicing entre individuos (Sebestyén et al. 2015). Aunque dicha variación individual podría ser considerada neutra en lo que respecta a la relación con estados patológicos, tampoco está claro si podría estar relacionada con riesgo a enfermedad. Por otro lado, muchas de las alteraciones del splicing detectadas en tumores no son de gran magnitud, y aquellas para las que se ha mostrado su relevancia, no tienen lugar en un gran número de pacientes, tal y como ocurre con las alteraciones genéticas. Una complejidad añadida consiste en la discrepancia que suelen mostrar las distintas herramientas computacionales dedicadas al estudio del splicing alternativo, principalmente para genes que presentan baja expresión, por lo que es difícil definir un consenso sobre cuáles son los cambios relevantes y reproducibles sólo a partir del análisis de los datos de secuenciación. Todo esto hace que la identificación de las variaciones del transcriptoma asociadas a enfermedad sean aún una cuestión por resolver.
A pesar de estas dificultades, las tecnologías de secuenciación están mejorando a gran velocidad y permiten medir el transcriptoma cada vez a mayor profundidad, e incluso en una sola célula, por lo que la precisión en la caracterización del splicing irá mejorando con el tiempo. También existen nuevas tecnologías capaces de secuenciar moléculas de ARN completas. Aunque dichas técnicas son aún caras y están limitadas a moléculas abundantes y de menor tamaño, pueden servir para determinar el transcriptoma en diferentes células tumorales. Nuevas técnicas computacionales también permiten identificar de forma efectiva las asociaciones entre las variaciones del transcriptoma y las variaciones de la secuencia genómica en regiones no codificantes por proteína, lo cual permitirá descubrir nuevos mecanismos que contribuyan al cáncer, y en especial en tumores pan-negativos. Por último, la caracterización de las alteraciones del splicing en cohortes de pacientes con seguimiento clínico ayudaría a definir de forma concluyente el valor pronóstico y terapéutico del splicing. Los cambios con relevancia clínica podrían usarse en el diseño de estudios de cribado, permitiendo así la incorporación del estudio del splicing alternativo en las estrategias actuales de genómica personalizada de cáncer.
Referencias
Bechara EG, et al. RBM5, 6, and 10 differentially regulate NUMB alternative splicing to control cancer cell proliferation. Mol Cell. 2013 Dec 12;52(5):720-33. doi: 10.1016/j.molcel.2013.11.010.
Brooks AN, et al. A pan-cancer analysis of transcriptome changes associated with somatic mutations in U2AF1 reveals commonly altered splicing events. PLoS One. 2014 Jan 31;9(1):e87361. doi: 10.1371/journal.pone.0087361.
Frampton GM, et. Activation of MET via diverse exon 14 splicing alterations occurs in multiple tumor types and confers clinical sensitivity to MET inhibitors. Cancer Discov. 2015 Aug;5(8):850-9. doi: 10.1158/2159-8290.CD-15-0285.
Han H, et al. MBNL proteins repress ES-cell-specific alternative splicing and reprogramming. Nature. 2013 Jun 13;498(7453):241-5. doi: 10.1038/nature12270
Karni R, et al. The gene encoding the splicing factor SF2/ASF is a proto-oncogene. Nat Struct Mol Biol. 2007 Mar;14(3):185-93.
Ma PC, et al. c-MET mutational analysis in small cell lung cancer: novel juxtamembrane domain mutations regulating cytoskeletal functions. Cancer Res. 2003 Oct 1;63(19):6272-81.
Misquitta-Ali CM, et al. Global profiling and molecular characterization of alternative splicing events misregulated in lung cancer. Mol Cell Biol. 2011 Jan;31(1):138-50. doi:10.1128/MCB.00709-10.
Paik PK, et al. Response to MET inhibitors in patients with stage IV lung adenocarcinomas harboring MET mutations causing exon 14 skipping. Cancer Discov. 2015 Aug;5(8):842-9. doi: 10.1158/2159-8290.CD-14-1467. Epub 2015 May 13. Erratum in: Cancer Discov. 2016 Mar;6(3):330.
Poulikakos PI, et al. RAF inhibitor resistance is mediated by dimerization of aberrantly spliced BRAF(V600E). Nature. 2011 Nov 23;480(7377):387-90. doi:10.1038/nature10662.
Salton M, et al. Inhibition of vemurafenib-resistant melanoma by interference with pre-mRNA splicing. Nat Commun. 2015 May 14;6:7103. doi: 10.1038/ncomms8103
Sebestyén E, et al. Detection of recurrent alternative splicing switches in tumor samples reveals novel signatures of cancer. Nucleic Acids Res. 2015 Feb 18;43(3):1345-56. doi: 10.1093/nar/gku1392
Sebestyén E, et al. Large-scale analysis of genome and transcriptome alterations in multiple tumors unveils novel cancer-relevant splicing networks. Genome Res. 2016 Jun;26(6):732-44. doi: 10.1101/gr.199935.115.
Sotillo E, et al. Convergence of Acquired Mutations and Alternative Splicing of CD19 Enables Resistance to CART-19 Immunotherapy. Cancer Discov. 2015Dec;5(12):1282-95. doi: 10.1158/2159-8290.CD-15-1020.
Wang Y, et al. The splicing factor RBM4 controls apoptosis, proliferation, and migration to suppress tumor progression. Cancer Cell. 2014 Sep 8;26(3):374-89. doi: 10.1016/j.ccr.2014.07.010.
Yoshida K, et al. Frequent pathway mutations of splicing machinery in myelodysplasia. Nature. 2011 Sep 11;478(7367):64-9. doi: 10.1038/nature10496.
Zong FY, et al. The RNA-binding protein QKI suppresses cancer-associated aberrant splicing. PLoS Genet. 2014 Apr 10;10(4):e1004289. doi: 10.1371/journal.pgen.1004289.

