Amparo Tolosa, Genotipia

Felipe Javier Chaves (Cuenca 1969) es doctor en Biología y experto en diagnóstico de enfermedades hereditarias e investigación en genética humana. En la actualidad, Chaves es director de la Unidad de Genómica y Diabetes del Instituto de Investigación Sanitaria del Hospital Clínico de Valencia (INCLIVA), donde investiga las bases genéticas de diferentes enfermedades complejas y desarrolla protocolos de diagnóstico genético. Además, desde 2013 es uno de los fundadores de Sequencing Multiplex SL (I+d+I, Seqplexing), Spin-off del INCLIVA de apoyo a la investigación y práctica clínica.
Chaves es el investigador principal del proyecto ‘Detección y caracterización rápida de COVID-19 y del paciente’, que recientemente ha recibido financiación del Instituto de Salud Carlos III (ISCIII). El proyecto, presentado en el marco de la convocatoria del ISCIII destinada a impulsar propuestas que puedan ser implementadas de forma temprana en el Sistema Nacional de Salud, está centrado en el desarrollo métodos de detección rápida de la infección por SARS-CoV-2, así como en obtener información genética del virus que pueda contribuir a estimar su virulencia.
Hemos hablado con el Dr. Chaves para conocer mejor este proyecto así como sus posibles aplicaciones en el control de la pandemia de COVID-19.
Acabáis de recibir financiación para un proyecto de investigación sobre la detección de SARS-COV-2 y la enfermedad COVID-19. ¿Cuál es el objetivo de este proyecto?
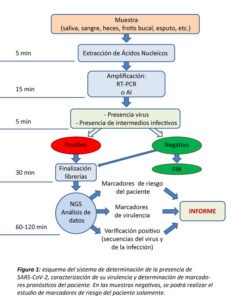
El proyecto consiste en dos fases, una primera fase es desarrollar métodos rápidos para detectar el virus mediante técnicas de biología molecular y luego desarrollar procedimientos que, permitan caracterizar la secuencia del virus para ver qué tipo de cepa. También se pueden identificar factores humanos que den resistencia a la infección o que vayan a predecir una mala o buena evolución de la infección. Lo urgente ahora es detectar el virus. Pero dentro de un tiempo será importante saber las cepas que tiene cada individuo y si es más susceptible o no a pasar la enfermedad.
¿Qué características tienen las pruebas de detección que estáis desarrollando?
Por un lado queremos hacer test muy rápidos de PCR que puedan proporcionar resultados en 15 minutos. También utilizamos amplificación isoterma, con una modificación diseñada desde Sequencing Multiplex SL, que tiene mayor capacidad de amplificación y lo hace en menos tiempo que otros protocolos existentes, que tardan más de 15 minutos en hacer la detección y tienen una sensibilidad más baja.
¿En cuánto tiempo podrían estar disponibles estas pruebas?
Yo espero que las primeras validaciones estén en un mes y a partir de ahí se puedan empezar a utilizar.
¿Es necesario el mismo equipamiento que requiere una PCR habitual?
Las pruebas están pensadas para que la PCR se haga a nivel de laboratorio, por ejemplo en un hospital. Pero por otro lado, la amplificación isoterma está pensada para que se pueda hacer en cualquier lugar. Simplemente se necesita un baño de agua o una estufa que se pueda mantener a temperatura determinada. Con un equipamiento mínimo y una aplicación, o a simple vista, se puede determinar quién es portador y quién no.
¿Qué tipo de muestra será necesaria para la prueba de detección?
Una torunda bucal o nasofaríngea permitiría detectar el virus. Nosotros vamos a intentar que no haya que hacer extracción de ARN, para ahorrar tiempo, y que se manipule lo menos posible la muestra. Las torundas que ya se están utilizando en algunos hospitales inactivan el virus, por lo que hay un riesgo mucho menor para el tema de manipulación y transporte.
En vuestro laboratorio también desarrolláis protocolos de diagnóstico de enfermedades o estudios farmacogenéticos. ¿Hay una elevada demanda de este tipo de pruebas?
Actualmente hay una gran demanda de estos estudios, aunque principalmente se centran en el diagnóstico de enfermedades hereditarias. Los estudios farmacogenéticos y farmacogenómicos tradicionales se tendrán que imponer antes o después dado que determinan la efectividad de los tratamientos y se evita la pérdida de tiempo probando diferentes fármacos que desde un principio podríamos saber su ineficiencia o la presencia de efectos secundarios. Por otro lado, la medicina de precisión o individualizada que se aplica en oncología podría incluirse en la farmacogenética, dado que diferentes mutaciones del tumor determinan la respuesta al tratamiento.
Los procedimientos que estáis desarrollando ¿podrían aplicarse a otros estudios?
Estos desarrollos serán aplicables a diferentes tipos de virus e incluso a la detección de diferentes infecciones simultáneamente.
En relación a los test rápidos, en Europa o España no estamos acostumbrados a que se hagan diagnósticos de una infección por un virus en el mismo sitio donde está el paciente. En otros países, sobre todo en países tropicales, donde hay infecciones víricas endémicas o en el caso de la malaria, sí puede ser muy útil hacer la determinación del agente infectivo donde se encuentra el propio paciente. Esto se haría mediante los test rápidos basados en amplificación isoterma.
Ahora mismo, con COVID-19 hay una gran necesidad de hacer este tipo de test. Sería muy interesante poder determinar quién tiene el virus para poder aislarlo o reconocerlo y tomar medidas de prevención. En el futuro, seguramente podrán existir otras pandemias y será muy útil disponer de estas herramientas.
¿Qué perspectivas tenéis para la prueba que estáis desarrollando?
Una vez tengamos el procedimiento validado se podrán hacer miles de test. Por ejemplo, por PCR se podrán entre 5 000 y 10 000 test en un día en un laboratorio que requerirá poco equipamiento y poco personal. Se pueden distribuir test también a otros centros de salud donde pueda haber personas con elevado riesgo a estar infectadas y hacerse una gran cantidad de test.
¿Se podrán hacer estudios a gran escala de detección de infectados?
Sí, una aplicación sería hacer un estudio masivo de población en riesgo para conocer quien es portador y tomar las medidas de aislamiento oportunas. Además, una persona puede haber pasado la enfermedad, los síntomas, y seguir teniendo virus, así como potencial para transmitirlos. Esto hay que controlarlo para saber quién tiene carga vírica y quién no. Incluso antes de los síntomas hay una carga viral importante.
La pandemia de COVID-19 ha tenido un gran impacto en muchos aspectos, incluida la investigación. Además de desarrollar protocolos de diagnóstico en vuestro laboratorio estudiáis la diabetes tipo 2 y otras enfermedades metabólicas con alto riesgo cardiovascular. ¿Cómo está afectando COVID-19 a la investigación de esas áreas?
Ahora mismo los laboratorios están prácticamente parados y estamos todos trabajando desde casa, analizando datos, preparando proyectos… La mayoría de los centros han cerrado y están todos teletrabajando. Esto, por una parte se va a aprovechar para estudiar los datos ya obtenidos, pero por otra parte implica que se están generando menos resultados en los casos que se precisa ir al laboratorio para generarlos. Solamente estamos trabajando aquellos grupos que tenemos proyectos en relación a COVID19.
¿Tenéis pensado algún otro proyecto relacionado con COVID-19?
Estamos pensando alguno, sobre todo para llenar los vacíos de información que existen, como por ejemplo la virulencia o patogenicidad de las cepas de virus y los factores genéticos de los pacientes, así como relacionar información de los virus con la de los pacientes.
Tenéis experiencia en identificar predisposiciones genéticas a desarrollar enfermedades, ¿qué aproximaciones pueden plantearse para identificar una susceptibilidad genética al virus?
Lo lógico sería estudiar el genoma de las personas infectadas que han desarrollado enfermedad grave, personas infectadas que no han manifestado la enfermedad y personas que han estado expuestas pero que no han sido infectadas. Además, habría que secuenciar el virus en cada caso para ver si el resultado final depende de la cepa o de la genética o de otros factores de cada individuo. Los estudios genéticos podrían dar información sobre quién se infecta y por qué, o quién no se infecta o responde muy bien a la infección. Hay varias aproximaciones y muchas formas de desarrollar la investigación.
Por ejemplo, se conocen algunas proteínas que son imprescindibles para que el virus nos infecte. Pero la variabilidad de los genes de estas proteínas es escasa en relación a su secuencia de aminoácidos y no explicaría las diferencias que se observan entre los pacientes. El estudio de las bases genéticas en relación a la respuesta frente al virus también podría ser una forma de encontrar dianas terapéuticas. Si una persona tiene hiperactiva o inactiva una proteína y esto le protege, se pueden buscar fórmulas mediante fármacos u otros métodos para actuar sobre esas proteínas.
Si te ha gustado esta noticia y quieres aprender más sobre Genética en Medicina, te interesan nuestros cursos y formación universitaria.

