Amparo Tolosa, Genotipia
Una colección de 14 nuevos artículos publicados en Nature, Nature Methods y Nature Communications muestran los últimos resultados de la tercera fase del proyecto ENCODE, destinado a catalogar y analizar los elementos reguladores del genoma.
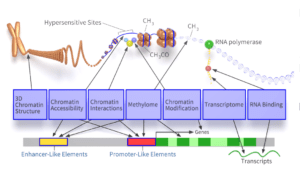
Tan importante es conocer qué genes hay en el genoma y qué proteína o elemento funcional codifican, como entender cómo se regula su expresión en cada célula o tejido. Desde su creación en el año 2003, el proyecto ENCODE (Enciclopedia de Elementos de ADN, en sus siglas en inglés) cataloga los elementos funcionales del genoma humano (entre otros) e investiga su papel en la regulación de la expresión génica y la salud.
Los objetivos principales de ENCODE han ido avanzando con los años. El proyecto piloto, por ejemplo, estaba centrado en encontrar las regiones del ADN que se transcriben a RNA e identificar las regiones reguladoras de la expresión, en un 1% del genoma humano. Posteriormente, la fase 2 amplió el análisis a todo el genoma y transcriptoma y comenzó a incluir análisis de metilación y modificación de histonas.
La fase 3 de ENCODE, que se presenta ahora, tiene varias novedades. Por una parte, incorpora información sobre nuevos tipos de ensayos destinados a evaluar la funcionalidad de los elementos del genoma, relacionados con la unión al ARN o la organización tridimensional de la cromatina. Por otra, amplia significativamente el número de muestras biológicas analizadas, incorporando nuevos tipos celulares y tejidos. Concretamente, esta fase incluye 503 tipos de células o tejidos, de más de 13 069 muestras biológicas.
Los experimentos realizados y los datos generales obtenidos de de ENCODE 3 aparecen resumidos en el primero de los artículos, dirigido por Zhiping Weng. En él se exponen grosso modo los 5 992 nuevos conjuntos de datos experimentales generados, que han permitido registrar 929 535 elementos reguladores candidatos en el genoma humano y 339 815 en el de ratón, que representan el 7.9% y el 3.4% de los genomas de cada especie.
Los otros ocho nuevos artículos evalúan resultados concretos del proyecto. Por ejemplo, tres artículos están relacionados con el impacto de la variación genética en la regulación del genoma y otro más analiza el potencial de la base de datos ENCODE como recurso para la genómica del cáncer. Tres trabajos más evalúan los elementos reguladores que intervienen en el desarrollo prenatal de mamíferos, para lo que se ha analizado el genoma funcional de ratón a través de ocho etapas de su desarrollo.
Los nuevos artículos exponen, bajo diferentes aproximaciones, la utilidad de ENCODE para empezar a entender cómo funciona tanto el genoma humano como el de otros organismos modelo estudiados. En la actualidad, el proyecto ENCODE representa una fuente de conocimiento sobre el genoma humano con importantes aplicaciones en el ámbito de la medicina. Por ejemplo, el ENCODE incluye variaciones en la expresión de genes relacionadas con la aparición de ciertas enfermedades humanas.
La terminación de la fase 3 de ENCODE representa el inicio de una nueva fase, el ENCODE 4, destinada a expandir la enciclopedia de elementos reguladores del genoma a través de un abanico más amplio de muestras y la utilización de nuevas tecnologías.
Referencias:
The ENCODE Project Consortium. Expanded encyclopaedias of DNA elements in the human and mouse genomes. Nature. 2020. DOI: http://dx.doi.org/10.1038/s41586-020-2493-4
The ENCODE Project Consortium . Perspectives on ENCODE. Nature. 2020. DOI: http://dx.doi.org/10.1038/s41586-020-2449-8
Van Nostrand EL, et al. A large-scale binding and functional map of human RNA-binding proteins. Nature. 2020. DOI: http://dx.doi.org/10.1038/s41586-020-2077-3
Grubert F, et al. Landscape of cohesin-mediated chromatin loops in the human genome. Nature. 2020. DOI: http://dx.doi.org/10.1038/s41586-020-2151-x
Partridge EC, et al. Occupancy maps of 208 chromatinassociated proteins in one human cell type. Nature. 2020. DOI: http://dx.doi.org/10.1038/s41586-020-2023-4
Gorkin DU, et al. An atlas of dynamic chromatin landscapes in mouse fetal development. Nature. 2020. DOI: http://dx.doi.org/10.1038/s41586-020-2093-3
He Y, et al. Spatiotemporal DNA methylome dynamics of the developing mouse fetus. Nature. 2020. DOI: http://dx.doi.org/10.1038/s41586-020-2119-x
He P, et al. The changing mouse embryo transcriptome at whole tissue and single-cell resolution. Nature. 2020. DOI: http://dx.doi.org/10.1038/s41586-020-2536-x
Vierstra J, et al. Global reference mapping of human transcription factor footprints. Nature. 2020. DOI: http://dx.doi.org/10.1038/s41586-020-2528-x
Meuleman W, et al. Index and biological spectrum of human DNase I hypersensitive sites. Nature. 2020. Doi: http://dx.doi.org/10.1038/s41586-020-2559-3
Zhang J, et al. An integrative ENCODE resource for cancer genomics. Nat Comm. 2020. DOI: http://dx.doi.org/10.1038/s41467-020-14743-w
Sisu C, et al. Transcriptional activity and strain-specific history of mouse pseudogenes. Nat Comm. 2020. DOI: http://dx.doi.org/10.1038/s41467-020-17157-w
Javed N, et al. Detecting sample swaps in diverse NGS data types using linkage disequilibrium. Nat Comm. 2020. DOI: http://dx.doi.org/10.1038/s41467-020-17453-5
Sethi A, et al. Supervised enhancer prediction with epigenetic-pattern recognition and targeted validation. Nat Methods. 2020. DOI: http://dx.doi.org/10.1038/s41592-020-0907-8
ENCODED expanded. Hon CC y Carninci P. Nature. 2020.DOI: https://doi.org/10.1038/s41586-020-2449-8
Si te ha gustado esta noticia y quieres aprender más sobre Genética en Medicina, te interesan nuestros cursos y formación universitaria.

