Autores: Beatriz Paumard-Hernández, Julia Berges-Soria, Eva Barroso, Carlos I Rivera-Pedroza, Virginia Pérez-Carrizosa, Sara Benito-Sanz, Eva López-Messa, Fernando Santos, Ignacio I García-Recuero, Ana Romance, María Juliana Ballesta-Martínez, Vanesa López-González, Ángel Campos-Barros, Jaime Cruz, Encarna Guillén-Navarro, Jaime Sánchez del Pozo, Pablo Lapunzina, Sixto García-Miñaur, Karen E Heath.
La craneosinostosis se define como un defecto congénito que se origina por la prematura fusión de las suturas craneales. Se caracteriza por la falta de crecimiento en la dirección de las suturas fusionadas que se ve compensado con el sobrecrecimiento en perpendicular de otra de las suturas no fusionadas. Este proceso se suele producir por un fallo en el sistema de señalización que rige los procesos de crecimiento y diferenciación de los márgenes de sutura. La craneosinostosis es una enfermedad rara, presenta una incidencia de 1 entre 2100 y 2500 nacimientos y está clasificada como sindrómica o no-sindrómica en función del tipo de suturas que esté afectada.
La mayoría de los genes asociados a craneosinostosis pertenecen a la familia de los FGFR (FGFR1, FGFR2, FGFR3) que codifican los receptores de factores de crecimiento fibroblástico y están asociados con la familia de receptores tirosina kinasa. Modulan una gran diversidad de procesos biológicos dentro de la embriogénesis, homeostasis de los tejidos, mitogénesis, diferenciación, apoptosis y migración. Otros genes implicados en el craneosinostosis son los genes TWIST1 y EFNB1. Mutaciones en el gen TWIST1 se asocian con el síndrome de Saethre-Chotzen (SCS) mientras que mutaciones en EFNB1, localizado en el cromosoma X, se asocian con el síndrome craneofrontonasal Al contrario que la mayoría de las enfermedades ligadas al cromosoma X, esta afecta de manera más severa a las mujeres. En el año 2010, Wilkie y colaboradores, presentaron una cohorte prospectiva de pacientes con craneosinostosis (n=326), donde se identificó el defecto molecular de 84 de ellos, el 86% eran alteraciones monogénicas (FGFR2>FGFR3>TWIST1>EFNB1) y el 14% anomalías cromosómicas.
Recientemente se han identificado mutaciones en el gen TCF12 en pacientes con sinostosis coronal y muchos con SCS pero sin mutaciones en el gen TWIST1. TCF12 codifica un factor de transcripción de tipo hélice-giro-hélice (bHLH). Se expresa en diversos tejidos y participa en la regulación de la expresión génica, mediante la formación de heterodímeros junto a otras proteínas de la familia E de tipo bHLH, como TWIST1. Otro gen recientemente implicado en esta enfermedad es el ERF, codifica el factor de transcripción ERF (¨Ets2 repressor factor¨). Se han identificado mutaciones en este gen asociadas a un tipo de craneosinostosis múltiple.
El objetivo de nuestro proyecto era mejorar el diagnóstico genético de la enfermedad de craneosinostosis en la población española. El diagnóstico genético y molecular de craneosinostosis es muy importante ya que no es fácil diferenciar entre los distintos síndromes debido a que los pacientes comparten ciertas características fenotípicas, todas ellas de expresión muy variable, por lo que muchos pacientes no tienen un diagnóstico clínico definido. Se basa en el cribado mutacional de los genes implicados en craneosinostosis mediante un sistema de cuatro niveles que se realizó en 182 probandos. Se halló diagnóstico molecular de 113 (62%) pacientes; 104 (92%) detectados en nivel 1; ocho (7%) en nivel 2 y uno (1%) en nivel 3, confirmando el diagnóstico clínico previo de 93 (82%) y corrigiendo el diagnóstico clínico de 20 (18%). Durante el desarrollo del proyecto, dos nuevos genes fueron descritos para la enfermedad, TCF12 y ERF. Estudiamos ambos genes y también otra región genómica en los pacientes que tras realizarles el cribado mutacional de los cuatro niveles, carecían de diagnóstico genético. Identificamos una duplicación de la región reguladora de IHH (¨Indian hedgehog¨) en un probando con craneosinstosis tipo Philadelphia y mutaciones patogénicas en TCF12 en cinco (2.7%) de los pacientes que presentaban sinostosis coronal unilateral ó bilateral. Cuatro de estos cinco pacientes fueron diagnosticados inicialmente con SCS, mientras que el quinto fue diagnosticado con el síndrome de Muenke. De 19 individuos con SCS 15 pacientes (79%) presentaban una variante en TWIST1, y cuatro (21%) tenían una variante TCF12. Por lo que TCF12 debería incorporarse al cribado mutacional de TWIST1 en los casos negativos de SCS y de FGFR3 p.Pro250Arg de Muenke.
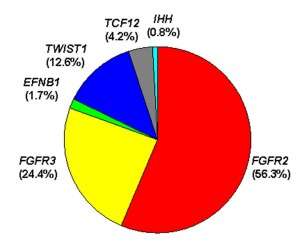
En este sentido, gracias al diagnóstico genético, hemos podido determinar la causa de la patología en un total de 119/182 probandos (65%, Figura 1), lo que permitirá corregir el diagnóstico clínico inicial, realizar una mejor predicción de la intervención quirúrgica, llevar a cabo un seguimiento clínico apropiado y favorecer el consejo genético.
Referencia: Paumard-Hernández B, et al. Expanding the mutation spectrum in 182 Spanish probands with craniosynostosis: identification and characterization of novel TCF12 variants. Eur J Hum Genet. 2014 Oct 1. doi: 10.1038/ejhg.2014.205.
Afiliaciones:
Instituto de Genética Médica y Molecular (INGEMM), Hospital Universitario La Paz, Universidad Autónoma de Madrid, IdiPAZ, Madrid: Beatriz Paumard-Hernández, Julia Berges-Soria, Eva Barroso, Carlos I Rivera-Pedroza, Virginia Pérez-Carrizosa, Sara Benito-Sanz, Eva López-Messa, Fernando Santos, Ángel Campos-Barros, Pablo Lapunzina, Sixto García-Miñaur, Karen E Heath
Centro de Investigación Biomédica en Enfermedades Raras (CIBERER), Instituto Carlos III, Madrid; Eva Barroso, Sara Benito-Sanz, Fernando Santos, Ángel Campos-Barros, María Juliana Ballesta-Martínez, Vanesa López-González, Encarna Guillén-Navarro, Pablo Lapunzina, Sixto García-Miñaur, Karen E Heath
Unidad de Cirugía Maxilofacial, Hospital 12 de Octubre, Madrid: Ignacio I García-Recuero, Ana Romance
Unidad de Genética Médica, Hospital Clínico Universitario Virgen de la Arrixaca, Murcia: María Juliana Ballesta-Martínez, Vanesa López-González, Encarna Guillén-Navarro
Unidad de Dismorfología, Hospital Universitario 12 de Octubre, Madrid: Jaime Cruz, Jaime Sánchez del Pozo
Cátedra de Genética Médica. UCAM-Universidad Católica San Antonio de Murcia, Murcia: Encarna Guillén-Navarro