Leticia Díaz Beltrán1,2,4, Francisco J. Esteban4, Dennis P. Wall1,2,3
1 Division of Systems Medicine, Department of Pediatrics, Stanford University, Stanford, CA, USA
2 Division of Systems Medicine, Department of Psychiatry, Stanford University, Stanford, CA, USA
3 Department of Biomedical Data Science, Stanford University, Stanford, CA, USA
4 Department of Experimental Biology, Experimental Sciences Faculty, University of Jaen, Jaen, Spain
El autismo es considerado un grupo heterogéneo de trastornos del desarrollo cerebral con una elevada prevalencia y que presenta una gran variedad de fenotipos agrupados bajo un término común, el Trastorno del Espectro Autista (TEA). Las características clínicas comunes a este amplio espectro de desórdenes incluyen dificultades en la interacción social, déficits en la comunicación y conductas repetitivas y estereotipadas. Aunque la comunidad científica aún no ha hallado una hipótesis común sobre las causas moleculares del TEA, las investigaciones realizadas sugieren que este desorden es altamente hereditario y que la combinación de factores genéticos, neurológicos, inmunológicos y ambientales desempeñan un papel importante.
Dada la compleja arquitectura genética subyacente al TEA, consideramos que el estudio integrado, mediante biología de sistemas, de la expresión génica detectada en diferentes experimentos puede ser útil para arrojar luz sobre los genes y los procesos biológicos aquí implicados. Así pues, hemos llevado a cabo un análisis comparativo de la expresión génica diferencial de un total de 1232 muestras diferentes (657 de pacientes autistas, 9 de pacientes con retraso mental y 566 de sujetos control), agrupadas a su vez en 27 conjuntos de datos (biosets) de 9 experimentos independientes, y obtenidas a través de la plataforma Nextbio (www.nextbio.com). Nuestro objetivo era comprobar la posible existencia de un patrón en común representativo de autismo y, en caso afirmativo, si éste puede ser detectado tanto en sangre como en cerebro.
Nuestro análisis comenzó con la conversión de las diferentes listas de genes, obtenidas de cada uno de los experimentos, en una matriz binaria de presencia/ausencia de genes. A partir de dicha matriz, y con el fin de determinar el grado de relación existente entre los biosets, realizamos un análisis de agrupamiento (clustering). Como era de esperar, la mayoría de biosets se agruparon en función del experimento y del tipo de tejido del que procedían las muestras. Sin embargo, detectamos un subgrupo (estadísticamente validado) de 5 biosets pertenecientes a 3 experimentos independientes en dos tejidos, sangre y cerebro, que compartían 66 genes. A este grupo de genes comunes los denominamos “Root 66”. Fue interesante comprobar que cuatro de ellos ya habían sido previamente asociados al TEA (MAP1LC3B, PDE4B, TCF4 and UPF2) y que otros 56 o bien tenían descritas interacciones directas con genes candidatos a autismo o estaban implicados en trastornos neurológicos asociados a este desorden. Esto nos hizo pensar que la causa de dicha agrupación podría basarse en los patrones de regulación asociados a los 66 genes.
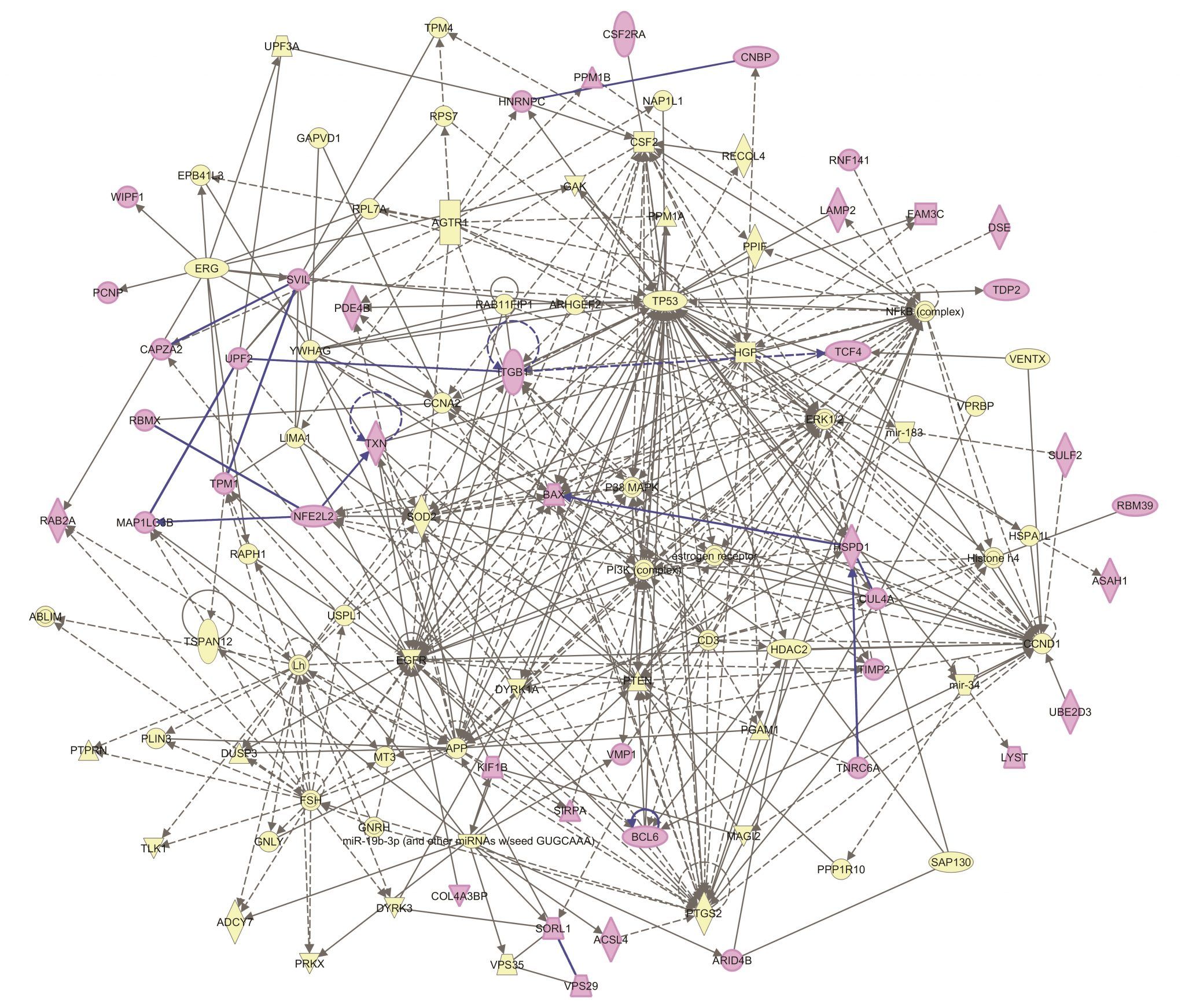
Para evaluar su significación funcional, comprobamos tanto si participaban en procesos biológicos específicos como su conexión (estructura de red funcional). Dicho análisis reveló que 42 de estos genes interactuaban en una densa red cuyas relaciones se basaban en procesos neurológicos tales como la transmisión sináptica, la neurodegeneración, una morfología anormal del cerebro, el aprendizaje y la memoria. Además, algunos “Root 66” estaban implicados en procesos relacionados con una disfunción en la actividad neuroendocrina y neurotransmisora, lo cual se sabe que afecta al comportamiento. En conjunto, todo indicaba que un buen número de estos 66 genes mostraba tener un papel clave en importantes procesos de disfunción neurológica lo que, por ende, apoyaba nuestra hipótesis inicial en cuanto a su relación, de forma relevante, con el TEA.
Para determinar si los “Root 66” constituían una huella única en autismo no encontrada al azar, procedimos a descargar de la misma fuente (Nextbio) listas de genes diferencialmente expresados en 450 experimentos de otras enfermedades no relacionadas y obtuvimos el grado de solapamiento de cada una de ellas con los 66 genes de interés en autismo. Como resultado detectamos que el número medio de genes que solapaban era sólo de seis, lo que apoyaba nuestra tesis de que el grupo cerebro-sangre formado por “Root 66” no fue encontrado por casualidad y representaba un patrón de expresión único en el TEA.
También había que averiguar si en otros análisis de datos de expresión génica, en sangre y cerebro normal, se había reportado algún tipo de agrupamiento de relación entre estos tejidos. Aunque algunos estudios mostraban la existencia de genes expresados tanto en sangre como en cerebro, ningún árbol de agrupamiento reflejó proximidad entre las muestras de estos dos tipos de tejidos. Es más, ambos aparecían siempre como grupos totalmente independientes, resultados muy diferentes (recordamos que llevados a cabo en sujetos sanos) a los de nuestro análisis (en TEA). Por último, comprobamos si los “Root 66” se expresaban de forma constitutiva en cualquier tejido. Para ello intersectamos nuestros 66 candidatos con una lista de 408 genes constitutivos (confirmados experimentalmente) y como resultado solo se obtuvo un solapamiento de 3 genes. Ambas comprobaciones apoyaban la hipótesis de que los 66 genes desempeñan una función única en autismo.
Los experimentos de expresión génica publicados hasta la fecha de manera individual han tenido un impacto relativamente limitado en el esclarecimiento de la etiopatología del autismo. Sin embargo, nuestro estudio pone de manifiesto, mediante un análisis integrador de dichos experimentos, la existencia de una relación estadísticamente significativa y no aleatoria entre sangre y cerebro en el TEA. Dicha relación, sustentada por 66 genes, podría tener una cierta relevancia en la etiología del autismo, dado que la mayoría de sus componentes están asociados a procesos biológicos cruciales para el normal desarrollo y funcionamiento del cerebro, además de estar implicados en otros trastornos neurológicos.
Aunque se trata de resultados preliminares que requieren confirmación a través de su validación experimental en otras poblaciones independientes, nuestro hallazgo sugiere la existencia de una firma biológica detectable en la sangre de individuos con autismo eco de una señal de disfunción neurológica cerebral.
Referencia: Diaz-Beltran L, et al. A common molecular signature in ASD gene expression: following Root 66 to autism. Transl Psychiatry 2016 Jan 5;6:e705. doi:10.1038/tp.2015.112 http://www.nature.com/tp/journal/v6/n1/full/tp2015112a.html