Cristina Mayor-Ruiz1
1 CeMM–Research Center for Molecular Medicine of the Austrian Academy of Sciences, Vienna, Austria
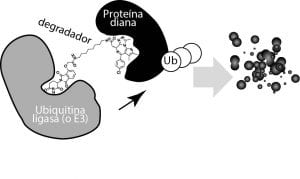
La degradación de proteínas dirigida (DPD) se basa en el uso de moléculas sintéticas llamadas degradadores, para reprogramar nuestra maquinaria de degradación proteica y eliminar proteínas de interés terapéutico. Supone un novedoso enfoque en el desarrollo de fármacos que actualmente representa una estrategia terapéutica muy prometedora, por ejemplo, en cáncer. El concepto es relativamente simple, estos degradadores cuentan con dos partes diferenciadas: una parte se une a una proteína de interés (por ejemplo, una proteína esencial para el crecimiento tumoral), y otra parte que se une a una ubiquitina ligasa o “E3″ (ver Figura) (Sakamoto et al, 2001). Las E3s son parte de la maquinaria implicada en reconocer y etiquetar con ubiquitina ciertas proteínas para su destrucción por el proteosoma, el principal sistema de degradación de proteínas presente en nuestras células. En concreto, la mayoría de los degradadores desarrollados hasta el momento, reclutan E3s de la familia de las Cullin-RING ligasas (CRLs). Las CRLs son complejos proteicos con una regulación muy dinámica que les permite adaptarse rápidamente a los requisitos celulares, y representan la familia más numerosa de ubiquitina ligasas con unos 250 miembros (Deshaies et al, 2009).
Gran parte de los fármacos tradicionales funcionan como inhibidores de proteínas. Para que sean eficientes, en necesario que la estructura de una determinada proteína diana contenga orificios accesibles, lo que limita el espectro de proteínas abordables (tan solo 10-20% del proteoma) (Hopkins et al, 2002). Los degradadores suponen un cambio de paradigma en lo que a desarrollo de fármacos se refiere: no es necesario “inhibir”, basta con “unir”. Esta particularidad, amplía enormemente el espectro de proteínas, incluyendo importantes dianas hasta ahora inaccesibles para el desarrollo terapéutico. Otra característica diferencial es que los degradadores consiguen eliminar proteínas. Esta es una propiedad clave, ya que la DPD no se limita a la inhibición de actividades proteicas individuales, sino que permite interrumpir todas las funciones asociadas a una proteína. Supone el equivalente farmacológico a un ¨knock-out¨ genético.
Estamos empezando a comprender este nuevo tipo de farmacología, mayoritariamente gracias a estudios estructurales sobre la formación de los agregados ternarios característicos de la DPD: E3-degradador-proteína diana (ver Figura) (Gadd et al, 2017). Sin embargo, los efectores celulares que gobiernan la eficacia y sensibilidad a la DPD permanecían inexplorados. Por ello, en el laboratorio del Dr. Georg Winter en el CeMM (Viena), nos propusimos identificar la red genética que determina la eficacia de la DPD. Actualmente se están llevando a cabo los primeros ensayos clínicos con PROTACs (un tipo de degradadores), subrayando la necesidad de identificar y comprender estos determinantes, y así aportar biomarcadores que guíen la estratificación de pacientes.
Con ese objetivo, seleccionamos un conjunto representativo de cinco degradadores que reclutan diferentes CRLs, y degradan proteínas de relevancia clínica como BRD4, CDK9 o GSPT1. Y llevamos a cabo estudios de resistencia. En primer lugar, generamos una colección de células mutantes utilizando la tecnología CRISPR-Cas9 en una línea de leucemia. Cada célula de la colección tenía mutado un gen diferente de nuestro genoma. Tras tratarlas con los degradadores, pudimos aislar células resistentes y posteriormente identificar las mutaciones que portaban. Los resultados nos permitieron identificar ciertos reguladores centrales del ensamblaje de las CRLs como los determinantes clave en la sensibilidad celular a los degradadores. Estos reguladores son, por tanto, imprescindibles para la eficacia de los mismos.
Centramos nuestros esfuerzos en entender por qué dos de estos reguladores eran tan esenciales: la proteína CAND1 (implicada en el intercambio de las proteínas reconocidas y etiquetadas por las CRLs) y el CSN (un complejo proteico conocido como “COP9 signalosome”, cuya función consiste en desactivar las CRLs). Combinando ensayos de genómica funcional y de proteómica cuantitativa, descubrimos que cuando esos reguladores no funcionan correctamente, las ubiquitina ligasas del tipo CRL pierden su capacidad de ensamblarse y desmontarse de manera flexible, comienzan a etiquetarse para su propia destrucción en un proceso llamado auto-degradación y, como consecuencia, los degradadores no pueden eliminar sus proteínas diana y son ineficaces para bloquear el crecimiento de las células tumorales.
Además de identificar la red genética que gobierna la eficacia de los degradadores, exploramos aspectos fundamentales de la regulación y la dinámica de las CRLs, siempre en el contexto de la DPD. Nuestros datos sugieren que la resistencia a la DPD se define por la naturaleza de la CRL que es utilizada, y no por proteína diana eliminada. También aprendimos que este fenómeno de auto-degradación afecta a diferentes CRLs dependiendo del tipo celular. En conjunto, nuestro trabajo destaca la relevancia y las sutilezas de los circuitos reguladores de las Cullin-RING ligasas, demostrando que su adecuado funcionamiento es imprescindible para la eficacia de los degradadores.
Ahora que los primeros degradadores están entrando en la clínica, las redes de genes moduladores que hemos identificado podrían servir como biomarcadores para optimizar su uso racional. Además, los mecanismos de resistencia estudiados pueden informar sobre formas de luchar contra ellos en el futuro. Por último, nuestro estudio arroja luz sobre aspectos fundamentales de la regulación y dinámica de nuestra maquinaria de degradación de proteínas. Es importante recalcar que la DPD hace uso de sistemas propios de nuestras células que redirigimos para degradar proteínas implicadas en diferentes enfermedades, como el cáncer. Se trata, por tanto, de sistemas que han evolucionado de forma natural para ser tremendamente eficientes. Tiene sentido que, si aprendemos a reprogramar estos mecanismos tan eficientes mediante el uso de degradadores, los resultados puedan ser reamente transformadores. Por eso es tan necesario profundizar en el estudio de las intricadas redes que gobiernan la degradación de proteínas, porque ese conocimiento supone la base para guiar el desarrollo de fármacos efectivos.
Referencia: Mayor-Ruiz, C. et al. Plasticity of the Cullin-RING ligase repertoire shapes sensitivity to ligand-induced protein degradation. Mol Cell . 2019, 75, 849–858. Doi: https://doi.org/10.1016/j.molcel.2019.07.013
Bibliografía:
Deshaies, R.J. & Joazeiro C.A.P. RING domain E3 ubiquitin ligases. Annu Rev Biochem. 2009. 78, 399-434.
Fischer, E.S. et al. The molecular basis of CRL4DDB2/CSA ubiquitin ligase architecture, targeting, and activation. Cell. 2011, 147, 1024-39.
Gadd, M.S. et al. Structural basis of PROTAC cooperative recognition for selective protein degradation. Nat Chem Biol, 2017, 13, 514-521.
Hopkins, A.L. & Groom, C.R. The druggable genome. Nat Rev Drug Discov, 2002, 1, 727-30.
Sakamoto, K.M. et al. Protacs: chimeric molecules that target proteins to the Skp1-Cullin-F box complex for ubiquitination and degradation. Proc Natl Acad Sci U S A, 2001, 98, 8554-9.
Si te ha gustado esta noticia y quieres aprender más sobre Genética en Medicina, quizás te interesen nuestros cursos. Tienes más información aquí.


