Amparo Tolosa, Genetica Médica News
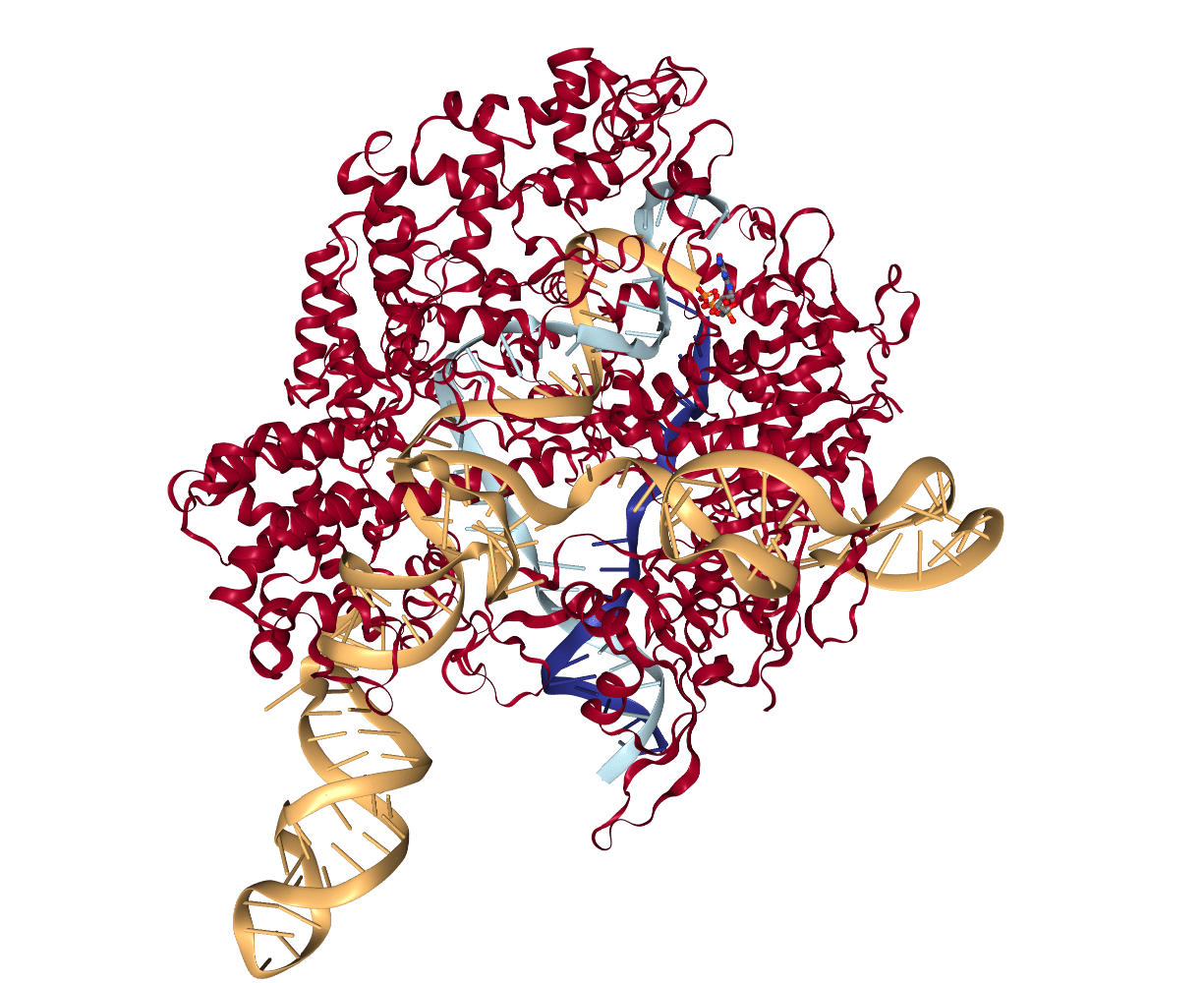
Investigadores del Whitehead Institute han conseguido modificar mediante la tecnología CRISPR el estado de metilación del ADN in vivo, consiguiendo activar genes específicos y convirtiendo fibroblastos de la piel en células musculares. El trabajo amplia el potencial de la técnica CRISPR hacia la modificación no sólo de la secuencia de ADN sino también a la regulación epigenética de la expresión génica.
La metilación del ADN constituye uno de los principales mecanismos epigenéticos con los que la célula puede controlar la expresión de genes sin modificar la secuencia del ADN. Consiste en la adición de grupos metilo en posiciones concretas del ADN, los cuales actúan como señales para la maquinaria proteica encargada de que los genes se transcriban a ARN. Normalmente, la metilación de las regiones reguladoras de los genes suele asociarse a una represión de la expresión. Además, la metilación a través de su acción directa sobre ciertas secuencias del ADN o a través de la interacción con otros mecanismos epigenéticos también contribuye a la arquitectura del genoma.
El sistema CRISPR utilizado para editar el genoma dispone de dos componentes principales, una enzima nucleasa que corta el ADN y un ARN guía que dirige la enzima hacia una posición concreta del genoma. Por complementariedad, el ARN localiza y se une al ADN, lo que hace la enzima quede colocada en posición correcta para cortar. Posteriormente, los mecanismos de reparación del ADN reparan el daño producido, pero en algunos casos, si se producen errores, se introduce un cambio, quedando modificado el ADN.
En el trabajo, los investigadores utilizaron un sistema CRISPR en el que la enzima nucleasa estaba inactivada y añadieron un módulo proteico con actividad metilasa (Dnmt3a), o desmetilasa (Tet1). Así, en la posición marcada por el ARN guía, en lugar de introducirse un corte en el ADN lo que se produciría sería la adición o eliminación de grupos metilo.
En un modelo celular en ratón, y fusionando Tet1 a los componentes del sistema CRISPR, los investigadores consiguieron convertir fibroblastos de la piel en células musculares. En este caso, utilizaron como diana una región reguladora del gen MyoD metilada en los fibroblastos. Al eliminar la metilación y facilitar la activación del gen, los fibroblastos fueron reprogramados en mioblastos. Con el mismo sistema CRISPR-Tet1 y utilizando como diana el promotor del gen BDNF, el equipo consiguió también activar la expresión de este gen en neuronas postmitóticas, activación que en situación normal es producida por medio de la actividad neuronal.
Los investigadores también comprobaron la efectividad de utilizar una enzima metilasa como parte del sistema CRISPR. En este caso, fusionaron Dnmt3a a la nucleasa inactiva y dirigieron el ARN guía hacia secuencias del ADN críticas para la organización tridimensional del genoma. Al añadir los grupos metilo en dichas posiciones, se evitó la formación de ciertos lazos en el ADN lo que afectó a la expresión de los genes cercanos.
“Creo que esta es una herramienta importante para estudiar el estado epigenético, el estado de metilación, de las secuencias reguladoras que controlan la expresión génica,” señala Rudolf Jaenisch, director del trabajo. “La epigenética es muy importante para los estados patológicos. Por lo tanto, ser capaz de cambiar el estado de metilación de un gen o una secuencia reguladora in vivo es importante.”
Por último, el equipo comprobó el funcionamiento del sistema en un modelo de ratón in vivo en el desmetilaron el promotor de un gen y activaron su expresión en las neuronas de la región cerebral del hipocampo.
“Este sistema es muy versátil,” señala Shawn Liu, investigador postdoctoral en el laboratorio de Jaenish y autor del trabajo. “Puede ser utilizado para desmetilar o metilar muchas regiones en el genoma de mamíferos, por lo que podemos estudiar su significado biológico o patológico.”
Los resultados del trabajo ofrecen un marco experimental con gran potencial para el estudio de la metilación del genoma, tanto en modelos celulares como en animales in vivo. Además, dado que la alteración de los patrones de metilación es una marca característica de algunas enfermedades humanas, el sistema podría –a muy largo plazo- tener valor terapéutico.
“Pensamos que este sistema podría suponer un cambio crítico para estudiar la metilación del ADN en el genoma y también controlar la expresión génica de forma dirigida,” añade Hao Wu, coautor del trabajo. “Además, pensamos que podría ser una buena herramienta para aproximaciones terapéuticas en el futuro.”
Investigación original: Liu XS, et al. Editing DNA Methylation in the Mammalian Genome. Cell. 2016 Sep 22;167(1):233-247.e17. doi: 10.1016/j.cell.2016.08.056.
Fuente: Scientists use CRISPR/CAS9 to flip DNA methylation states in vivo. http://wi.mit.edu/news/archive/2016/scientists-use-crisprcas9-flip-dna-methylation-states-vivo