Amaia Carrión-Castillo, Basque Center on Cognition, Brain and Language (BCBL)
La dislexia es una condición caracterizada por dificultades en la adquisición de la lectura, que no se explican por la falta de oportunidades educativas adecuadas, por la falta de instrucción convencional o por otras dificultades neurológicas. La dislexia tiene una etiología compleja que incluye factores tanto genéticos como ambientales. Presenta agrupación familiar: la probabilidad de tener dislexia es mayor si tiene un familiar en primer grado que tenga dislexia. Efectivamente, se trata de una condición heredable, con estimaciones de heredabilidad entre 0.3 y 0.8 (Peterson y Pennington, 2015).
Dada la prevalencia de entre 5-10% en la población general, resulta extraordinario que en algunas familias la dislexia sea un rasgo presente en hasta la mitad de sus miembros. Estas familias sugieren un patrón hereditario mendeliano, en el que una o pocas variantes genéticas raras con un alto grado de penetrancia pudieran contribuir al fenotipo. El estudio de estas familias excepcionales ha mostrado ser una estrategia útil para la identificación de varios de los genes candidatos de la dislexia. Por ejemplo, se encontró que un haplotipo raro en ROBO1 cosegregaba con dislexia en la gran mayoría de los individuos de una familia multigeneracional, y que el mismo gen estaba afectado en una translocación de otro individuo no relacionado (Hannula-Jouppi et al. 2005). Otro gen candidato de la dislexia, DYX1C1, se identificó porque estaba afectado en una reorganización cromosómica que cosegregaba en múltiples miembros de otra familia (Taipale et al. 2003).
En la última década, la tecnología Next-generation sequencing (NGS) ha revolucionado el análisis genético de las enfermedades humanas, ya que permite secuenciar genomas y exomas de manera masiva, y realizar un análisis sistemático y rápido de las mutaciones raras y comunes de un individuo. La NGS también ha permitido estudiar a otro nivel a estas familias que presentan una alta frecuencia de dislexia. Por ejemplo, un estudio que realizó secuenciación total del exoma (WES por sus siglas en inglés, Whole Exome Sequencing) en una familia sueca extensa identificó una mutación de dos bases que resulta en un cambio de aminoácido en el gen CEP63 que cosegregaba parcialmente con el fenotipo (Einarsdottir et al. 2015). Otro estudio de ligamiento realizó WES y secuenciación total del genoma (WGS por sus siglas en inglés, Whole Genome Sequencing) en dos individuos críticos e identificó una mutación no-sinónima en el gen NCAN (Einarsdottir et al. 2017).
Un nuevo estudio publicado recientemente en la revista Human Genetics (Carrión-Castillo et al. 2021), ha identificado variación en el gen SEMA3C que cosegrega con la dislexia en una familia multigeneracional. Este trabajo liderado por la Dra. Amaia Carrión-Castillo y el Dr. Simon E. Fisher se realizó en el Instituto Max Planck de Psicolingüística junto a colaboradores de la Universidad Radboud y la Universidad de Groningen (Países Bajos).
En este estudio se combinó el diseño de familia multigeneracional y de la tecnología WGS para estudiar una familia holandesa con una alta frecuencia de dislexia. En primer lugar, se realizó un análisis de ligamiento que permitió identificar una región en el cromosoma 7q21.11 que cosegregaba con el fenotipo de dislexia de manera imperfecta en esta familia (valor LOD de 2.83), con un haplotipo compartido por 11 de los 13 individuos con dislexia, y en ninguno de los demás individuos (sin dislexia, o de fenotipo desconocido). Después, se secuenciaron 7 individuos clave de la familia para caracterizar la variación genética a nivel de nucleótido y estructural, centrándose en variación relativamente rara que estuviera compartida entre individuos con dislexia. No se encontró variación exónica nueva o rara en esta región. Sin embargo, se identificaron dos variantes no-codificantes relativamente raras en el primer intrón del gen SEMA3C (localizado en el 7q21) con putativos efectos reguladores según las anotaciones in silico.
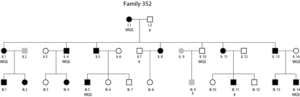
El rol biológico de la variante más prometedora (rs144517871) se evaluó mediante experimentos funcionales en líneas celulares humanas, confirmando que esta región en el primer intrón de SEMA3C tiene actividad reguladora, y que rs144517871 modula la expresión génica en líneas celulares. Hay que tener en cuenta que las variantes identificadas en este estudio no son mutaciones nuevas, ya que se encuentran en la población general en frecuencias bajas (alrededor de 1%). Estas variantes se podrían consideran como factores de riesgo de penetrancia particularmente alta en esta familia, que actuarían en conjunto con otros efectos genéticos y/o de ambiente.
El gen SEMA3C codifica una semaforina de clase III. Estas proteínas secretadas se unen a la plexina y tienen un rol importante en la regulación de procesos de desarrollo, incluyendo las señales de guía para neuronas corticales en migración (Chen et al. 2008; Van Battum et al. 2015). Debido a su rol en el desarrollo del cerebro, SEMA3C es un gen candidato convincente para la susceptibilidad de la dislexia.
La estrategia de combinar el análisis de ligamiento y secuenciación ha permitido identificar nuevas variantes genéticas posiblemente implicadas en dislexia. Como resultado, se ha propuesto SEMA3C como gen de susceptibilidad para la dislexia, abriendo nuevas vías de investigación para entender mejor cuáles son los genes y las redes biológicas que permiten el desarrollo de las habilidades lectoras.
Artículo de referencia:
Carrion-Castillo A., Estruch S.B., Maassen B., Franke B., Francks C., Fisher S.E. Whole-genome sequencing identifies functional noncoding variation in SEMA3C that cosegregates with dyslexia in a multigenerational family. Human Genetics. 2021. doi: https://doi.org/10.1007/s00439-021-02289-w
Bibliografia
Bishop DV (2015) The interface between genetics and psychology: lessons from developmental dyslexia. Proc Biol Sci 282(1806):20143139.
Chen G, Sima J, Jin M, Wang KY, Xue XJ, Zheng W et al (2008) Semaphorin-3A guides radial migration of cortical neurons during development. Nat Neurosci 11(1):36–44.
Einarsdottir E, Svensson I, Darki F, Peyrard-Janvid M, Lindvall JM, Ameur A et al (2015) Mutation in CEP63 co-segregating with developmental dyslexia in a Swedish family. Hum Genet 134(11–12):1239–1248
Einarsdottir E, Peyrard-Janvid M, Darki F, Tuulari JJ, Merisaari H, Karlsson L et al (2017) Identification of NCAN as a candidate gene for developmental dyslexia. Sci Rep 7(1):9294.
Hannula-Jouppi K, Kaminen-Ahola N, Taipale M, Eklund R, Nopola-Hemmi J, Kaariainen H, Kere J (2005) The axon guidance receptor gene ROBO1 is a candidate gene for developmental dyslexia. PLoS Genet 1(4):e50.
Peterson RL, Pennington BF (2015) Developmental dyslexia. Annu Rev Clin Psychol 11:283–307.
Taipale M, Kaminen N, Nopola-Hemmi J, Haltia T, Myllyluoma B, Lyytinen H et al (2003) A candidate gene for developmental dyslexia encodes a nuclear tetratricopeptide repeat domain pro tein dynamically regulated in brain. Proc Natl Acad Sci USA 100(20):11553–11558.
Van Battum EY, Brignani S, Pasterkamp RJ (2015) Axon guidance proteins in neurological disorders. Lancet Neurol 14(5):532–546.
Si te ha gustado esta noticia y quieres aprender más sobre Genética en Medicina, te interesan nuestros cursos y formación universitaria, así como nuestro canal audiovisual, Genotipia TV.



