Mª Llanos Martínez Martínez. Universidad Católica de Murcia. (UCAM). Campus de los Jerónimos, nº 135 Guadalupe 30107, Murcia. España.
Jerónimo Lajara Blesa. Universidad Católica de Murcia. Campus de los Jerónimos, nº 135 Guadalupe 30107, Murcia. España.
Por primera vez, se consigue en roedores ciegos, introducir ADN en una localización concreta del genoma de células que no se dividen, mediante edición génica HITI, devolviéndoles parcialmente la visión.
La edición génica dirigida por endonucleasas es un área de la investigación biomédica con un alto potencial para aplicaciones clínicas. Sin embargo, a pesar de los rápidos avances en este campo, la integración transgénica “in vivo” es todavía inviable porque las técnicas utilizadas hasta ahora para modificar el ADN a través del sistema de Repeticiones Palindrómicas Cortas y Regularmente Espaciadas (CRISPR Cas9) son ineficaces (Naldini, 2015), especialmente para células que no se dividen, las cuales componen la mayor parte de los tejidos adultos. Esto supone una barrera para el descubrimiento de principios fundamentales de la biología y el desarrollo de tratamientos para el abordaje en las enfermedades que producen desórdenes genéticos (Cox, 2013).
En este estudio, basado en la tecnología CRISPR Cas9, (Mali, 2014; Cong 2013) se desarrolla la técnica de Integración Dirigida Homóloga Independiente (HITI), la cual permite realizar en el ADN un “knock in” (Maresco, 2013; Auer, 2014) tanto en células que no se dividen como en las que se dividen, y lo más importante, in vivo. Además, se puede incorporar ADN nuevo, mediante la inserción de un exón, en células que no se dividen, con una eficacia diez veces superior a la técnica CRISPR Cas9 usada de forma aislada.
La metodología utilizada se basa en el uso de una vía de reparación del ADN llamada NHEJ (Unión de Extremos No Homólogos) que repara errores del ADN uniendo los extremos de las hebras originales (Bétermier, 2014). Gracias a esta técnica de reparación génica, se consigue colocar con éxito el ADN reparado en el “locus” preciso de las células que no se dividen.
Para ello, en primer lugar, se creó un paquete genético compuesto por ácidos nucleicos y un virus inerte, el cual se insertó en neuronas de ratones adultos y cuyo objetivo era obtener una mejora de la maquinaria de reparación de la vía NHEJ para el uso del sistema CRISPR Cas9, que permite insertar el ADN en localizaciones muy precisas del genoma y, a su vez, comprobar que la metodología HITI era efectiva in vitro en células que no se dividen.
Actualmente, las técnicas de edición génica cuyo objetivo es la integración transgénica en lugares específicos, están basadas en la recombinación homóloga directa y no son eficaces en células que no se dividen (Maresco, 2013; Auer, 2014). Sin embargo la recombinación no homóloga sí que resulta útil para reparar la rotura de ADN tanto en células en proliferación como en fases postmitóticas.
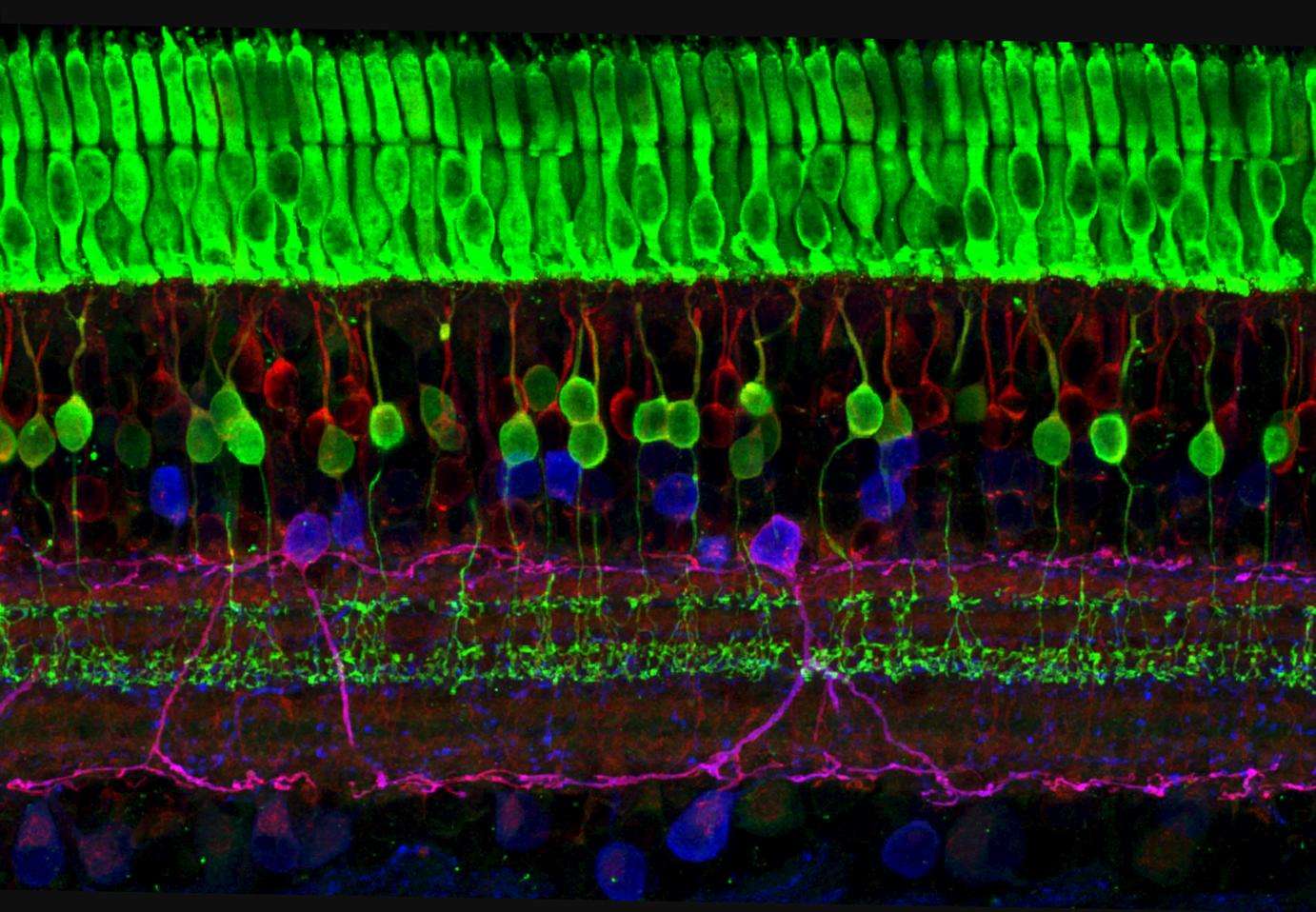
Para confirmar el potencial terapeútico y la eficacia de HITI en terapia génica in vivo, en este estudio se ha utilizado, como prueba de concepto, la aplicación de la técnica en un modelo de rata con degeneración retiniana a causa de una retinosis pigmentaria, con el fin de observar la función visual. Se usó la técnica HITI para insertar en el ojo de ratas de tres semanas de edad una copia correcta del gen MERKT, uno de los genes dañados en dicha enfermedad (D´Cruz, 2000). Los análisis realizados a las ratas, pasadas cinco semanas del tratamiento, mostraron que éstas eran capaces de responder a la luz y superaron diferentes pruebas que indicaban la curación de las células de su retina.
El resultado del experimento fue que las ratas ciegas mejoraron su visión, lo que indica el potencial de esta técnica para el tratamiento de enfermedades causadas por mutaciones monogénicas.
El método HITI, presentado aquí, establece nuevos avances para investigación básica y terapias génicas dirigidas, con la gran ventaja de que esta técnica HITI se puede adoptar para trabajar con cualquier sistema de ingeniería genética, no sólo con CRISPR Cas 9.
Referencia: Suzuki K, et al. In vivo genome editing via CRISPR/Cas9 mediated homology-independent targeted integration. Nature. 2016 Dec 1;540(7631):144-149. doi: 10.1038/nature20565.
Bibliografía:
Auer TO et al. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology independent DNA repair. Genome Res. 2014.24, 142–153.
Bétermier M et al. Is non-homologous end-joining really an inherently error-prone process? PLoS Genet. 2014. 10, e1004086 .
Cong L et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 2013. 339, 819–823.
Cox DBT et al. Therapeutic genome editing: prospects and challenges. Nat. Med. 2015. 21, 121–131.
D’Cruz PM et al. Mutation of the receptor tyrosine kinase gene Mertk in the retinal dystrophic RCS rat. Hum. Mol. Genet. 2000. 9, 645–651.
Mali P et al. RNA-guided human genome engineering via Cas9. Science.2013. 339, 823–826 .
Maresca M et al. Obligate ligation-gated recombination (ObLiGaRe): custom-designed nuclease-mediated targeted integration through nonhomologous end joining. Genome Res.2013 23, 539–546.
Naldini L. Gene therapy returns to centre stage. Nature. 2015. 526, 351–360.

