Reconstrucción de proteínas de los sistemas CRISPR-Cas ancestrales
Amparo Tolosa, Genotipia
Un estudio internacional liderado por investigadores españoles ha reconstruido las proteínas de los sistemas CRISPR-Cas que podrían haber existido en las bacterias de hace muchos millones de años. El trabajo, que contribuye a entender cómo han evolucionado los sistemas CRISPR-Cas a lo largo del tiempo, ofrece nuevas herramientas de interés para la edición genómica y sus aplicaciones. Los resultados se publican en Nature Microbiology.
Desde su descubrimiento y desarrollo como herramientas para modificar el genoma, los sistemas CRISPR-Cas se han convertido en elementos indispensables en la investigación médica y el diseño de terapias para enfermedades genéticas.
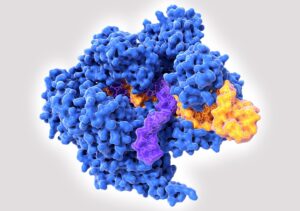
Los sistemas CRISPR de edición genómica derivan de un mecanismo de defensa de las bacterias frente a virus descrito por primera vez por el investigador español Francis Mojica. Un elemento fundamental de estos sistemas son las nucleasas Cas, responsables de cortar ácidos nucleicos en las posiciones indicadas por otros elementos.
En la actualidad la mayor parte de las nucleasas Cas utilizadas derivan del primer sistema CRISPR-Cas descrito en profundidad, el correspondiente a la bacteria Streptococcus pyogenes. No obstante, existe un gran interés en identificar otras proteínas Cas. Por una parte, nuevas proteínas Cas pueden ampliar el rango de reconocimiento de las secuencias a editar, aportar nuevas características o funcionalidades de interés. Por otra parte, en el ámbito de la edición del ADN para tratar enfermedades interesa obtener proteínas Cas de bacterias que no hayan tenido contacto excesivo con humanos, para prevenir la presencia de anticuerpos frente a ellas, que puedan interferir con su función terapéutica.
Frente a la opción de buscar nuevas proteínas Cas en las especies actuales de bacterias y arqueas distribuidas por todo el territorio geográfico, los investigadores españoles han seguido una estrategia alternativa: resucitar proteínas Cas ancestrales. El equipo ha reconstruido la secuencia de proteínas Cas9 que pudieron existir hace millones de años a partir de la información de microorganismos actuales y la historia evolutiva inferida a partir de sus secuencias. Interesantemente, al igual que sus equivalentes modernas, las nucleasas “resucitadas” funcionan y permiten editar el genoma de células humanas. Además, presentan propiedades que favorecen su utilidad en el ámbito de las tecnologías de edición genética.
“Resulta sorprendente que podamos revitalizar proteínas Cas que debieron existir hace miles de millones de años y constatar que ya tenían entonces la capacidad de operar como herramientas de edición genética, algo que hemos confirmado en la actualidad editando con éxito genes en células humanas”, ha señalado Lluís Montoliu, investigador del Centro Nacional de Biotecnología del CSIC (CNB-CSIC) y del CIBERER, cuyo equipo ha validado funcionalmente las nucleasas ancestrales en células humanas en cultivo.
Una reconstrucción de la evolución de los sistemas CRISPR-Cas9
Para resucitar las proteínas Cas ancestrales, los investigadores necesitaban sus secuencias proteicas, que han obtenido a partir de la reconstrucción de la historia evolutiva de las proteínas Cas9 de 59 bacterias actuales.
Alineando las secuencias de las proteínas actuales, y utilizando un algoritmo para inferir los cambios producidos a lo largo del tiempo, el equipo ha reconstruido la secuencia de 5 proteínas Cas 9 ancestrales probables: PDCA, PCA, SCA, BCA y FCA. Las fechas estimadas en las que existieron se remontan en el pasado mucho más que cualquier resto de ADN obtenido hasta el momento: 37, 137, 200, 1.000 y 2.600 millones de años.
El siguiente paso de los investigadores fue sintetizar en el laboratorio las proteínas ancestrales y evaluar su función y efectividad para editar el genoma. En este punto, los resultados indican que las versiones ancestrales de Cas9 mostraban mayor flexibilidad que las actuales y apuntan a un proceso adaptativo en el que se fueron adquiriendo funciones más específicas.
Por ejemplo, las formas de Cas más antiguas muestran actividad independiente a las secuencias conocidas como PAM. En el contexto biológico actual, estas secuencias actúan como mecanismo de seguridad en la respuesta antiviral bacteriana ya que son utilizadas por las bacterias para diferenciar entre su propio material genético y el de los virus. En el contexto de la edición génica, la presencia de secuencias PAM es un requerimiento también, aunque en este caso limita las posiciones en las que puede editarse el genoma.
Otras características de las formas más ancestrales de las proteínas Cas9 reconstruidas son la capacidad para introducir cortes en una sola de las cadenas del ADN, o su capacidad para cortar ARN o ADN de cadena sencilla. Estas características son propias de un entorno antiguo donde el ADN de doble cadena era menos prevalente que en la actualidad.
“Esta investigación supone un extraordinario avance en el conocimiento sobre el origen y evolución de los sistemas CRISPR-Cas. En cómo la presión selectiva de los virus ha ido puliendo a lo largo de miles de millones de años una maquinaria rudimentaria, poco selectiva en sus inicios, hasta convertirla en un sofisticado mecanismo de defensa capaz de distinguir con gran precisión el material genético de invasores indeseados que debe destruir, de su propio ADN que debe preservar”, ha señalado Francis Mojica, investigador de la Universidad de Alicante y descubridor del sistema CRISPR-Cas9, al que dio nombre.
Nuevas herramientas para la edición genética
Además de analizar las características de las formas ancestrales de Cas9 los investigadores analizaron su potencial para editar dos genes relacionados con el desarrollo de albinismo, enfermedad genética de interés para los grupos de investigación. Las cuatro Cas ancestrales más recientes permitieron editar el ADN de ambos genes en células humanas, con diferente eficiencia, siendo más eficientes cuanto más recientes.
Los resultados del trabajo demuestran que es posible “resucitar” proteínas Cas9 ancestrales con características de interés para la edición del genoma actual y aplicaciones biotecnológicas derivadas. Además, el análisis de su evolución a lo largo del tiempo ofrece una perspectiva del escenario ancestral en el que evolucionó el sistema CRISPR-Cas y, por tanto, una forma de mejorar la comprensión sobre su funcionamiento.
“Los sistemas actuales son muy complejos y están adaptados para funcionar dentro de una bacteria. Cuando el sistema se utiliza fuera de ese entorno, por ejemplo, en células humanas, el sistema inmune provoca un rechazo y existen además determinadas restricciones moleculares que limitan su uso. Curiosamente, en los sistemas ancestrales algunas de estas restricciones desaparecen, lo que les confiere una mayor versatilidad para nuevas aplicaciones”, ha indicado Rául Pérez-Jiménez, investigador Ikerbasque de CIC nanoGUNE que ha dirigido el proyecto.
“Es un trabajo genuinamente original con una aproximación imaginativa para, de manera última, entender la evolución del sistema CRISPR-Cas9 durante miles de millones de años. La mejora de la tecnología CRISPR-Cas pasa por entender bien cómo funcionan estos sistemas desde un punto de vista bioquímico y estructural. Conocer cómo han cambiado a lo largo de la evolución nos va a permitir abordar estas mejoras desde un nuevo punto de vista, con potenciales aplicaciones en biotecnología y biomedicina”, ha señalado Miguel ángel Moreno-Mateos, profesor de la Universidad Pablo de Olavide e investigador Ramón y Cajal en el Centro Andaluz de Biología del Desarrollo (CABD) a Science Media Center.
Referencia: Alonso-Lerma, B., Jabalera, Y., Samperio, S. et al. Evolution of CRISPR-associated endonucleases as inferred from resurrected proteins. Nat Microbiol (2023). https://doi.org/10.1038/s41564-022-01265-y
Fuentes: Resucitan ancestros de la herramienta de edición genética CRISPR de hace 2600 millones de años. https://www.ciberer.es/noticias/resucitan-ancestros-de-la-herramienta-de-edicion-genetica-crispr-de-hace-2600-millones-de-anos
Resucitando proteínas CRISPR-Cas ancestrales de hace millones de años. https://montoliu.naukas.com/2023/01/02/resucitando-proteinas-crispr-cas-ancestrales-de-hace-millones-de-anos/
Si te gusta esta noticia y quieres aprender más sobre Genética en Medicina, te interesan nuestros cursos, como el de Introducción a la Terapia Génica y Edición del Genoma, y formación universitaria, así como nuestro canal audiovisual, Genotipia TV.