Jesús Duque-Afonso
Department of Pathology, Stanford University School of Medicine, Stanford, California, USA
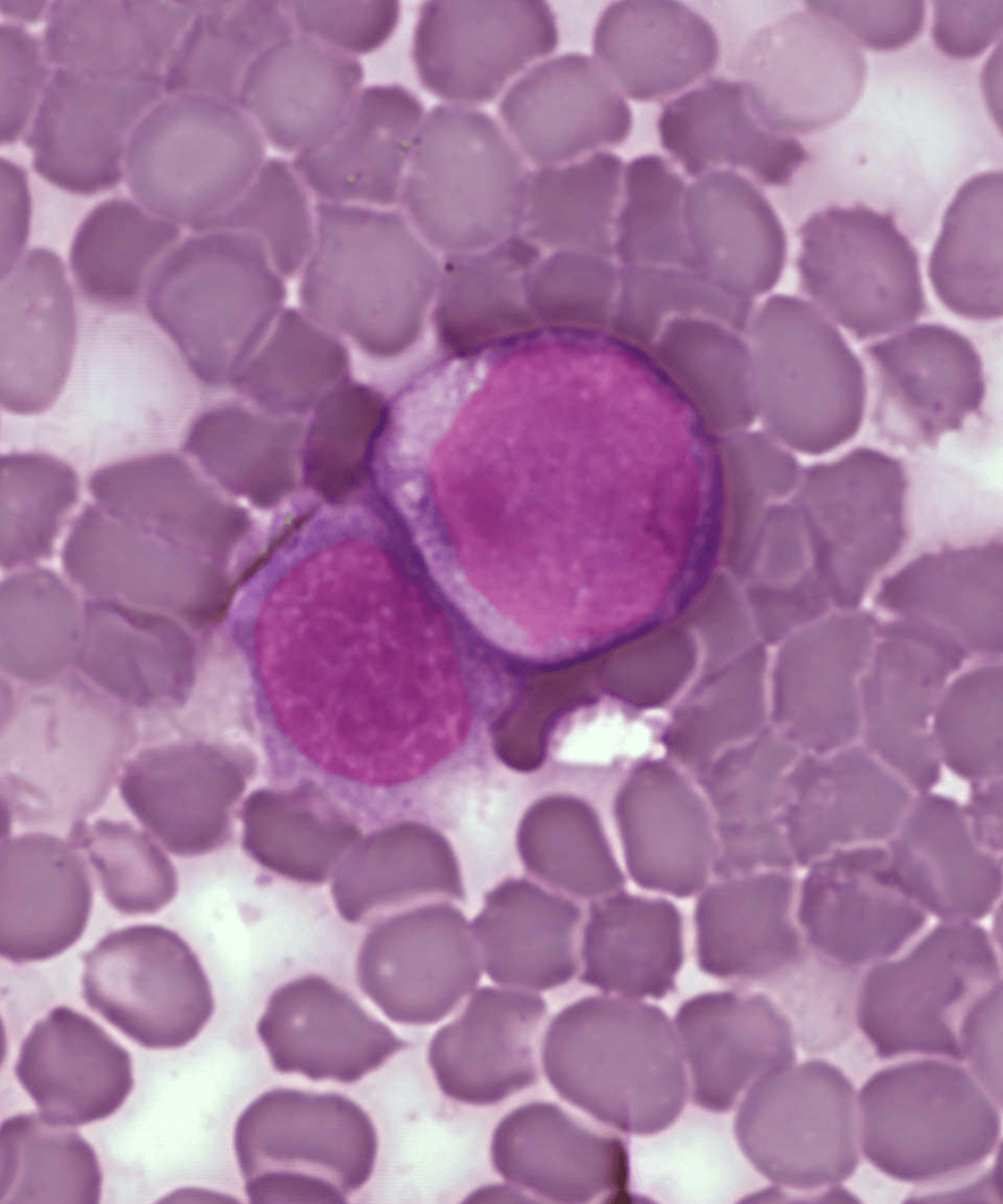
La leucemia linfoblástica aguda (LLA) tiene una incidencia de 3-4 casos por cada 100.000 habitantes al año, y está considerada como el cáncer pediátrico más frecuente. A pesar de haber mejorado el tratamiento en las últimas décadas, el 20% de los niños y el 60% de los adultos diagnosticados con LLA todavía fallecen, debido a la enfermedad o a causas relacionadas con el tratamiento. Además, la LLA es una enfermedad genética heterogénea que se puede clasificar según el origen de las células leucémicas en LLA de células B o T. Dentro de cada grupo hay distintos subtipos según la aberración genética primaria que posean las células leucémicas.
Debido a la baja incidencia, el mal pronóstico y la diversidad genética de la enfermedad, se necesitan modelos preclínicos como ratones transgénicos para investigar la patogénesis molecular y biología traslacional, que pueden llevar al descubrimiento de nuevas terapias más efectivas y menos tóxicas.
Estudios recientes usando tecnologías de secuenciación de próxima generación han demostrado que las leucemias son más complejas y diversas de lo que fue observado inicialmente. En el modelo actual de patogénesis multipaso de LLA, una lesión inicial confiere auto-replicación a las células madre hematopoyéticas o progenitores linfáticos. Una segunda mutación altera factores de transcripción de células B bloqueando la diferenciación celular. Por último, múltiples eventos genéticos cooperantes inducen proliferación celular incontrolada que lleva a la leucemia. Sin embargo, aunque este modelo multipaso está basado en elegantes estudios genómicos de LLA humana, no ha sido validado in vivo o en un sistema experimental.
En un trabajo recientemente publicado en la revista Journal of Clinical Investigation, el grupo liderado por el Prof. Michael Cleary, de la Universidad de Stanford, USA, ha desarrollado un modelo de ratón de LLA de células B que recapitula las características genéticas de la enfermedad humana.
Este modelo de ratón se basa en la expresión de la proteína de fusión E2A-PBX1, que se genera en la translocación t(1;19), detectada en un 5-7 % de las leucemias pediátricas. A diferencia de estudios previos, la estrategia que usaron los investigadores fue dirigir la expresión del oncogén específicamente al compartimento de células madres hematopoyéticas o a los progenitores de células B. Interesantemente, los tres tipos diferentes de promotores que se emplearon en este estudio para controlar la expresión de la Cre recombinasa mostraron que la frecuencia de las leucemias aumentaba cuanto antes se expresara E2A-PBX1 durante el desarrollo de células B.
Las células leucémicas de este modelo de ratón son muy similares desde el punto de vista citomorfológico y del immunofenotipo a las leucemias humanas. Concretamente, los investigadores encontraron células leucémicas con cadenas μ citoplasmáticas y expresión del receptor de células pre-B (pre-BCR), características de las LLA que expresan E2A-PBX1. Además, estas leucemias fueron especialmente susceptibles in vitro e in vivo a los efectos del inhibidor de tirosina quinasas dasatinib, cuyo uso ha sido aprobado para el tratamiento del subtipo de LLA con cromosoma Filadelfia.
La larga latencia (~9 meses) y la penetrancia parcial de las leucemias desarrolladas por el modelo de ratón sugieren que E2A-PBX1 necesita aberraciones genéticas cooperantes para inducir leucemia. Para ello, los investigadores realizaron estudios de secuenciación del exoma completo para identificar variaciones del número de copias, mutaciones, deleciones e inserciones. Uno de los genes afectados con más frecuencia por variación del número de copias fue el factor de transcripción de células B Pax5. Sorprendentemente, la incidencia del deleciones de Pax5 fue muy similar en leucemias de ratón (30%) a las descritas en leucemias humanas (45%). Para comprobar la cooperación entre E2A-PBX1 y Pax5, el modelo de ratón transgénico de E2A-PBX1 fue cruzado con ratones haplo-insuficientes de Pax5, lo cual incrementó la frecuencia y redujo la latencia de las leucemias.
Un elevado número de genes se vieron afectados por variaciones de la secuencia de nucleótidos durante el desarrollo de las leucemias. Específicamente, las vías de transducción de señales JAK/STAT y RAS/MAPK se vieron afectadas por mutaciones espontáneas, las cuales han sido detectadas previamente en pacientes con LLA. Asimismo, las células leucémicas presentaron un aumento de fosforilación de las proteínas implicadas en estas vías, como STAT5 y ERK. Este nuevo modelo de ratón se puede utilizar como plataforma preclínica para estudios de biología traslacional que requieran un sistema inmune intacto. De hecho, este trabajo constituye la primera evidencia de que el inhibidor de las JAK quinasas ruxolitinib aumenta la supervivencia de ratones trasplantados con leucemias con la translocación E2A-PBX1 y mutación de Jak1.
En resumen, se han generado ratones condicionales transgénicos que modelan de manera precisa y robusta la LLA de células B humana. Utilizando estudios de genómica comparada, la patogénesis multipaso de LLA humana ha sido confirmada en este modelo experimental in vivo. Este modelo de ratón, que es muy similar a las LLA humanas, puede ser usado como plataforma preclínica para el descubrimiento y desarrollo de nuevos fármacos que sean más efectivos y menos tóxicos para los pacientes.
* Esta investigación ha sido financiada por becas de la Deutsche Forschungsgemeinschaft (DU 1287/2-1 and SCHE 1870/1-1), Alex’s Lemonade Stand Foundation for Childhood Cancer, Lucile Packard Foundation for Children’s Health, the Child Health Research Institute, and the Stanford NIH-NCATS-CTSA grant.
Referencia: Duque-Afonso J, et al. Comparative genomics reveals multistep pathogenesis of E2A-PBX1 acute lymphoblastic leukemia. J Clin Invest. 2015 Sept;125(9):3667-80. doi: 10.1172/JCI81158.