María Hernández Sánchez1,2,3, Isabel González-Gascón y Marín4,5, José Ángel Hernández4,5, Jesús María Hernández Rivas1,2,3
1 – Genética molecular en Oncohematología. Instituto de Investigación Biomédica de Salamanca, Salamanca.
2 – Unidad de Citogenética Molecular. Centro de Investigación del Cáncer, Salamanca.
3 – Servicio de Hematología. Hospital Universitario de Salamanca, Salamanca.
4 – Servicio de Hematología. Hospital Universitario Infanta Leonor, Madrid.
5 – Departamento de Medicina. Universidad Complutense, Madrid.
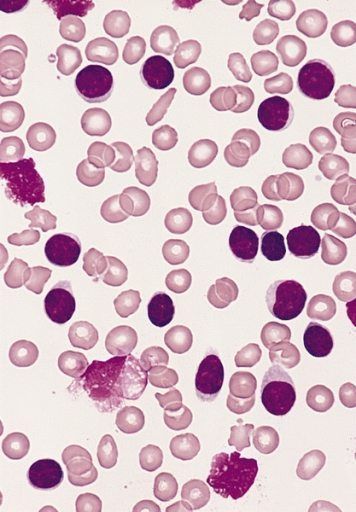
La leucemia linfocítica crónica (LLC) es un cáncer hematológico que se caracteriza por la acumulación progresiva de linfocitos B maduros en la sangre, la médula ósea y el tejido linfático. Esta enfermedad es muy heterogénea a nivel clínico debido en gran medida a la gran variabilidad de defectos genéticos que pueden presentar las células tumorales de cada paciente. Una de las técnicas que se utiliza para identificar anomalías cromosómicas que ayuda a definir el pronóstico de los enfermos es la hibridación in situ fluorescente (FISH). De los pacientes con LLC a los que se realiza un estudio citogenético de FISH, aproximadamente el 80% presentan alteraciones cromosómicas. Entre ellas, la deleción del brazo largo del cromosoma 13 [del(13q)] se asocia con buen pronóstico, la trisomía 12 con pronóstico intermedio, y la deleción del brazo largo del cromosoma 11 [del(11q)] y la deleción del brazo corto del cromosoma 17 [del(17p)] con pronóstico desfavorable.
Identificación de un pequeño subgrupo de pacientes de leucemia linfocítica crónica con ganancias cromosómicas y mal pronóstico
Un primer estudio retrospectivo con 1 359 enfermos de LLC analizados mediante FISH en el Hospital Universitario de Salamanca permitió identificar 7 pacientes que tenían células tumorales con ganancias de material cromosómico en al menos tres de las cinco sondas usadas mediante la técnica de FISH (11q22/ATM, 12q13, 13q34, 14q34/IGH y 17p13/TP53) (González-Gascón y Marín I, et al. 2016). Estos resultados sugerían que este pequeño grupo de enfermos presentaban un cariotipo hiperdiploide caracterizado por la presencia de más de 46 pares de cromosomas. La hiperdiploidía es una anomalía citogenética muy conocida en otras neoplasias hematológicas como la leucemia aguda linfoblástica, el mieloma múltiple, o la leucemia mieloide aguda. Se asocia con un pronóstico favorable en mieloma múltiple y leucemia linfoblástica aguda infantil. Por el contrario, en la LLC hemos descrito que es un evento citogenético muy poco frecuente (menos del 1% de las LLCs), y se asocia con mal pronóstico. En comparación con un grupo control de pacientes de LLC sin hiperdiploidía, las LLCs con hiperdiploidía presentaban de manera significativa menores niveles de hemoglobina, mayores niveles de LDH, estadios clínicos de Rai avanzados, y un mayor porcentaje de casos con hepatomegalia. Además, un mayor porcentaje de pacientes no presentaban mutaciones en la cadena pesada de las inmunoglobulinas. Todos estos marcadores pronósticos clínicos y biológicos se asocian con un curso clínico desfavorable en la LLC.
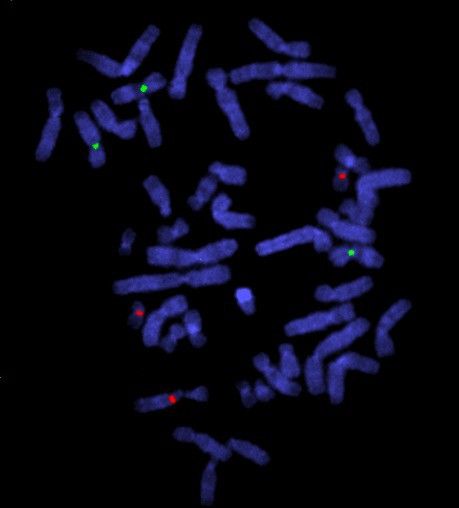
Seis de los siete pacientes de LLC con hiperdiploidía necesitaron tratamiento durante el curso de su enfermedad. Cabe destacar que el tiempo hasta el primer tratamiento era significativamente menor en el grupo de enfermos con hiperdiploidía respecto al grupo control (1 mes versus 63 meses). También presentaron una supervivencia global significativamente menor (66 meses versus 147 meses). En general, la LLC es una enfermedad asintomática al diagnóstico y la mayoría de los pacientes no necesitan tratamiento. Por tanto, hemos definido un subgrupo de pacientes de LLC de alto riesgo que, aunque no es común, presenta un pronóstico muy adverso.
Presencia de alteraciones genéticas en genes involucrados en respuesta al daño del ADN
Resultados previos en otras neoplasias han descrito que la hiperdiploidía se relaciona con la inestabilidad genética debido a defectos en el gen TP53. Así, en un segundo estudio nuestro grupo planteó analizar el perfil mutacional de los enfermos de LLC con hiperdiploidía (Hernández-Sánchez M, et al. 2019). Para ello, en el laboratorio se diseñó un panel de 54 genes incluyendo los genes más frecuentemente mutados en la LLC e involucrados en su patogénesis para analizar la presencia de mutaciones somáticas mediante secuenciación masiva. En primer lugar, se observó que la frecuencia de genes mutados es más alta que en una cohorte de LLCs sin seleccionar y al diagnóstico, siendo los genes más frecuentemente mutados TP53 (43%), ATM (29%), SF3B1 (29%) y BRAF (29%). Además, el 62% de las mutaciones habían sido descritas en genes previamente asociadas con mal pronóstico en la LLC (ATM, TP53, SF3B1, BRAF, NOTCH1, RPS15).
Al analizar qué alteraciones genéticas podrían estar relacionadas con el cariotipo hiperdiploide de estos enfermos, se observó que la mayoría de estos pacientes (72%) tenían deleciones o mutaciones en genes como TP53 o ATM. La inactivación de estos genes involucrados en el mecanismo de respuesta a daño del ADN se ha asociado con una mayor inestabilidad genómica. Tres de los pacientes tenían alteraciones en TP53: uno tenía TP53 mutado, y los otros dos tenían mutación y pérdida de TP53 (la deleción fue descrita mediante FISH). Dos de estos pacientes tenían mutado también SF3B1, un gen conocido por su papel en la maquinaria de splicing del ARN, pero que recientemente se ha descrito que mutaciones en este gen pueden llevar a un fallo en la respuesta del daño del ADN. También uno de los enfermos con TP53 mutado presentaba una mutación en el gen RPS15, que se ha relacionado con alteraciones de TP53 y se ha descrito como un nuevo driver de la LLC. Por otra parte, dos casos tenían alteraciones en ATM: uno tenía una mutación, mientras que el otro tenía un defecto bialélico de este gen debido a la presencia de mutación y deleción. Llama la atención que estos dos pacientes presentaban mutaciones en el gen BRAF. Este oncogén desempeña un papel importante en la ruta biológica RAS/MAPK/ERK, y recientemente se han descrito que defectos en esta vía de señalización se asocian con mal pronóstico en la LLC.
Al comparar las mutaciones identificadas en el grupo de pacientes con hiperdiploidía y en el grupo control sin este perfil citogenético, se observó un porcentaje significativamente mayor de enfermos con mutaciones genéticas involucradas en respuesta al daño del ADN (72% versus 12%), y que podrían resultar en una mayor inestabilidad genómica en estos pacientes. De hecho, la media de mutaciones fue significativamente mayor en los enfermos con hiperdiploidía.
Será necesario integrar estos conocimientos clínicos y genéticos en cohortes más numerosas de pacientes de LLC para confirmar los resultados observados en este subgrupo de LLCs con tan baja incidencia. En este sentido, se debe recordar el contexto de envejecimiento que sufre la población española que tiene la esperanza de vida más elevada de toda la Unión Europea, lo que supondrá un aumento de las enfermedades crónicas en los próximos años. Por ello, serán de especial relevancia para el futuro la realización de estudios de investigación dirigidos a profundizar en el conocimiento de neoplasias como la LLC. En paralelo, la práctica de estudios funcionales permitirá validar los defectos en la respuesta y la reparación de daño de ADN en células primarias de enfermos de LLC con hiperdiploidía. Con el objetivo de aprovechar los defectos de las células tumorales para inducir su muerte, la letalidad sintética podría presentarse como una aproximación ideal de medicina personalizada en este subgrupo de pacientes diagnosticados de LLC.
Estos trabajos han sido financiados por el Fondo de Investigación Sanitaria (PI15/01471 y PI18/01500) del Instituto de Salud Carlos III (ISCIII) cofinanciado por el Fondo Europeo de Desarrollo Regional (FEDER) “Una manera de hacer Europa”. MHS ha recibido financiación de la Sociedad Española de Hematología y Hemoterapia (FEHH-Janssen).
Referencias
González-Gascón y Marín I, et al. 2016. Hyperdiploidy as a rare event that accompanies poor prognosis markers in CLL. European Journal of Haematology. doi: http://dx.doi.org/10.1111/ejh.12812.
Hernández-Sánchez M, et al. 2019. DNA damage response-related alterations define the genetic background of patients with chronic lymphocytic leukemia and chromosomal gains. Experimental Hematology. doi: http://dx.doi.org/10.1016/j.exphem.2019.02.003.



