Ainara Castellanos-Rubio1,2,3 y Jose Ramón Bilbao1,3
1 UPV-EHU, 2 Columbia University, 3 BioCruces Research Institute.
Los estudios de asociación de genoma completo (GWAS) han descrito alrededor de 40 regiones asociadas a riesgo de enfermedad celiaca localizadas en partes no codificantes del genoma (Trynka et al, 2011). El SNP asociado rs917997 se encuentra 1.5 kilobases aguas abajo del gen IL18RAP, un gen que se ha relacionado con la enfermedad celiaca y otras patologías autoinmunes. Dado que en el equivalente a esta región en el genoma de ratón se transcribe un lncRNA (lnc13), se analizó la región en humanos y se encontró que las células humanas también sintetizan este lncRNA, describiéndose un gen nuevo que alberga una variante de riesgo a desarrollar la enfermedad.
Este lncRNA se transcribe y regula de forma independiente de IL18RAP (a pesar de que ambos genes se transcriben de la misma hebra de DNA y comparten 400 pares de bases de secuencia). La expresión de IL18RAP esta inducida tanto en las biopsias intestinales de los celiacos como en macrófagos estimulados con lipopolisacarido (LPS) (tratamiento que simula la respuesta inmune). Sin embargo, en las mismas condiciones, la expresión de lnc13 se ve reducida. Esta reducción es dependiente del factor de transcripción NFkB que esta constitutivamente activo en el intestino de los pacientes celiacos (incluso con dieta sin gluten) (Fernandez-Jimenez, 2014). Se observó que en los macrófagos activados con LPS el factor de transcripción NFkB se activa e induce la expresión de la proteína Dcp2, un regulador negativo de la estabilidad de los RNAs (Li et al, 2008) que desestabiliza lnc13 y mantiene bajos sus niveles. Así, los pacientes celiacos presentan niveles reducidos de lnc13 debido a la desestabilización del lncRNA mediada por Dcp2 y el factor de transcripción NFkB.
También se observó que los niveles de expresión de lnc13 se correlacionan negativamente con la de varios genes inflamatorios, algunos de los cuales están sobreexpresados en las biopsias de pacientes celiacos. Se pudo confirmar que la función de lnc13 es inhibir la expresión de estos genes inflamatorios en células en las que la respuesta inmune esta inactiva. Al igual que en otros lncRNAs conocidos, lnc13 se encuentra principalmente en el núcleo celular y silencia la expresión génica mediante su unión a factores de la cromatina (Huarte et al, 2010; Carpenter et al, 2013). Concretamente, lnc13 forma un complejo con la proteína hnRNPD (heterogenous nuclear ribonucleoprotein D) y con la enzima histona-deacetilasa 1 (HDAC1). En células no estimuladas el complejo represor formado por lnc13 se localiza en los promotores de los genes proinflamatorios manteniéndolos transcripcionalmente inactivos. En este trabajo se demuestra cómo lnc13, hnRNPD y HDAC1 se asocian a los promotores de varios genes pero aún queda por aclarar si existen más genes diana de este lncRNA.
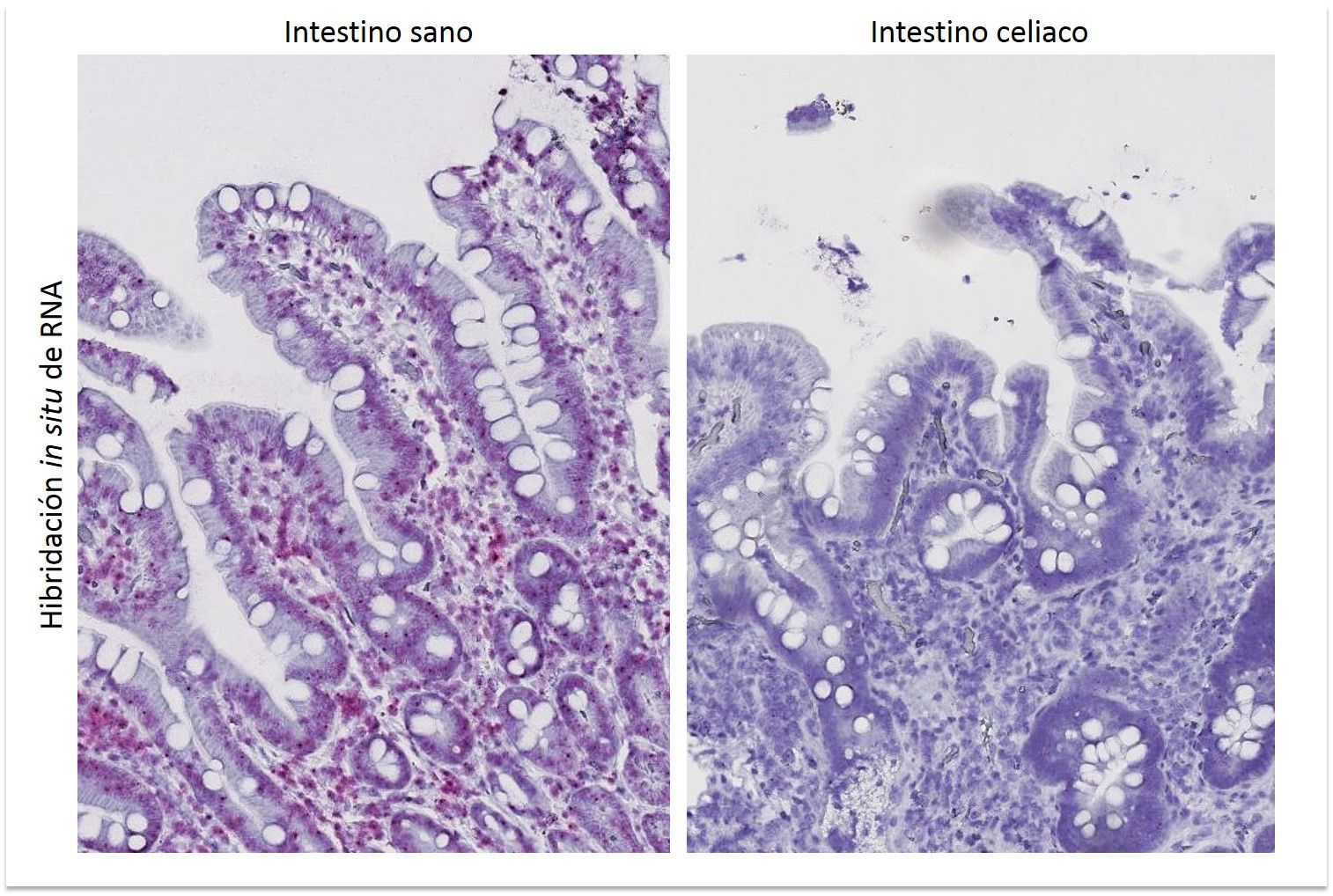
La actividad de lnc13 está mediada por su interacción con la proteína hnRNPD, y esta interacción es dependiente del genotipo del SNP asociado. En presencia del alelo de riesgo rs917997*T la interacción del lncRNA con la proteína hnRNPD es más débil, lo que reduciría la capacidad de lnc13 para regular la expresión de los genes inflamatorios a los que se une. Este descubrimiento subraya la relevancia de la estructura tridimensional de las moléculas de RNA, y revela cómo cambios pequeños en la secuencia de RNA pueden tener un impacto funcional importante.
En resumen, en este estudio se observa que lnc13, un lncRNA desconocido hasta ahora, está alterado en la enfermedad celiaca y además representa cémo un SNP asociado a una enfermedad compleja puede directamente afectar la función de un lncRNA. Es posible que otros SNPs asociados con riesgo a enfermedades comunes actúen de manera similar, pero por el momento no es posible predecir cómo actúan estas moléculas. Estudios experimentales sobre el funcionamiento de los lncRNAs ayudarán a descifrar los principios moleculares que gobiernan su Biología y permitirá sentar las bases para el diseño de nuevas terapias dirigidas.
Referencia
Castellanos-Rubio et al. A long-noncoding RNA associated with susceptibility to celiac disease. Science 2016. DOI: http://dx.doi.org/10.1126/science.aad0467
Bibliografia
Trynka G, et al. Dense genotyping identifies and localizes multiple common and rare variant association signals in celiac disease. Nat Genet. 2011. 43, 1193-1201, doi:10.1038/ng.998
Fernandez-Jimenez, N. et al. Coregulation and modulation of NFkappaB-related genes in celiac disease: uncovered aspects of gut mucosal inflammation. Hum Mol Genet. 2014. 23, 1298-1310, doi:10.1093/hmg/ddt520.
Li Y, et al. Transcript‐specific decapping and regulated stability by the human Dcp2 decapping protein. Mol Cell Biol. 2008. 28, 939-‐948, doi:10.1128/MCB.01727-07.
Huarte, M. et al. A large intergenic noncoding RNA induced by p53 mediates global gene repression in the p53 response. Cell. 2010. 142, 409-419, doi:10.1016/j.cell.2010.06.040.
Carpenter, S. et al. A long noncoding RNA mediates both activation and repression of immune response genes. Science. 2013. 341, 789‐792, doi:10.1126/science.1240925.