Investigadores de la Universidad de Pensilvania han creado un atlas con información genética y molecular sobre las proteínas de superficie de las células del cáncer.
Como bien ha confirmado la pandemia de COVID-19 la superficie de las células humanas tiene un papel muy relevante para la salud del organismo. En ocasiones, como ocurre con el coronavirus, la superficie de ciertas células contiene la puerta de entrada para agentes patógenos. En otras, como se observa en cáncer, las proteínas de la membrana pueden identificar a las células tumorales como tales y favorecer el desarrollo de terapia dirigidas a ellas de forma específica.
En la actualidad, ya existen estrategias terapéuticas para hacer frente al cáncer basadas en la presencia de determinadas proteínas en su superficie, como los anticuerpos dirigidos a reconocer proteínas que se expresan en gran cantidad en la membrana de las células tumorales o los linfocitos modificados con el mismo objetivo.
Se estima que alrededor del 65% de los fármacos de terapia inmunológica o dirigida frente al cáncer están dirigidos a genes que codifican proteínas de la superficie de las células tumorales. Sin embargo, estos genes representan una proporción muy pequeña de todos lo que se encargan de producir la amplia variedad de proteínas externas a la célula.
Dado el potencial de conocer en detalle qué proteínas se encuentran habitualmente en la superficie de las células tumorales, un equipo de investigadores de la Universidad de Pensilvania ha abordado la compleja tarea de identificarlas y catalogarlas. Para ello ha analizado más de 11000 muestras de tumores de 33 tipos de cáncer diferentes.
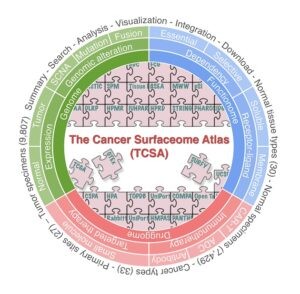
Los investigadores han combinado seis aproximaciones genómicas y moleculares diferentes para caracterizar los genes codifican para proteínas localizadas en la superficie de las células tumorales y responder a múltiples cuestiones, como cuál es el patrón de expresión de estos genes en tejido sano y en cáncer, qué alteraciones son más frecuentes, cuáles de estos genes son esenciales para el crecimiento tumoral y con qué otras moléculas interaccionan las proteínas que codifican
.
Un atlas de proteínas de superficie del cáncer para el futuro desarrollo de tratamientos
La lista de genes que codifican para proteínas de superficie obtenida por el equipo incluye más de 3500 elementos, la mitad de los cuales pertenecen a familias de genes con potencial para ser diana de moléculas pequeñas. Dentro de este conjunto de datos, los investigadores destacan que un 17,21% de los genes codifican para moléculas que modulan la respuesta inmunitaria.
Los investigadores también destacan el hecho de que 409 genes que codifican para proteínas de superficie se expresan de forma específica en al menos un cáncer, lo que los convierte en candidatos para el desarrollo de tratamientos de inmunoterapia. Estos tratamientos están dirigidos a potenciar la acción del sistema inmunitario de los pacientes hacia las células tumorales de forma específica. Algunos de estos genes identificados ya están siendo estudiados en el desarrollo clínico de linfocitos modificados o tratamientos basados en anticuerpos. Otros representan nuevas oportunidades.
Dentro de las posibilidades terapéuticas planteadas para los genes identificados, el equipo de investigadores señala que lo ideal sería poder desarrollar terapias dirigidas a múltiples dianas simultáneamente. Esta estrategia permitiría seleccionar mejor las células a las que se dirige el tratamiento evitando posibles efectos adversos en las células sanas. En esta línea, el equipo ha identificado pares de proteínas de superficie que podrían utilizarse en el diseño de linfocitos T para el cáncer.
Por otra parte, los investigadores también han analizado, en 33 tipos de cáncer, qué alteraciones se producen de forma recurrente en los genes que codifican para proteínas de superficie y han identificado 1433 dianas potenciales que muestran alteraciones recurrentes al menos en un tipo de cáncer. Estudios futuros deberán analizar en detalle cuáles de ellas pueden derivar en fármacos, como ha ocurrido con las conocidas mutaciones en EGFR o la amplificación de Her2 en cáncer de mama.
Los resultados del trabajo, publicado en Nature Genetics, están accesibles en la base de datos creada por los investigadores: http://fcgportal.org/TCSA/index.php
Referencia: Hu, Z., Yuan, J., Long, M. et al. The Cancer Surfaceome Atlas integrates genomic, functional and drug response data to identify actionable targets. Nat Cancer 2, 1406–1422 (2021). https://doi.org/10.1038/s43018-021-00282-w
Si te ha gustado esta noticia y quieres aprender más sobre Genética en Medicina, te interesa nuestra formación, como el “Máster de Medicina de Precisión y Genética Clínica“, o el “Experto Universitario en Oncogenética“.



