Centro Nacional de Análisis Genómico
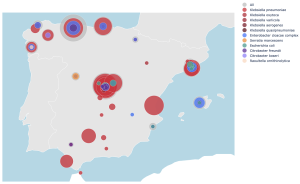
- La base de datos online proporciona el perfil genómico de 461 cepas bacterianas resistentes a los antibióticos, procedentes de 41 hospitales situados en 13 regiones diferentes de España.
- El recurso médico fue posible gracias a una nueva metodología genómica que permite obtener secuencias completas de ADN bacteriano de manera más rápida y a gran escala, abriendo la puerta al seguimiento continuo de la evolución de la resistencia a los medicamentos de estos tipos de bacterias.
- El estudio, codirigido por el Centro Nacional de Análisis Genómico (CNAG), Roche Diagnostics y el Instituto de Investigación Biomédica A Coruña (INIBIC), arroja luz sobre los mecanismos responsables de la transmisión de la resistencia a los antimicrobianos en una clase de bacterias altamente resistentes a los antibióticos.
- La investigación española contribuye al plan de acción de la Organización Mundial de la Salud (OMS), fortaleciendo el conocimiento de las infecciones con bacterias resistentes a los antibióticos que representan hoy en día una amenaza para la salud pública global.
En las secuencias genómicas de las bacterias se encuentra la clave para conocer si estas bacterias podrán combatirse con ciertos antibióticos. Dada su importancia para la salud pública, estos perfiles genómicos se han agrupado en la primera biblioteca española de bacterias resistentes a los antibióticos. La base de datos digital, con cerca de 500 bacterias, ha sido publicada por el Centro Nacional de Análisis Genómico (CNAG), Roche Diagnostics y el Complejo Hospitalario Universitario A Coruña-Instituto de Investigación Biomédica A Coruña (INIBIC), gracias a una nueva metodología genómica que permite obtener genomas bacterianos completos de manera mucho más rápida y a gran escala, posibilitando realizar un seguimiento rutinario de los patrones de transmisión de esta resistencia.
Según Tyler Alioto, autor del estudio y líder del equipo de Ensamblaje y Anotación del Genoma del CNAG: «La caracterización genómica completa de las bacterias solo se puede lograr con ensamblajes completos del genoma que proporcionen el cromosoma principal, además del conjunto completo de plásmidos y su número de copias. Para demostrar que esto se puede hacer a gran escala, secuenciamos 500 bacterias resistentes a los antibióticos con una combinación de tecnologías genómicas de última generación, como las lecturas cortas de Illumina y las lecturas largas de Oxford Nanopore Technologies (ONT) y desarrollamos un flujo de trabajo automatizado para procesar los datos».
El portal inCREDBle, que es el nombre de esta biblioteca de ADN, recopila un total de 461 cepas bacterianas resistentes a los antibióticos, recogidas de 41 hospitales situados en 13 regiones diferentes de España. La nueva base de datos digital complementa el perfil genómico de estas bacterias con datos clínicos, geográficos y microbiológicos, y con otros estudios online relacionados con la resistencia antimicrobiana, convirtiéndose en el recurso más completo para estudiar las enterobacterias, una de las familias de bacterias más resistentes a los antibióticos. La biblioteca (https://genomes.cnag.cat/incredble/) está disponible para consultas, exploración y descargas.

Cómo se transmite la resistencia a los antibióticos, clave para futuros tratamientos
El principal objetivo de la investigación española, publicada recientemente en la revista Microbial Genetics, consiste en proporcionar más información sobre los mecanismos por los cuales las especies del orden Enterobacterales adquieren o desarrollan esta resistencia a los medicamentos, así como también de los mecanismos subyacentes de diseminación.
«Una de las estrategias que propone el Centro para el Control y Prevención de Enfermedades (CDC) en EEUU para el control de la resistencia antimicrobiana es la vigilancia y monitoreo de patógenos resistentes o multirresistentes. Justamente la base de datos genómica que hemos creado permite la identificación, localización y seguimiento de las bacterias gramnegativas que portan uno de los mecanismos de resistencia a antibióticos más peligrosos, las enzimas llamadas carbapenemasas. Este trabajo puede servir para futuros estudios con un mayor número de aislados o cepas y que incluyan una mayor diversidad de patógenos resistentes. Esto permitiría tener información dinámica y a tiempo real sobre la transmisión de microorganismos multirresistentes en nuestro país», destaca Germán Bou, autor del estudio y responsable del Grupo de Investigación de Microbiología del INIBIC, y Jefe de Servicio de Microbiología del Complejo Hospitalario Universitario de A Coruña.
Uno de los principales factores que contribuyen a la resistencia a los antibióticos en las enterobacterias es la presencia de enzimas carbapenemasas. Estas enzimas tienen la capacidad de propagarse rápidamente debido a su asociación con elementos móviles y su presencia en los plásmidos, las moléculas de ADN circulares que se transmiten de célula a célula.
El estudio proporciona nuevos datos relacionados con la estructura de los plásmidos, precisamente por su papel importante en la transmisión de la resistencia antimicrobiana (RAM). Un conocimiento profundo de estos mecanismos podría ayudar a producir tratamientos más efectivos que eviten la resistencia antimicrobiana (RAM).
«Las bases de datos de alta calidad de genomas microbianos son realmente importantes para comprender la genética y biología con las que algunas de estas bacterias actúan contra los antibióticos. Son una herramienta muy valiosa para la investigación en este campo, ayudando en el desarrollo de nuevos métodos de diagnóstico y terapias de alto impacto para estos peligrosos microorganismos», afirma Miguel Álvarez-Tejado, Jefe de Marketing de Soluciones Moleculares en Roche Diagnostics.
Resistencia antimicrobiana, en el top de la lista de amenazas para la salud pública
Las bacterias resistentes a los antibióticos, causantes mayormente de infecciones respiratorias y urinarias, se han convertido en una preocupación para la salud pública a nivel mundial debido a su capacidad para desarrollar resistencia a los antibióticos más efectivos hasta la fecha, los carbapenémicos.
Según la Organización Mundial de la Salud (OMS), como resultado de la resistencia a los medicamentos, los antibióticos se vuelven ineficaces y las infecciones se vuelven difíciles o imposibles de tratar. La aparición y propagación de patógenos resistentes a los medicamentos amenaza nuestra capacidad para tratar infecciones comunes y llevar a cabo procedimientos que salvan vidas, como la quimioterapia contra el cáncer, la cesárea, los reemplazos de cadera, los trasplantes de órganos y otras cirugías.
Para hacer frente a esa amenaza global, la OMS ha promovido un plan de acción múltiple dividido en varios objetivos. El presente estudio, que proporciona la primera base de datos de bacterias resistentes a los antibióticos en España y una nueva metodología genómica para generar el perfil de ADN de estas bacterias, contribuye al segundo objetivo de este plan, relacionado con fortalecer la base de conocimiento y las pruebas a través de la vigilancia y la investigación de estas bacterias.
Artículo científico: Alioto, Tyler S., et al. ‘Development of a Novel Streamlined Workflow (AACRE) and Database (inCREDBle) for Genomic Analysis of Carbapenem-Resistant Enterobacterales’. Microbial Genomics, vol. 9, no. 11, 2023, p. 001132. Microbiology Society Journals. DOI: https://doi.org/10.1099/mgen.0.001132
#genetica #microbiología #microbiologíaclínica #genómica #bacteriasresistentes #antibióticos #basededatos #antimicrobianos #ADN
Si te ha gustado esta noticia y quieres aprender más sobre Genética en Medicina, te interesan nuestros cursos, como el de “Microbiología molecular en la práctica clínica” y formación universitaria, así como nuestro canal audiovisual, Genotipia TV.