AlphaFold3 predice la estructura e interacciones de proteínas con otras moléculas como el ADN y el ARN.
Desde su desarrollo, hace poco más de 3 años, el modelo de inteligencia artíficial AlphaFold 2 ha revolucionado la predicción de la estructura de las proteínas, abriendo numerosas oportunidades para la investigación biológica y aplicada. Sus aplicaciones se extienden desde el desarrollo de nuevos antibióticos a la identificación de nuevos métodos para administrar fármacos o de dianas de precisión para tratar el cáncer.
La estructura es una parte esencial para la función de las proteínas. Pero estas no trabajan solas en las células. Interaccionan con otras proteínas o moléculas como el ADN o ARN en sistemas que pueden alcanzar una gran complejidad. Por lo tanto, para entender bien cómo funcionan las proteínas y utilizar ese conocimiento en aplicaciones médicas o biotecnológicas es crucial saber también cómo interaccionan con las otras moléculas.
El pasado día 8 de mayo, Google Deepmind e Isomorphic Labs presentaron la última versión del modelo de inteligencia artificial, AlphaFold 3, en paralelo a la publicación de un trabajo en Nature que describe la herramienta. AlphaFold 3 amplía su capacidad predictiva y abarca una gama más amplia de moléculas biológicas: además de predecir estructuras de proteínas, estima la forma en que estas interactúan con otros tipos de moléculas, como ácidos nucleicos o ligandos.
“AlphaFold 3 lleva el mundo biológico a la alta definición. Permite a los científicos ver los sistemas sistemas celulares en toda su complejidad, a través de estructuras, interacciones y modificaciones”, han destacado desde Google DeepMind. “Esta nueva ventana a las moléculas de la vida revela cómo están conectadas entre sí y ayuda a comprender cómo influyen esas conexiones en las funciones biológicas”.
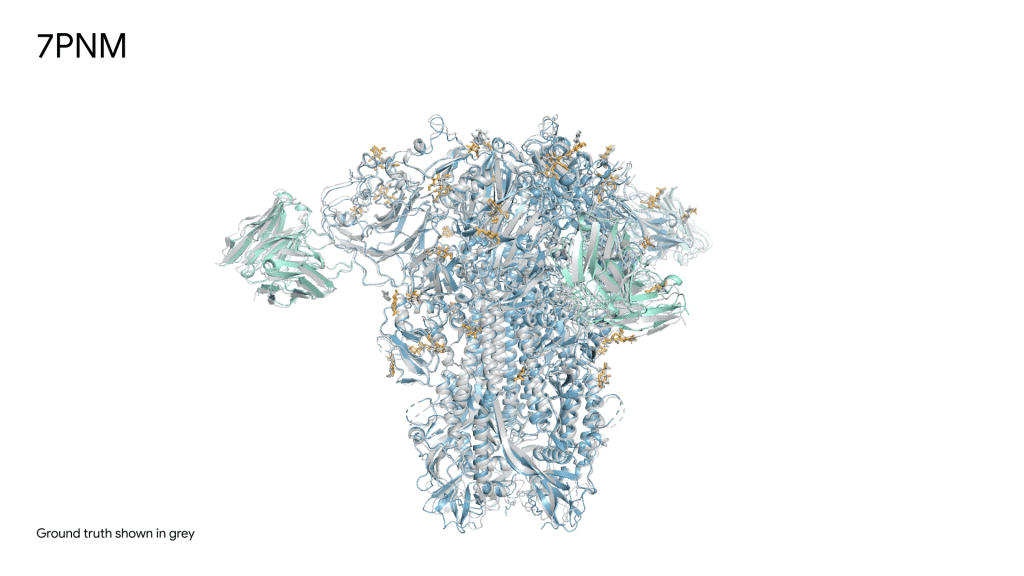
De predecir proteínas a predecir un sistema de interacciones
AlphaFold es un sistema avanzado de inteligencia artificial desarrollado por Google DeepMind. Las versiones anteriores, especialmente AlphaFold 2, utilizan aprendizaje profundo para predecir cómo se pliegan las cadenas de aminoácidos en estructuras tridimensionales. Además, AlphaFold-Multimer permitía la predicción de complejos proteína-proteína.
Todos estos modelos se basan en una arquitectura de redes neuronales que se entrena con datos de secuencias de proteínas y estructuras conocidas (como los de la conocida Protein Data Bank), e infiere las estructuras de proteínas desconocidas con una precisión comparativa a la de los métodos experimentales tradicionales.
AlphaFold 3 se ha desarrollado como una evolución natural de sus predecesores en la que se amplía el número de moléculas biológicas y se predicen las interacciones entre ellas.
A partir de una lista de moléculas, AlphaFold 3 genera su estructura tridimensional conjunta. Esta nueva versión modela complejos de proteínas con otras proteínas, ADN, ARN y moléculas pequeñas como los ligandos, entre los que se encuentran muchos fármacos. También predice interacciones anticuerpo-antígeno o modificaciones químicas de proteínas. En conjunto, AlphaFold3 permite predecir los complejos e interacciones que implican a prácticamente todos los tipos moleculares incluidos en Protein Data Bank.
Nueva arquitectura para estimar la estructura de todas las moléculas de la vida
Según indican los desarrolladores de AlphaFold 3, en las interacciones de las proteínas con otros tipos de moléculas se observa una mejora de al menos el 50% en comparación con los métodos de predicción existentes. Además, para algunas categorías de interacciones se ha duplicado la precisión de la predicción.
“Las capacidades de AlphaFold 3 proceden de su arquitectura y entrenamiento de nueva generación, que ahora abarca todas las moléculas de la vida”, señalan los desarrolladores. “El núcleo del modelo es una versión mejorada de nuestro módulo Evoformer, una arquitectura de aprendizaje profundo que sustentó el increíble rendimiento de AlphaFold 2. Tras procesar las entradas, AlphaFold 3 ensambla sus predicciones mediante una red de difusión, similar a las que se encuentran en los generadores de imágenes de inteligencia artificial.”
“AlphaFold 3 amplía el avance transformador que supuso AlphaFold 2. En resumen, lo hace más grande, más rápido y mejor”, ha destacado Julien Bergeron, investigador en el King´s College London, que ha utilizado AlphaFold 2 en el desarrollo de nuevos antibióticos. “Mientras que AlphaFold 2 nos permitió generar muy rápidamente hipótesis para ser probadas en el laboratorio, con esta nueva versión ahora podemos probar inmediatamente múltiples hipótesis in silico”.
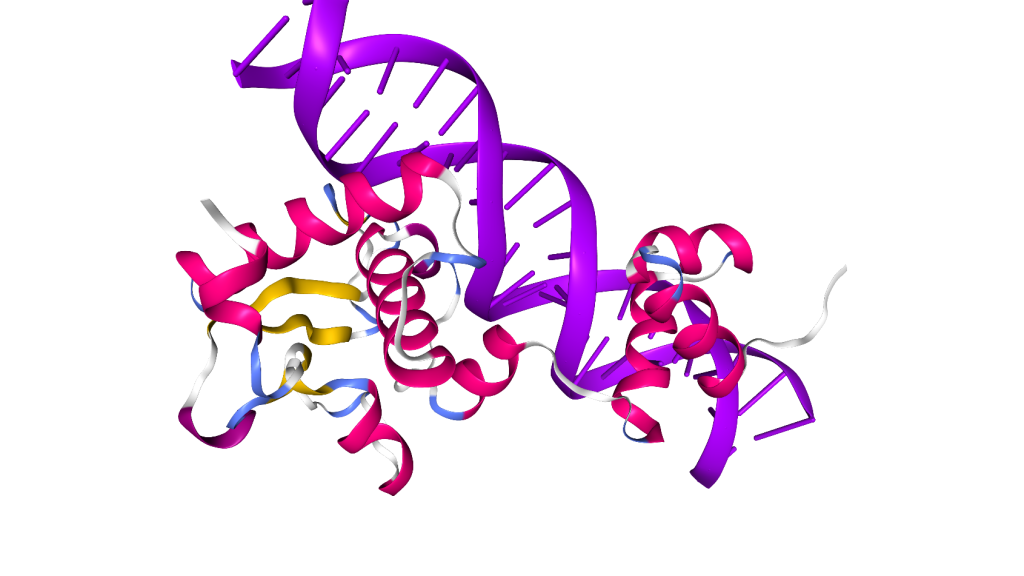
Aplicaciones en diseño de fármacos
Una de las aplicaciones más directas y destacables de AlphaFold3 es el diseño de fármacos. “ AlphaFold 3 proporciona a nuestros diseñadores de fármacos la capacidad de predecir con rapidez y precisión la estructura de complejos que nunca antes se habían caracterizado, proporcionando una herramienta fundamental que nos permite adoptar nuevos enfoques en el diseño de fármacos”, destacan desde Isomorphic Labs. “Ahora podemos crear y probar hipótesis a nivel atómico, y producir predicciones de estructura muy precisas en cuestión de segundos, lo que contrasta con los meses, o incluso años, necesarios para responder experimentalmente a preguntas similares”.
Por ejemplo, la herramienta de inteligencia artificial permite acelerar el diseño de moléculas pequeñas que se unan a sitios específicos de proteínas objetivo y puedan modificar su función. También abre nuevas posibilidades en el desarrollo de fármacos basados en anticuerpos u otras proteínas terapéuticas.
Por último, la predicción de las interacciones de proteínas con moléculas como el ARN o el ADN amplía también el tipo de dianas terapéuticas a considerar en el desarrollo de fármacos. “Creemos que esta comprensión más amplia del contexto en el que operan las dianas de fármacos se trasladará en fármacos más efectivos para la clínica”, han destacado desde Isomorphic Labs.
Un ejemplo concreto del potencial de AlphaFold 3 es la predicción de interacción entre moléculas pequeñas y la proteína TIM3, objetivo para la inmunoterapia del cáncer.. A través de AlphaFold 3 los investigadores detectaron una región de esta proteína no identificada previamente, con la que interaccionan los ligandos y a la que podría dirigirse el diseño de fármacos.
En la actualidad Isomorphic Labs, uno de los desarrolladores de AlphaFold 3 utiliza la herramienta en combinación con otros modelos de IA para diseñar fármacos en proyectos propios o compartidos con colaboradores farmacéuticos.
Aplicaciones en enfermedades genéticas
A través de su capacidad para predecir estructuras de complejos moleculares, AlphaFold 3 ofrece nuevas vías para entender cómo afectan las mutaciones genéticas a la estructura y función de las proteínas.
En el ámbito del diagnóstico, AlphaFold 3 podría facilitar la interpretación de la patogenicidad de variantes genéticas raras o desconocidas. Precisamente el año pasado, un equipo de investigadores desarrolló a partir de la versión anterior, AlphaFold 2, una herramienta, AlphaMissense, que permitía predecir el efecto de cualquier sustitución de aminoácido en el conjunto de proteínas de la especie humana.
Además, AlphaFold 3 podría facilitar la identificación y caracterización de las conformaciones estructurales de las proteínas afectadas por mutaciones patogénicas. Pequeñas alteraciones en la secuencia de aminoácidos pueden provocar cambios dramáticos en la estructura y función de algunas proteínas, con efectos importantes para la salud. Al estimar cómo alteran estas mutaciones la estructura proteica, sería posible desarrollar moléculas pequeñas que estabilicen la forma correcta de la proteína o que modifiquen la interacción patológica de proteínas mal plegadas con otras moléculas en la célula.
Por último, al permitir explorar interacciones proteína-ADN y proteína-ARN, AlphaFold 3 amplía notablemente la posibilidad de conocer mejor mecanismos implicados en enfermedades susceptibles de intervención farmacológica e identificar nuevas dianas terapéuticas. Por otra parte, podría contribuir a mejorar el diseño de terapias de edición genómica que requieren interacción entre proteínas y ácidos nucleicos. Este tipo de terapias está emergiendo como una solución terapéutica para diferentes enfermredades genéticas, con algunos tratamientos ya aprobados, como la primera terapia CRISPR aprobada para la anemia falciforme y la beta talasemia.
Gran potencial, todavía limitado para usos no comerciales
Desde la publicación de AlphaFold 2, numerosos científicos en todo el mundo han utilizado la herramienta de inteligencia artificial con objetivos que incluyen desde la mejora de cultivos al desarrollo de fármacos para la medicina de precisión. Actualmente, la base de datos AlphaFold DB, creada por Google DeepMind y el Instituto Europeo de Bioinformática, incluye más de 200 millones de predicciones de estructura de proteínas, dato que pone de manifiesto su potencial.
AlphaFold 3 amplía las capacidades para predecir las estructuras biológicas a nivel molecular, por lo que la expectativa es que sus aplicaciones sean más potentes y diversas.
La herramienta, sin embargo, no es perfecta. Algunas limitaciones a tener en cuenta todavía son errores en la quiralidad de las estructuras (una propiedad de las moléculas importante a nivel funcional) o lapredicción reducida de algunas estructuras secundarias. Para mejorar las predicciones y evitar las “alucinaciones”, predicciones plausibles pero no realistas que se generan con las técnicas de difusión utilizadas, los desarrolladores han indicado que será necesario generar grandes conjuntos de predicciones y ordenarlas en función de la estructura obtenida.
Para facilitar la utilización de AlphaFold 3, Google DeepMind ha lanzado AlphaFold Server, una plataforma gratuita que puede ser utilizada por investigadores de todo el mundo. De momento, no obstante, AlphaFold Server solo puede ser utilizada en investigaciones con fines no comerciales.
Artículo científico: Abramson, J., Adler, J., Dunger, J. et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature (2024). https://doi.org/10.1038/s41586-024-07487-w
Fuentes:
AlphaFold 3 predicts the structure and interactions of all of life’s molecules. https://blog.google/technology/ai/google-deepmind-isomorphic-alphafold-3-ai-model/
Rational drug design with AlphaFold 3. https://www.isomorphiclabs.com/articles/rational-drug-design-with-alphafold-3
AlphaFold Protein Structure Database: https://alphafold.ebi.ac.uk/
Si te ha gustado esta noticia y quieres aprender más sobre Genética en Medicina, te interesan nuestros cursos y formación universitaria, como el “Máster en Medicina de Precisión y Genética Clínica” así como nuestro canal audiovisual, Genotipia TV.


