Jennifer Valero-Garcia1, María del Carmen González-Espinosa2, Manuel Barrios2, Greta Carmona-Antoñanzas1, Javier García-Planells1, Carlos Ruiz-Lafora1, Ainhoa Fuentes-Gálvez2, Antonio Jiménez-Velasco2
1 Instituto de Medicina Genómica (imegen), Paterna, Valencia
2 Departamento de Hematología, Hospital Regional Universitario de Málaga, Instituto de Investigación Biomédica de Málaga (IBIMA), Málaga
Un estudio recientemente publicado por el equipo del Instituto de Medica Genómica de Valencia (Valero-Garcia et al., 2019) en colaboración con el Dr Antonio Jiménez-Velasco del Hospital Regional Universitario de Málaga ha dirigido su investigación a elucidar la capacidad de la PCR digital (dPCR) en el seguimiento de quimerismo molecular hematopoyético (QH).
El análisis de QH es un método clínicamente establecido para el seguimiento de pacientes con enfermedades hematológicas como las leucemias o los síndromes mielodisplásicos que han sido tratados con trasplantes alogénicos de células madre provenientes de un donante sano (Figura 1). En primer lugar hay que identificar marcadores moleculares tanto en el donante sano como en el receptor o paciente enfermo, los cuales permiten evaluar el riesgo de que el paciente sufra una recaída o un rechazo del trasplante a lo largo del periodo postrasplante.
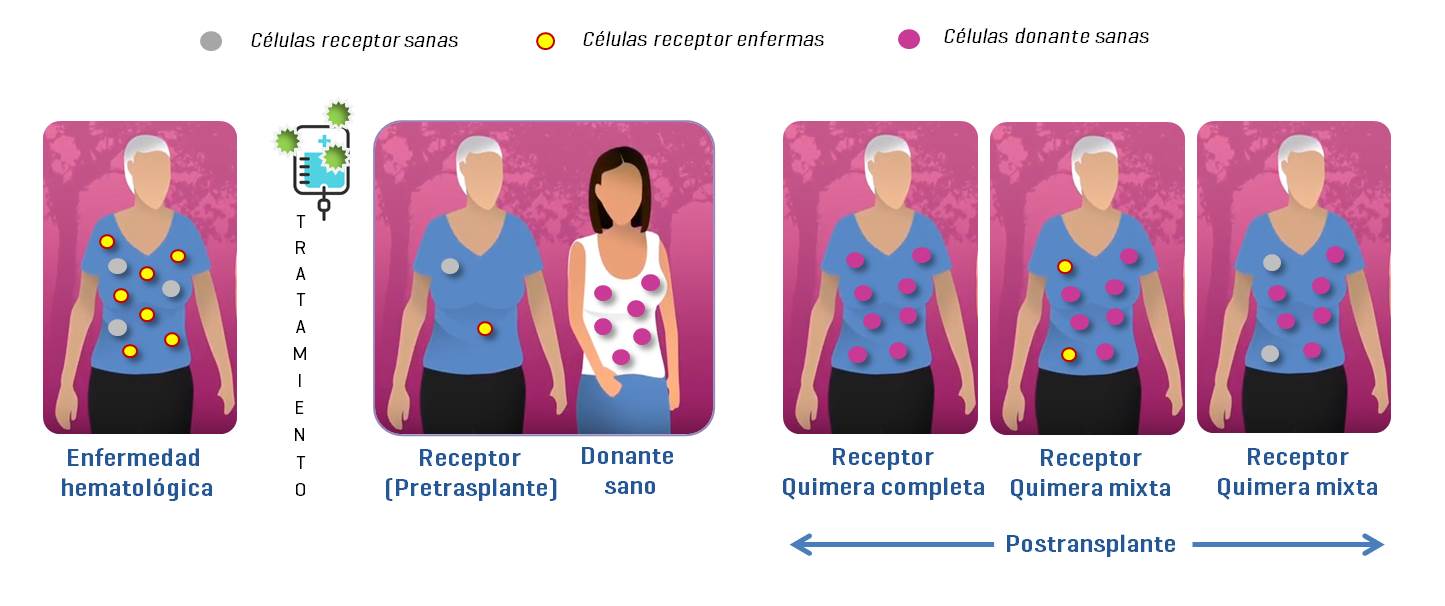
Históricamente, el seguimiento de quimerismo postrasplante ha empleado distintas técnicas de análisis y marcadores moleculares, utilizando estrategias citogenéticas como la hibridación in situ fluorescente (FISH) de las células de la sangre y la médula ósea y el análisis molecular de polimorfismos VNTR (número variable de repeticiones en tándem) o STR (repeticiones cortas en tándem) mediante reacción en cadena de la polimerasa convencional (PCR) y análisis de fragmentos (Barrios et al., 2003) actualmente considerado el gold standard en el análisis de quimerismo (Lion et al., 2012). Sin embargo, múltiples estudios clínicos demostraron que ninguna de las estrategias citadas anteriormente proporcionaba suficiente sensibilidad y precisión cuantitativa como para ser considerada la estrategia óptima en la predicción de la recaída morfológica temprana (Stahl et al., 2015).
En la última década, el uso de la PCR cuantitativa a tiempo real (qPCR) ha demostrado aportar una mayor sensibilidad, llegando a detectar hasta un 0,01% de ADN del receptor en muestras de sangre (Alizadeh et al., 2002). Con esta estrategia, la sensibilidad mejoró significativamente y la recaída de leucemia aguda se anticipó antes en el 88% de los casos en comparación con el 44% cuando se utilizó el análisis de fragmentos (Jiménez-Velasco et al., 2005). El análisis del quimerismo mediante qPCR ha demostrado ser más sensible; sin embargo, el error técnico inherente al método puede resultar en una desviación sustancial del valor verdadero (hasta una variación del 25%), lo que podría ser terapéuticamente significativo en casos de quimerismo mixto persistente (Stahl et al., 2015). El uso de réplicas puede minimizar ligeramente el error técnico, sin embargo, esta técnica requiere la inclusión de muestras de seguimiento previas en cada determinación y la construcción de una curva estándar, generalmente utilizando las muestras previas al trasplante (pretrasplante), para cuantificar con exactitud el valor del quimerismo hematopoyético.
La PCR digital (dPCR), tecnología state-of-the-art, se encuentra actualmente en el punto de mira de una amplia gama de especialidades médicas ya que ofrece el potencial para realizar una cuantificación absoluta con precisión, agregando sensibilidad y exactitud superiores a los métodos moleculares tradicionales. La dPCR permite la cuantificación absoluta mediante la partición de la muestra en miles de sub-réplicas funcionales, eliminando así la necesidad de utilizar réplicas técnicas y curvas de calibración. Asimismo, la dPCR ha demostrado una mayor precisión y robustez en presencia de inhibidores de la PCR, salvando las limitaciones técnicas inherentes de la qPCR (Huggett et al., 2005) y permite el diseño de ensayos multiplexados sin comprometer la sensibilidad del sistema de cuantificación.
El estudio recientemente publicado por el equipo del Instituto de Medica Genómica de Valencia (Valero-Garcia et al., 2019) y el Hospital Regional Universitario de Málaga seleccionó, en primer lugar, marcadores informativos para cada paciente mediante ensayos de genotipado de muestras pretrasplante del receptor y del donante, utilizando sistemas de PCR específicos para la detección de genes presentes en el cromosoma Y, alelos nulos o polimorfismos de inserción / deleción (INDEL) (imegenTM-Quimera kits, imegen). Una vez seleccionados los marcadores informativos para cada caso clínico, se analizaron 225 muestras postrasplante de ADN genómico procedentes de sangre periférica pertenecientes a 28 pacientes adultos que se habían sometido a un trasplante alogénico de médula ósea proveniente de un donante compatible sano.
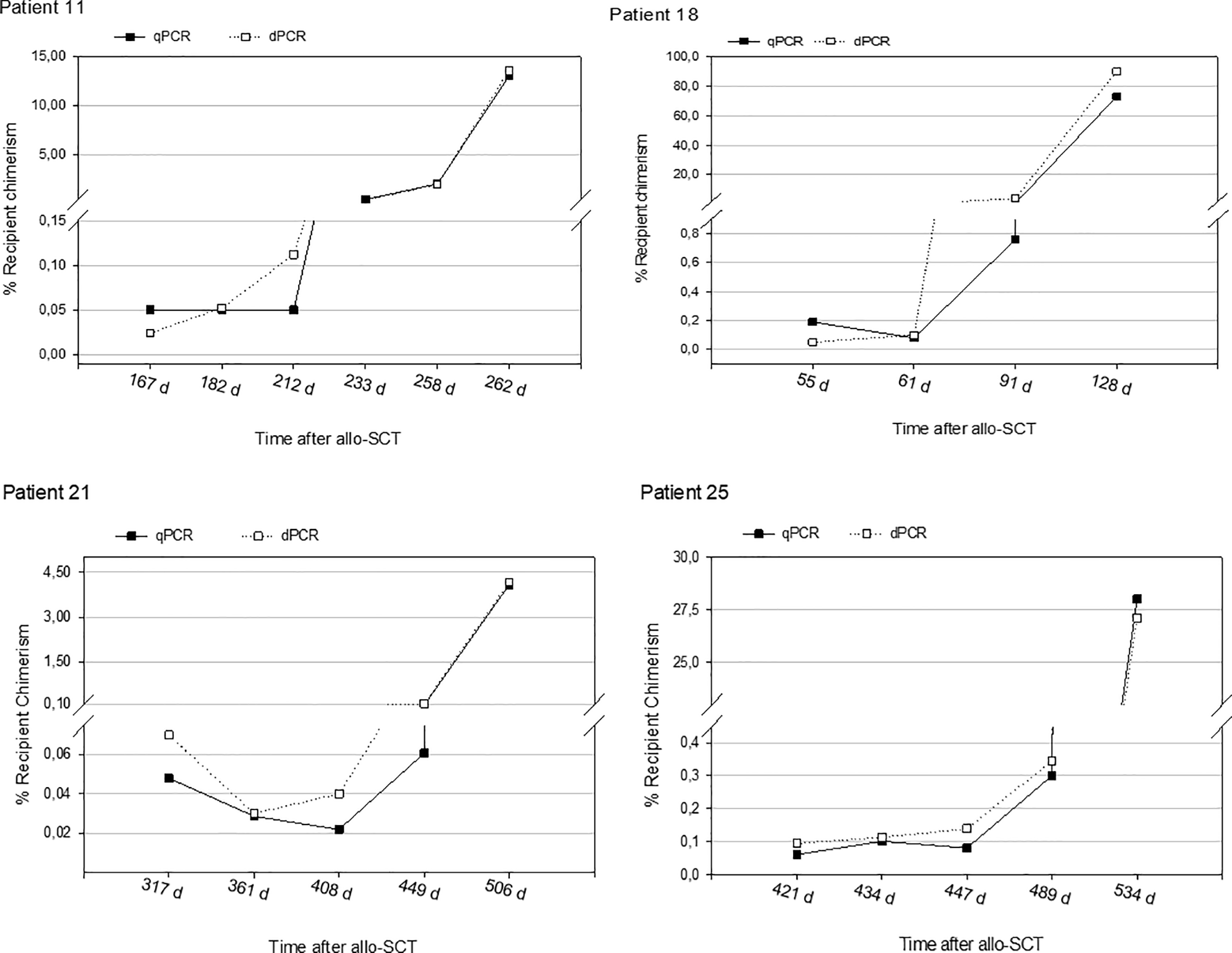
Los resultados indican que el incremento del quimerismo mixto (iQM), un parámetro altamente informativo en el estudio de la evolución de la salud del paciente y del trasplante, fue detectado previamente a la recaída en el 100% de los pacientes (18 recaídas) tanto mediante el uso de la qPCR como de la dPCR. En general, el 56% de las recaídas fueron predichas con antelación mediante dPCR (Figura 2) mientras que el 38% de las recaídas se predijeron simultáneamente usando ambas técnicas y sólo en un caso, la recaída fue predicha con antelación mediante qPCR. Sin embargo, cuando se evaluaron los tiempos de análisis postrasplante en detalle, se descubrió que la dPCR permitió predecir la recaída con una media de anticipación de 63 días frente a 45,5 días de anticipación media mediante qPCR, ofreciendo al equipo médico un periodo ventajoso de 17,5 días durante el cual se pueden aplicar terapias dirigidas para evitar la recaída del paciente.
En conclusión, nuestros resultados indicaron que la dPCR a partir de ADN extraído de sangre periférica constituye un enfoque adecuado para la detección del quimerismo mixto que permite un pronóstico más rápido de la recaída temprana en comparación con la qPCR lo cual podría afectar favorablemente a la salud y la supervivencia de los pacientes sometidos a trasplantes alogénicos de células hematopoyéticas.
Referencia: Valero-Garcia J et al. Earlier relapse detection after allogeneic haematopoietic stem cell transplantation by chimerism assays: Digital PCR versus quantitative real-time PCR of insertion/deletion polymorphisms. PLoS ONE. 2019; 14(2): e0212708. Doi: https://doi.org/10.1371/journal.pone.0212708
Bibliografía:
Alizadeh M et al. Quantitative assessment of hematopoietic chimerism after bone marrow transplantation by real-time quantitative polymerase chain reaction. Blood. 2002;99: 4618–25.
Barrios M et al. Chimerism status is a useful predictor of relapse after allogeneic stem cell transplantation for acute leukemia. Haematologica. 2003;88: 801–10.
Huggett JF et al. Considerations for Digital PCR as an Accurate Molecular Diagnostic Tool. Clin Chem. 2015;61: 79–88. Doi:10.1373/clinchem.2014.221366
Jiménez-Velasco A et al. Reliable quantification of hematopoietic chimerism after allogeneic transplantation for acute leukemia using amplification by real-time PCR of null alleles and insertion/deletion polymorphisms. Leukemia. 2005;19: 336–343. Doi:10.1038/sj.leu.2403622
Lion T et al. The EuroChimerism concept for a standardized approach to chimerism analysis after allogeneic stem cell transplantation. Leukemia. 2012;26: 1821–1828. Doi:10.1038/leu.2012.66
Stahl T et al. Digital PCR to assess hematopoietic chimerism after allogeneic stem cell transplantation. Exp Hematol. 2015;43: 462–468.e1. Doi:10.1016/j.exphem.2015.02.006


