
Genética Médica News
Coronavirus SARS-CoV-2: estructura, mecanismo de infección y células afectadas
- mayo 18, 2020
Amparo Tolosa, Genotipia
En los últimos meses el coronavirus SARS-CoV-2 ha pasado de ser un virus desconocido a convertirse en objeto de estudio de miles de investigadores. Posiblemente, nunca antes se ha avanzado tanto y en tan poco tiempo en la investigación de un agente infeccioso.
SARS-CoV-2 comparte muchas características con otros virus ya conocidos. No obstante, su estudio también ha revelado algunos rasgos que contribuyen a su virulencia y podrían ser aprovechados para desarrollar fármacos o vacunas. A continuación presentamos la información que se ha ido recopilando sobre su estructura, cómo entra a las células y cómo afecta al funcionamiento de las mismas.
Estructura del virus
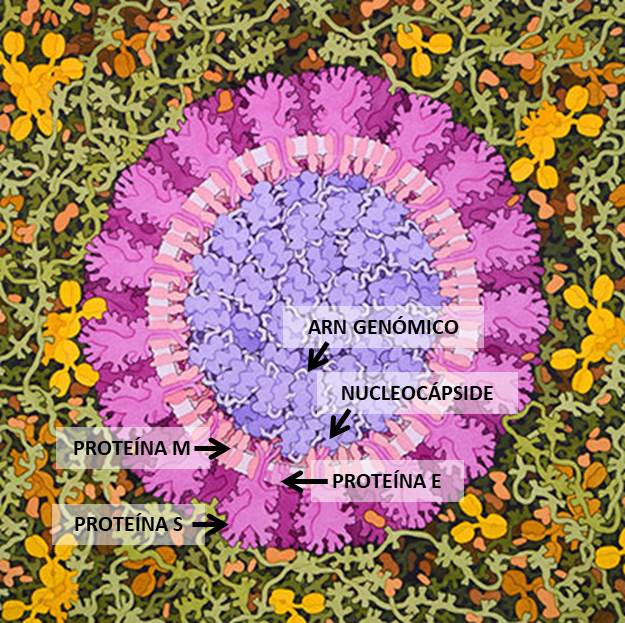
SARS-CoV-2 es un virus de ARN perteneciente a la familia de coronavirus, que recibe este nombre por la apariencia característica de las proteínas de su cubierta. Su genoma contiene 29891 nucleótidos que codifican para 9860 aminoácidos.
El virión de SARS-CoV-2, su forma infectiva, consiste en una membrana con glicoproteínas dentro de la que se empaqueta el material hereditario compactado con proteínas. En la superficie destaca la presencia de la proteína S, denominada así por formar la espícula, estructura en forma de aguja que tiene un papel relevante en la infección.
Mecanismo de infección y entrada a las células del organismo
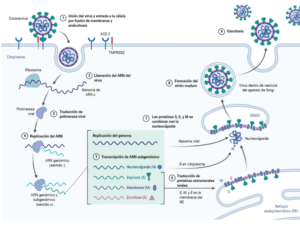
El primer paso de la infección por coronavirus es la entrada del virus en las células. El coronavirus SARS-CoV-2 entra en las células a través de la unión de una proteína de su superficie, la proteína S, con el receptor ACE-2 (enzima convertidora de angiotensina 2) de las células huésped. ACE-2 forma parte de una ruta bioquímica que interviene en la regulación de procesos como la inflamación o la presión sanguínea y su función habitual es modular la actividad de la angiotensina 2 para contrarrestar sus efectos dañinos.
La proteína S está formada por tres unidades idénticas organizadas en forma de círculo que encajan con el receptor ACE-2 como una llave y median la fusión de la cubierta membranosa del virus con la membrana de la célula que está siendo infectada. La unión entre la proteína S y el receptor ACE-2 marca el punto de destino del virus en el organismo, pero es la activación de la proteína S lo que abre las puertas de la célula al virus.
La activación de la proteína S está mediada la proteasa celular TMPRSS2, que suele localizarse cerca de ACE-2. TMPRSS2 corta la proteína S, lo que activa proteínas de la envoltura viral que favorecen la fusión con la membrana celular. De este modo, los virus entran en la célula rodeados de membrana celular, formando endosomas. En estas pequeñas bolsas celulares, se liberan catepsinas, otras proteínas que modifican de nuevo la proteína S, y proteasas que favorecen la liberación del ARN viral al citoplasma. Para este proceso son importantes las condiciones de pH en el interior de las vesículas.
Al tratarse de un ARN de sentido positivo, al ser liberado, el ARN viral se traduce directamente a poliproteínas, que son procesadas en proteínas funcionales responsables de la replicación y transcripción del virus. Así, por una parte, se producen ARNs que son traducidos en proteínas estructurales del virus y por otra se generan ARNs genómicos que serán empaquetados en los nuevos viriones que se van formando. Por último, los viriones se liberan al exterior de la célula y pueden infectar otras células.
El importante papel de la proteína S, el receptor ACE-2 y la proteasa TMPRSS2 en la entrada del virus a la célula ha hecho que estas moléculas se conviertan en moléculas clave para el desarrollo de tratamientos o vacunas.
Por ejemplo, diversos centros de investigación y empresas están desarrollando vacunas para COVID-19 basadas en la proteína S. En este caso, el objetivo es utilizar la proteína o fragmentos para generar una respuesta inmunitaria que prepare a la persona para una posible entrada del virus en el organismo. Otra estrategia en investigación es la administración de grandes cantidades de ACE-2 para que actúen de “señuelo” o trampa para el virus.
Qué células expresan ACE2
Dado que ACE-2 es la puerta de entrada del virus a las células del organismo, una cuestión relevante para determinar el alcance o las consecuencias clínicas de COVID-19 es conocer qué células expresan esta proteína y pueden ser infectadas.
ACE-2 está presente en células de órganos como el pulmón, corazón, riñón, vejiga y órganos del sistema digestivo. Si se considera también TMPRSS2, proteína implicada en la activación de la proteína S viral el rango de células susceptibles de ser infectadas es menor. El análisis de expresión de diferentes tejidos indica que ACE2 y TMPRSS2 se expresan especialmente en ciertas células de la cavidad nasal (las células caliciformes que producen mucus) de los pulmones (en los neumocitos que mantienen los alveolos abiertos) y el intestino (enterocitos responsables de la absorción de nutrientes). Interesantemente el interferón, una molécula implicada en la activación del sistema inmunitario, estimula la expresión de ACE2 en los pulmones.
Cómo afecta el virus a la expresión de las células infectadas
Otra cuestión interesante es cómo afecta el virus al funcionamiento habitual de las células que infecta. Estudiar qué rutas moleculares de la célula son importantes para el virus puede ayudar a identificar dianas para impedir su replicación.
El análisis de células en cultivo infectadas con SARS-CoV-2 indica que el virus se replica rápidamente en las células. Además, SARS-CoV-2 aumenta la actividad ciertas rutas celulares como la traducción de proteínas, el procesado de ARN o el metabolismo de los ácidos nucleicos y el carbono. Estudios preliminares en cultivo celular, indican que inhibidores de estas rutas inhiben la replicación del virus. Investigaciones futuras deberán evaluar si estos resultados tienen aplicación clínica en pacientes
Otro estudio, publicado en Cell, muestra también las alteraciones en la expresión de las células infectadas por SARS-CoV-2. Investigadores del Icah School of Medicine en el Hospital Monte Sinaí de Nueva York, han encontrado que una vez dentro de las células, el coronavirus SARS-CoV-2 no se comporta como otros virus respiratorios como el SARS. En general la presencia de los virus respiratorios activa la expresión de los genes que producen interferón, que inhiben la replicación del virus, y la expresión de quimiocinas, que son las señales que alertan a células cercanas y sistema inmunitario de la infección. Sin embargo, los estudios en células humanas o células de pacientes indican que SARS-CoV-2 bloquea los genes relacionados con la producción de interferón y activa los que producen las quimiocinas, que inducen el reclutamiento de macrófagos y la inflamación. Con este mecanismo, la respuesta del organismo al virus está desequilibrada respecto a lo que ocurre normalmente. Los investigadores sugieren que esta característica podría estar relacionada con la mayor frecuencia de casos graves de COVID-19 en pacientes con otras enfermedades que no tienen un sistema inmunitario robusto.
Conocer cómo funciona el coronavirus SARS-CoV-2 y cuáles son sus similitudes y diferencias respecto a otros coronavirus causantes de enfermedades es la mejor oportunidad de la que disponemos para desarrollar tratamientos efectivos para COVID-19 o diseñar vacunas que prevengan la infección. Además, esta información podría resultar de gran utilidad en potenciales futuras pandemias, causadas por coronavirus u otros agentes infecciosos.
Referencias:
Cascella M, et al. Features, Evaluation and Treatment Coronavirus (COVID-19). StatPearls. https://www.ncbi.nlm.nih.gov/books/NBK554776/
Ziegler C GK, et al. SARS-CoV-2 Receptor ACE2 Is an Interferon-Stimulated Gene in Human Airway Epithelial Cells and Is Detected in Specific Cell Subsets across Tissues. Cell. 2020. Doi: https://doi.org/10.1016/j.cell.2020.04.035
Bojkova D, et al. Proteomics of SARS-CoV-2-infected host cells reveals therapy targets. Nature. 2020. Doi: https://doi.org/10.1038/s41586-020-2332-7
Centro de Coordinación de Alertas y Emergencias Sanitarias. Enfermedad por coronavirus, COVID-19. https://www.mscbs.gob.es/profesionales/saludPublica/ccayes/alertasActual/nCov-China/documentos/20200417_ITCoronavirus.pdf
Ou X, et al. Characterization of spike glycoprotein of SARS-CoV-2 on virus entry and its immune cross-reactivity with SARS-CoV. Nat Com. 2020. Doi: https://dx.doi.org/10.1038%2Fs41467-020-15562-9
Hoffmann M, et al. SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor. Cell. 2020. Doi: https://dx.doi.org/10.1016%2Fj.cell.2020.02.052
Blanco-Melo D, et al. Imbalanced Host Response to SARS-CoV-2 Drives Development of COVID-19. Cell. 2020. doi: https://doi.org/10.1016/j.cell.2020.04.026
ACTUALIZACIÓN 21-05-2020: Añadida información sobre el artículo publicado en Cell que identifica diferencias en los cambios de expresión provocados por SARS-CoV-2 en la célula infectada respecto a otros virus.
Si te ha gustado esta noticia y quieres aprender más sobre Genética en Medicina, te interesan nuestros cursos y formación universitaria.
Categorías
- Actualidad
- Congresos
- Coronavirus
- CRISPR
- Debates
- Diagnóstico Genético
- Enfermedades Raras
- Entrevistas
- Envejecimiento y longevidad
- Epigenética
- Farmacogenética
- Formación
- Genética del cáncer
- Genética en Cardiología
- Medicina Reproductiva
- Microbiología molecular
- Neurociencia
- Noticias de Genotipia
- Noticias de investigación
- Noticias patrocinadas
- Proyectos
- Reseñas de libros
- Terapia Génica
- Tratamientos
Cursos relacionados
Contacto
¿Quieres publicar con nosotros? ¿Tienes dudas?
Contacta con nosotros de la manera que prefieras y te responderemos a la mayor brevedad.





