Amparo Tolosa, Genotipia
Investigadores del Instituto Arc, la Universidad de California Berkeley y la Universidad de Tokio han desarrollado una nueva técnica de edición del genoma que permite la inserción, inversión o eliminación de secuencias largas de ADN en posiciones concretas del genoma.
Las recientes herramientas de edicion del genoma como CRISPR o los editores de bases han abierto un abanico de posibilidades para la biotecnología y la medicina. Estas aproximaciones son útiles para realizar modificaciones puntuales en el genoma con eficiencia. Con ellas es posible, por ejemplo cambiar, un nucleótido del ADN para corregir una mutación. En esto precisamente, consiste la primera terapia de edición genómica aprobada para la anemia falciforme. Sin embargo, no es posible realizar con precisión modificaciones que afecten a fragmentos largos del genoma.
Dos artículos publicados recientemente en Nature ofrecen una nueva solución de edición genómica para fragmentos grandes a través de una nueva técnica basada en una recombinasa programable dirigida por una pequeña molécula de ARN puente. Esta tecnología permite realizar tres tipos fundamentales de reordenamientos del ADN, inserciones, excisiones e inversiones, en posiciones concretas del genoma. Los dos estudios demuestran que este método es efectivo en bacterias y plantean su potencial para otros organismos y células. Si funciona y se optimiza podría derivar en tratamientos para enfermedades causadas por alteraciones cromosómicas.
«El sistema de ARN puente es un mecanismo completamente nuevo para la programación biológica», ha señalado Patrick Hsu, investigador principal del Instituto Arc y la Universidad de California Berkeley y uno de los directores del trabajo. «La recombinación puente puede modificar universalmente el material genético a través de la inserción, escisión, inversión y más, haciendo posible un procesador de textos para el genoma vivo más allá de CRISPR».
Edición de grandes fragmentos de ADN desde la naturaleza al laboratorio
El nuevo sistema para modificar el ADN recurre a una enzima recombinasa. Este tipo de enzimas, presentes en la naturaleza, facilitan el corte y unión de secuencias de ADN en nuevas combinaciones. Por lo tanto son muy atractivas para diseñar sistemas de edición genómica. Especialmente en el caso de recombinasas que reconocen secuencias específicas en los fragmentos de ADN implicados.
En general, estas recombinasas reconocen las secuencias de ADN por un complejo sistema de interacción entre la proteína y el ADN. La novedad y ventaja principal del sistema desarrollado por los investigadores de Instituto Arc y la Universidad de California Berkeley, es que el reconocimiento de las secuencias concretas de ADNs implicadas en el reordenamiento genético está mediado por un pequeño ARN puente, que puede diseñarse según las necesidades de los investigadores.
El ARN puente contiene una región que especifica la secuencia del ADN origen y otra región que especifica el sitio de destino en el ADN. Y lo más importante, ambas regiones pueden reprogramarse. Se pueden crear ARNs específicos que se reconozcan a un amplio rango de secuencias de ADN y permitan diferentes tipos de reorganización de material genético.
Como ocurrió con el conocido sistema de edición del genoma CRISPR, la nueva técnica con recombinasas guiadas por ARN deriva de unos elementos genéticos móviles presentes en genomas procariotas, denominados IS110, que codifican una recombinasa y un ARN puente.
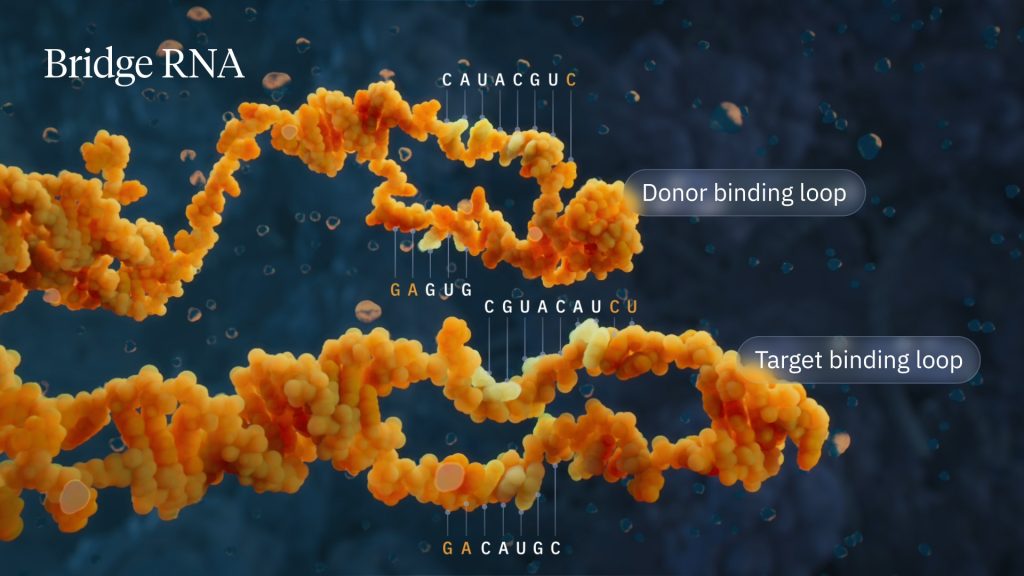
Ventajas de la nueva técnica de edición basada en recombinación con ARN puente
El puente de ARN representa el primer ejemplo de una molécula biespecífica dirigida a un ADN origen (ADN que se integra) y un ADN diana (donde se incorpora el fragmento de ADN). Su combinación con una recombinasa para crear un sistema de edición del genoma ofrece múltiples ventajas.
La primera de ellas es la ya mencionada posibilidad de diseñar el ARN para secuencias de ADN específicas. Además, se trata de un sistema muy compacto, lo que facilita su utilización como herramienta de edición, ya que puede ser empaquetado fácilmente en los vectores que lo transporten al interior de las células. Y, por último, el sistema con el ARN puente promueve la inserción, deleción o inversión de fragmentos de ADN de forma limpiade forma limpia. Los cortes y empalmes entre las cadenas de ADN implicadas son limpios, sin arrastrar parte del ADN colindante. Esto es una mejora significativa respecto a otros métodos de edición de fragmentos largos menos eficaces.
«El mecanismo de recombinación puente resuelve algunos de los retos más fundamentales a los que se enfrentan otros métodos de edición del genoma», ha afirmado Matthew Durrant, investigador en el Instituto Arc de Palo Alto y la Universidad de California Berkeley, así como codirector de la investigación. «La capacidad de reordenar de forma programable dos moléculas cualesquiera de ADN abre la puerta a grandes avances en el diseño de genomas».
Grandes expectativas por cumplir
De momento, el sistema de edición basado en recombinasas con ARN puente ha mostrado eficiencia, aunque variable, en bacterias. También se han observado inserciones en puntos no deseados del genoma. Hará falta, por lo tanto optimizar, su funcionamiento, y, sobre todo, determinar si también funciona en células de mamífero o más específicamente, en humanos.
“Es previsible que lo hagan, aunque deberíamos esperar a ver estos resultados antes de lanzar las campanas al vuelo demasiado pronto, para un sistema de edición mediante recombinación basado en los elementos móviles IS que promete resolver las carencias de los sistemas CRISPR a la hora de reconstruir las graves alteraciones cromosomales que frecuentemente son causa de enfermedades congénitas”, ha destacado Lluís Montoliu, investigador del Centro de Nacional de Biotecnología especializado en edición genómica, quien no ha participado en el estudio, a Science Media Center.
También con el objetivo de desarrollar herramientas para editar el genoma a gran escala, recientemente, investigadores del laboratorio de David Liu en el Broad Institute de MIT, Harvard y HHMI mejoraron la tecnología de edición genética denominada prime editing para insertar o sustituir genes completos en células humanas de manera eficiente. Publicado en Nature Biomedical Engineering, el método eePASSIGE combina la edición prime con enzimas recombinasas de diseño para insertar secuencias de ADN largas en sitios específicos del genoma.
Artículos científicos:
Hiraizumi, M., Perry, N.T., Durrant, M.G. et al. Structural mechanism of bridge RNA-guided recombination. Nature 630, 994–1002. 2024. https://doi.org/10.1038/s41586-024-07570-2
Durrant, M.G., Perry, N.T., Pai, J.J. et al. Bridge RNAs direct programmable recombination of target and donor DNA. Nature 630, 984–993. 2024. https://doi.org/10.1038/s41586-024-07552-4
Pandey, S., Gao, X.D., Krasnow, N.A. et al. Efficient site-specific integration of large genes in mammalian cells via continuously evolved recombinases and prime editing. Nat. Biomed. Eng. 2024. https://doi.org/10.1038/s41551-024-01227-1
Otras fuentes:
Arc Institute Scientists Discover Next-Generation System for Programmable Genome Design. https://arcinstitute.org/news/news/bridgeTou CJ, Kleinstiver BP. Programmable RNA-guided enzymes for next-generation genome editing. Nature. 2024 Jun;630(8018):827-828. doi: http://dx.doi.org/10.1038/d41586-024-01461-2